
方案详情
文
应用一种新的高通量SNP 检测方法-双色荧光杂交芯片技术进行近交系小鼠遗传监测。应用双色荧光杂交芯片技术对4 个品系近交系小鼠的多个基因组DNA 样本进行SNP 分型,整合6 个SNP 位点的芯片杂交信
息,对样本所属品系进行判断。
方案详情

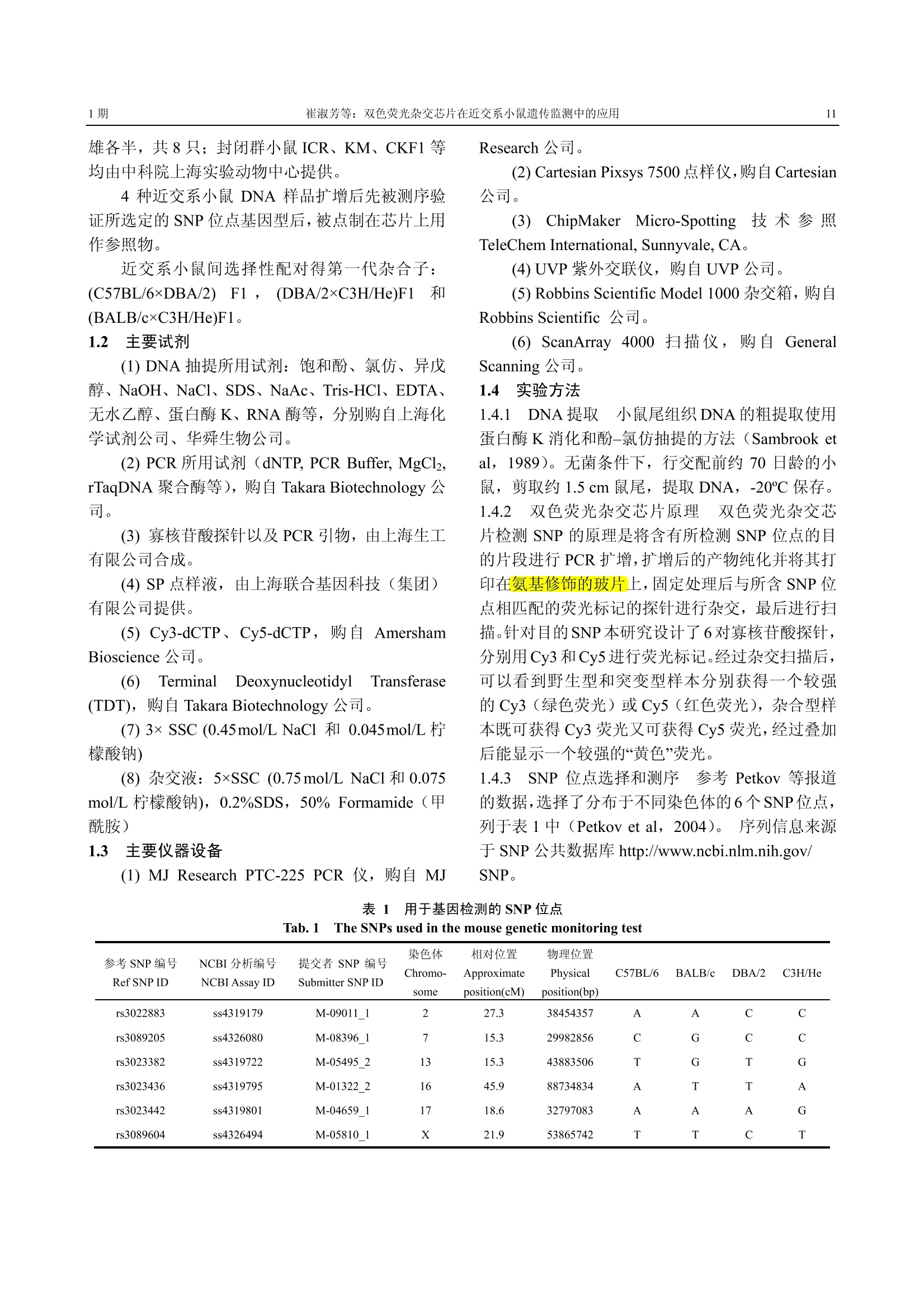
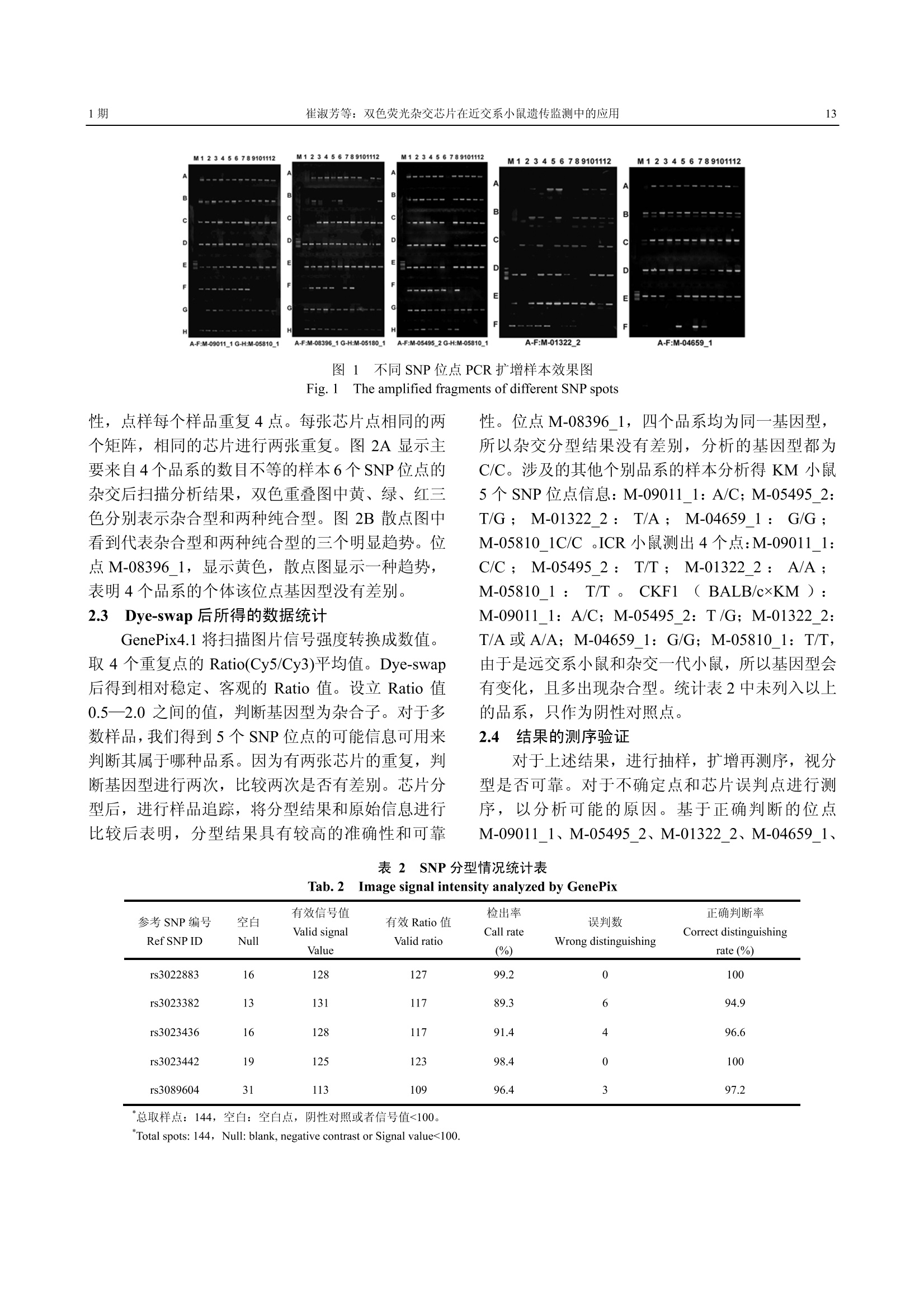
动 物 学 研 究 2008, Feb.29(1):10-16Zoological ResearchCN 53-1040/Q ISSN 0254-5853 崔淑芳等:双色荧光杂交芯片在近交系小鼠遗传监测中的应用1期11 双色荧光杂交芯片在近交系小鼠遗传监测中的应用 崔淑芳1*,瞿秀华1,韩士忠?,余琛琳林,孙 伟1,汤 球 (1.第二军医大学训练部实验动物中心,上海 200433;2.复旦大学生命科学院遗传研究所生物遗传工程重点实验室,上海200433) 摘要:应用一种新的高通量 SNP 检测方法-双色荧光杂交芯片技术进行近交系小鼠遗传监测。应用双色荧光杂交芯片技术对4个品系近交系小鼠的多个基因组 DNA 样本进行 SNP 分型,整合6个 SNP 位点的芯片杂交信息,对样本所属品系进行判断。研究结果表明 SNP 检测方法-双色荧光杂交芯片技术能够对选定的6个 SNP 位点进行高准确率分型;双色荧光杂交芯片技术是一种高通量 SNP检测的良好工具,适合于对少量近交系品系来源的大样本量小鼠进行遗传污染监测和品系鉴定,并具有扩大应用的潜力。 关键词:SNP;双色荧光杂交;小鼠 中图分类号:Q341;Q524 文献标识码:A 文章编号:0254-5853-(2008)01-0010-07 Application of Dual-color Fluorescence HybridizationChip Technique in the Monitoring of the GeneticQuality of Inbred Mice Strains CUI Shu-fang*, QU Xiu-hua, HAN Shi-zhong , YU Chen-lin, SUN Wei ,, TANG Qiu (1. Experiment Animal Center; Training Department, Second Military Medical University, Shanghai 200433, China;2. State Key Lab of Genetic Engineering, Institute ofGenetics, School ofLife Science, Fudan University, Shanghai200433,China) Abstract: This study aimed to use a microarray-based method for scoring a number of genomic DNA in parallel foione SNP marker on a glass slide for the genetic monitoring of inbred mice. We applied the method to identify inbredlaboratory mice strains to validate the reliability and stability of this genotyping panel. The amplified PCR products fromfour inbred strains were spotted and immobilized onto amino-modified glass slides to generate a microarray, which wasthen interrogated by hybridization with dual-color probes to determine the SNP genotype of each sample. The resultsindicated that this microarray-based method could effectively determine the genotype of the six selected SNPs with ahigh degree of accuracy. This SNP genotyping method is feasible for monitoring genetic contamination in colonies oflimited genetic diversity. Moreover, an enlarged version of the panel could be used to analyse a large number of strains. Key words: SNP; Dual-color fluorescence hybridization; Mouse 随着生命科学的不断发展,对实验动物遗传品质的要求也越来越高,对动物群体遗传的均一性和个体的差异性更为关注。如何对越来越多的近交系动物遗传质量进行监测,是目前急需解决的问题。只有研制出更方便,更可靠的监测方法,才能保证我国实验动物遗传质量的提高。基因芯片技术是90年代初随着人类基因组计划而兴起的新技术,具有高通量、大规模、平行性等特点,本研究首次将此方法运用于近交系动物的遗传监测,探讨双色荧光 ( 收稿日期:20 0 7-10-26;接受日期 : 2007-12-10 ) ( 基金项目:上 海 市科技发展基金(044909004) ) ( 通 讯 作者(Corre s p o nding author), E-mail: sfcui@smmu.edu.cn ) 杂交芯片技术在近交系实验动物遗传监测的可行性。 材料与方法 1.1 实验材料 (1)低密度氨基修饰的基片,由上海联合基因科技(集团)有限公司提供。 (2) 实验动物:4种不同品系2月龄近交系小鼠C57BL/6N, BALB/cAnN, C3H/HeJ 和 DBA/2J, 雌 雄各半,共8只;封闭群小鼠 ICR、KM、CKF1等均由中科院上海实验动物中心提供。 4 种近交系小鼠 DNA 样品扩增后先被测序验证所选定的 SNP 位点基因型后,被点制在芯片上用作参照物。 近交系小鼠间选择性配对得第一代杂合子:(C57BL/6xDBA/2) F1,,((DBA/2xC3H/He)F1 和(BALB/cxC3H/He)F1。 1.2 主要试剂 (1) DNA 抽提所用试剂:饱和酚、氯仿、异戊醇、NaOH、NaCl、SDS、NaAc、Tris-HC1、EDTA、无水乙醇、蛋白酶K、RNA酶等,分别购自上海化学试剂公司、华舜生物公司。 (2) PCR 所用试剂 (dNTP, PCR Buffer, MgCl2,rTaqDNA 聚合酶等),购自 Takara Biotechnology 公司。 (3)寡核苷酸探针以及 PCR 引物,由上海生工有限公司合成。 (4) SP 点样液,由上海联合基因科技(集团)有限公司提供。 (5) Cy3-dCTP、Cy5-dCTP, 购自 AmershamBioscience 公司。 (6) Terminal Deoxynucleotidyl Transferase(TDT), 购自 Takara Biotechnology 公司。 (7) 3× SSC (0.45mol/L NaCl 和 0.045mol/L 柠檬酸钠) (8) 杂交液:5×SSC (0.75 mol/L NaCl 和 0.075mol/L柠檬酸钠), 0.2%SDS, 50% Formamide (甲酰胺) 1.3 主要仪器设备 (1) MJ Research PTC-225 PCR 仪, 购自 MJ Research公司。 (2) Cartesian Pixsys 7500 点样仪,购自 Cartesian公司。 (3) ChipMaker Micro-Spotting技术参照TeleChem International, Sunnyvale, CA。 (4) UVP紫外交联仪,购自UVP公司。 (5)Robbins Scientific Model 1000 杂交箱,购自Robbins Scientific 公司。 (6) ScanArray 4000 扫描仪,购自 GeneralScanning公司。 1.4 实验方法 1.4.1 DNA提取 小鼠尾组织DNA 的粗提取使用蛋白酶K消化和酚-氯仿抽提的方法(Sambrook etal, 1989)。无菌条件下,行交配前约70日龄的小鼠,剪取约 1.5 cm鼠尾,提取 DNA, -20℃保存。 1.4.2 双色荧光杂交芯片原理 双色荧光杂交芯片检测 SNP 的原理是将含有所检测 SNP 位点的目的片段进行 PCR 扩增,扩增后的产物纯化并将其打印在氨基修饰的玻片上,固定处理后与所含 SNP位点相匹配的荧光标记的探针进行杂交,最后进行扫描。针对目的 SNP 本研究设计了6对寡核苷酸探针,分别用 Cy3 和Cy5进行荧光标记。经过杂交扫描后,可以看到野生型和突变型样本分别获得一个较强的Cy3(绿色荧光)或 Cy5(红色荧光),杂合型样本既可获得 Cy3 荧光又可获得 Cy5 荧光,经过叠加后能显示一个较强的“黄色”荧光。 1.4.3 SNP 位点选择和测序 参考 Petkov 等报道的数据,选择了分布于不同染色体的6个SNP位点,列于表1中 (Petkov et al, 2004)。 序列信息来源于 SNP 公共数据库 http://www.ncbi.nlm.nih.gov/ SNP。 表1 用于基因检测的 SNP 位点 Tab.1 The SNPs used in the mouse genetic monitoring test 参考 SNP 编号 Ref SNP ID NCBI分析扁号 提交者 SNP 编号 染色体 相对位置 物理位置 Chromo- Approximate Physical C57BL/6 BALB/c DBA/2 C3H/He NCBI Assay ID Submitter SNP ID some position(cM) position(bp) rs3022883 ss4319179 M-09011_1 2 27.3 38454357 A A C C rs3089205 ss4326080 M-08396_1 15.3 29982856 C G C C rs3023382 ss4319722 M-05495_2 13 15.3 43883506 T G T G rs3023436 ss4319795 M-01322_2 16 45.9 88734834 A T T A rs3023442 ss4319801 M-04659_1 17 18.6 32797083 A A A G rs3089604 ss4326494 M-05810_1 X 21.9 53865742 T T C T 近交系共8只小鼠的 DNA 样本经过 PCR扩增后,进行测序,确认基因组序列的 SNP 位点信息是否与表1列出的相同。 1.4.4 PCR 扩增 四个品系的标准品经过测序确认各 SNP 位点的基因型后,确认引物: M-09011_1f, 5 '-cctcaaaggaagacaggtccaa-3;M-09011_1r,5'-gccgctcacaactgaatagaaat-3';M-05495_2f,5'-accaaggatgtagagggtgaa-3';M-05495_2r, 5'-ggatgggtatgtaagtatgtgc-3';M-01322_2f, 5'-ggcatcatctattgggttgttaa-3';M-01322_2r, 5'-ctgcttccctgtgaatccgtgag-3';M-04659_1f,5'-gcttctcggacggttcttcttat-3';M-04659_1r, 5'-ctttgggcgtttcttctcattct-3' 80uL PCR 反应体系包括 0.2mmoL dNTP, 1.5mmoL MgCl2, 0.1一1ng模板, 0.2umoL 正向引物,0.2umoL 反向引物, 5U rTaq DNA 聚合酶(Takara).MJ research PTC-225 PCR 仪的反应条件包括:95℃5 min 预变性; 94℃ 30s, 54C 30s,72℃ 30s,335个循环;72℃ 10min 延伸。 PCR产物4℃冰箱保存,用于点样。 1.4.5 探针荧光标记寡核苷酸探针通过 TDT 酶(terminal deoxynucleotidyl transferase)标记 Cy3 或Cy5 荧光染料。标记体系包括 0.16umoL 寡核苷酸探针, 0.16mmoL Cy5(Cy3), 3.5U TDT。在 MJresearch PTC-225 PCR 仪内, 37℃ 2.5h, 95℃ 1min。寡核苷酸探针序列依次为: ( M-09011_ 1 t 5'ctggaaaattctgcaga3', M -09011_ 1 g 5'ctggaaaagtetgcaga3; ) ( M-08396_1c 5'ttctgctgcatcatggc3', M -08396_1 g 5"ttctgctggatcatggc3; ) M-05495_2t 5'caggcagatctgctgga3, M-054952g 5'caggcagagctgctgga3'; ( M-01322_2a 5'acatcaggaattcagaag3' , M-01322_ 2t 5'-acatcaggtattcagaag-3'; ) M-04659_1g 5'tacctcacgccaacact3', M-046591a 5'tacctcacaccaacact3': M-05810_lt 5'tcactgttcttgcctctc3', M-05810_1c5'tcactgttcctgcctctc3; 1.4.6 点样制片Cartesian Pixsys 7500 点样仪将溶解于点样缓冲液和50% DMSO 的 PCR产物点制到氨基修饰的基片,矩阵24×24,每个样品重复 4次,每张芯片上下两个矩阵。然后将芯片置于相对湿度70%环环水合2h, 室温干燥 0.5h, UV 强度为 65 MJ/cm 的条件下进行紫外交联。最后芯片用0.2%SDS 洗涤10min, 蒸馏水洗涤 10min, 0.2%琥珀酸酐洗涤 10min,干燥后密封盒内4℃保存。 1.4.7 杂交与洗涤 杂交:将已点样的芯片95℃变性2min, 然后置于预冷的无水乙醇中冷却 1 min,室温晾干。等量混合已标记的探针(6uL),加入等体积的杂交液(12pL),再加阳性对照 CCOB 2pL,将混合液加到玻片的点样区域内,盖上盖玻片,放入杂交箱内42C 杂交 1h。 洗片:将分别装有溶装I(1xSSC+0.2% SDS),溶液Ⅱ (1xSSC+0.2%T ween)的两个染色缸,放在25℃水浴锅中。杂交后的芯片依次浸入以上两个染色缸中洗涤6min, 用0.1xSSC冲洗玻片,室温下晾干。 1.4.8 图像扫描与数据处理 杂交后的芯片通过Packard ScanArray 4000 微阵列分析系统在相应波长扫描。对每一种染料扫描后得到图像,并合得到每个点的重叠图。通过 Axon 公司的 Gene Pix Pro4.1软件处理分析荧光信号强度。每个点的信号用控制好的强度进行扫描测量。净信号值由每个点的平均强度减去背景强度。利用杂合子的信号值对芯片数据中系统误差进行校正。求取重复点的 Ratio 中位值或平均值。 1.4.9 测序验证 每张芯片进行抽样, PCR 扩增再测序,来判断芯片的准确性。对于不确定点和芯片误判点进行测序,分析可能的原因。 2 结果 2.1 PCR 扩增样本效果图 小鼠样品在96孔板随机排列,记录各孔位所对应的品系个体。分两批样品进行扩增,对于一次扩增效果较差者,进行重新扩增,第二次未能扩出的,则在芯片点样时,将其作为阴性对照(见图1—5)。 2.2 SNP 位点杂交结果 理论上,4种近交系品系至少能够通过3个标记相互区分开。交叉配对经过测序的小鼠个体得F代,得到的杂合子覆盖这6个位点。设计17十18 mer寡核苷酸和500 bp 长度的 PCR 产物。384孔板中随机排列包括4个品系的及其他品系来源的少数样本,加上原始的8个体和其杂合子作为参照。设立空白对照、阴性对照。加入 DMSO 有利于减少 DNA一级结构, 使(G+C)%含量高的模板易于完全变 图1 不同 SNP 位点 PCR 扩增样本效果图 Fig.1The amplified fragments of different SNP spots 性,点样每个样品重复4点。每张芯片点相同的两个矩阵,相同的芯片进行两张重复。图2A 显示主要来自4个品系的数目不等的样本6个 SNP 位点的杂交后扫描分析结果,双色重叠图中黄、绿、红三色分别表示杂合型和两种纯合型。图2B 散点图中看到代表杂合型和两种纯合型的三个明显趋势。位点M-08396_1,显示黄色,散点图显示一种趋势,表明4个品系的个体该位点基因型没有差别。 2.3 Dye-swap后所得的数据统计 GenePix4.1将扫描图片信号强度转换成数值。 取4个重复点的 Ratio(Cy5/Cy3)平均值。Dye-swap后得到相对稳定、客观的 Ratio 值。设立 Ratio 值0.5一2.0之间的值,判断基因型为杂合子。对于多数样品,我们得到5个 SNP 位点的可能信息可用来判断其属于哪种品系。因为有两张芯片的重复,判断基因型进行两次,比较两次是否有差别。芯片分型后,进行样品追踪,将分型结果和原始信息进行比较后表明,分型结果具有较高的准确性和可靠 性。位点M-08396 1,四个品系均为同一基因型,所以杂交分型结果没有差别,分析的基因型都为C/C。涉及的其他个别品系的样本分析得 KM 小鼠5个 SNP 位点信息: M-09011 1: A/C; M-05495 2:T/G;M-01322 2::T/A; M-046591:G/G;M-05810_1C/C .ICR小鼠测出4个点:M-09011_1:C/C:;M-05495 2::T/T: M-013222:: A/A:M-05810 1::T/T。CKF1 ( BALB/cxKM):M-09011 1:A/C; M-05495 2:T/G; M-01322 2:T/A或A/A; M-04659_1: G/G; M-05810_1: T/T,由于是远交系小鼠和杂交一代小鼠,所以基因型会有变化,且多出现杂合型。统计表2中未列入以上的品系,只作为阴性对照点。 2.4 结果的测序验证 对于上述结果,进行抽样,扩增再测序,视分型是否可靠。对于不确定点和芯片误判点进行测序,以分析可能的原因。基于正确判断的位点M-09011_1、M-05495 2、M-01322 2、M-04659_1、 Tab.2 Image signal intensity analyzed by GenePix 参考SNP编号 空白 有效信号值 有效 Ratio 值 检出率 误判数 正确判断率 Valid signal Call rate Correct distinguishing Ref SNP ID Null Valid ratio Wrong distinguishing Value (%) rate(%) rs3022883 16 128 127 99.2 0 100 rs3023382 13 131 117 89.3 6 94.9 rs3023436 16 128 117 91.4 4 96.6 rs3023442 19 125 123 98.4 0 100 rs3089604 31 113 109 96.4 3 97.2 总取样点:144,空白:空白点,阴性对照或者信号值<100。 图2 不同品系小鼠样本6个 SNP 位点的杂交后扫描分析结果(每个样品重复4点) Fig.2Six SNP microarray image from DNA samples of the different inbred mice and their hybrids(quadruplicate for each spot) A:双色重叠图,黄、绿、红三色分别表示杂合型和两种纯合型;B:散点图。 A: Dual-color fluorescence hybridization image. In these dual-color results the three colors yellow, green, and red represent the three genotypesof two homozygotes and a heterozygote,respectively; B: 3-D scatter plot. M-058101的芯片结果,测序结果与芯片一致。 扫描图像显示全为黄色的位点 M-08396 1,样品追踪选择2个芯片上有信号的,一个没有信号的样品,扩增后测序结果都是 C/C, 由于参考 BALB/cJ的 SNP 位点信息 G/G, 并且撇开实验开始不完整的测序信息(初步显示基因型是C/C), 我们假设存在杂合子、两种纯合子,重新取样,测序结果都是 C/C。经分析,Cy5 和 Cy3 在扫描过程中出现了相同的信号图像,双色重叠呈黄色,可能是因为芯片的扫描强度是人为调整的,如果只出来C的信号,将另——个荧光信号通道的强度人为调整到非常高的情况下,也会出现跟C相对应的信号。如此,测序和芯片的结果吻和,可说明这些样本都是 C/C 基因型,证明选择的4个品系的样本该 SNP 位点基因型相同, BALB/cAnN 该 SNP位点基因型是 C/C,而非BALB/cJ的 G/G。 M-01322 2错判点,由T/A 错判成 A/A。测序后峰图上A 的峰高大于T两倍以上,而芯片是以2倍以上来判断纯合与杂合的,这可能就是芯片判为A的原因。 3 讨 论 3.1 基因分型的准确性、重复性 基因分型的准确性:通过测序验证所得到的结果的相近程度来判断。为了确认基因分型的准确性,本研究抽样选择了每张芯片上的数个有结果的样本, PCR 扩增后测序,芯片结果同测序结果一致。如果有成功的 PCR 点样,该系统能够高效地区分品 系,准确判断率达95%以上。不可控因素或者说操作上的因素导致了一些样品低效率的 PCR扩增,这样也出现了一些低信号值和模糊Ratio 值影响结果,这样的点忽略不不。对于3个 SNP位点,出现了错误的判断,比如说杂合子的被判成纯合子,或者纯合子的被判成杂合子。上述 M-013222位点,样本经过测序确定由 T/A 错判成A/A,其原因可能一是样品细胞为嵌合体,纯合与杂合都有,从绝对数量上某-一个型的占主要。二是引物设计时可能与A的有连锁,因而A的产量高。三是可能发生突变了。其他的原因如有缺点的统计方法导致了得到的信息有偏颇。对于这样的样本,需使用测序或其他方法做进一步研究(Hao et al,2004; Kang et al, 2004;Tintle et al, 2005)。 重复性:本研究统计每个样本重复点样4次,每次同时用2张芯片,共计8次重复的情况下,8次重复性的基因分型的一致性。位点M-09011 1共8次重复检测的基因型结果中,1020个基因型结果中,3个不一致,重复性为 99.7%。说明此芯片技术具有较好的重复性。 检出率:检出率是指每张芯片上所检测到的基因型除以每张芯片上总的 SNPs。如果不考虑用来作为对照的未见杂交信号的空白对照和阴性对照,和具有很强信号的内对照 CCOB, 那么检出率如表3中 Call Rate 所示,总体上具有较高的检出率。 实验结果表明双色荧光杂交芯片技术是一个高通量 SNP 检测的良好工具,适合于对少量近交系品系来源的大样本量小鼠进行遗传污染监测和品 系鉴定,并具有扩大应用的潜力。 3.2 芯片误差来源分析 芯片实验的许多可变因素可归结于两个方面:生物学变异和实验系统误差,生物学上的差异是内在的,受到遗传和环境因素的影响。实验系统误差可包括芯片制作带来的误差和样本检测过程中的误差。涉及我们实验的主要是实验系统误差,首先是基因芯片制备过程中:PCR扩增及纯化过程,由于是提取小鼠尾尾织 DNA, 量较难掌握、有伴随污染的可能,导致有些样本没有能够扩增出产物。点样及点样后处理,氨基修饰基片带有正电荷,可以与带负电荷的 DNA 形成离子键,从而固定 DNA,DNA 长度越长,固定效率越高,因此它可以直接用于固定 DNA。在点样的过程中可能由于点样针的堵塞,造成4个重复点中某个点的样品缺失,以致杂交没有信号,点样后若未能洗脱没有固定的 DNA带来高的背景,洗脱没有固定好的 DNA 则信号缺失 (Potter et al, 1981)。 其次还在于样本的检测过程中:标记过程中荧光染料的问题,荧光的量决定了每个 DNA 分子有多少荧光素分子被掺入,染料之间存在着差异。杂交过程中,杂交的时间、温度、杂交液的配方等的不同也会带来误差,本实验参照实验第一部分的杂交动力学的实验结果,采用相对稳定的杂交条件,尽量避免偏差。图像扫描和处理过程中扫描仪和软件的差异也可能导致误差,我们在实验室内部两台ScanArray 扫描所得数据有较明显差别,并都存在Cy3 扫描信号偏低的现象。我们选择了一台相对偏差较小的扫描仪,分多个激光强度和 PMT 值进行扫描,以获得最佳的效果。分析软件如我们使用的Axon 的 Genepix4.1软件,取点和背景的原理有好几种,不能100%反映原始信息。由于各软件核心算法不同,同样的原始图片,得到的数据也会有不同。统计软件的误差很可能导致一些重要信息的偏差或丢失 (Potter et al,1981; Brazma et al, 2001)。 减少误差的最好方法是进行重复,我们在实验中进行了点样点的重复,每个样本重复4次,每张芯片点两个矩阵,相同的荧光标记重复2张芯片。取同一张芯片相对信号较强的矩阵进行分析,获取信号值计算 Ratio 值,取4个点重复的平均 Ratio值。大多数的芯片杂交过程中会出现明显的染料偏差。芯片使用的标记染料为 Cy5和 Cy3, 虽然这两 种染料的性质非常相近,从它们的化学结构看, Cy5只比 Cy3 多了一个碳原子,但是它们的标记效率还是存在着差别。为了得到更客观的数据,我们试图除去类似误差。正反标记荧光标记实验(Dye-swap)能很好地平衡数据减少误差的产生 (Rosenzweig etal, 2004; Yang et al, 2002)。 Dye-swap 使用两张芯片来引较两种基因型 A/A 和 C/C, 例如实验中 SNP位点 M-09011 1二等位基因 A/C, 第一张芯片上,带有A的寡核苷酸序列标记 Cy5,带有C的寡核苷酸序列标记 Cy3;第二张芯片则相反。各自得到的平均 Ratio 值,经过Ln (Ratio)取平均之后,算得最终 Ratio 值,进行品系判定。通过两次的正反标记实验得出的基因型判断,我们能较为客观地判定样本的 SNP 基因型。 3.3 扫描图像质量分析 完美的阵列图像很少见,许多问题很难追踪原因,而且在阵列扫描之前无法用可检测手段证明这些原因。我们扫描所得的双色重叠图暴露了以下的几点问题,样点的形状问题:样点的大小不一致,但实验结果表明如能保证点够大而独立,点的大小、形状差异并不是一个严重问题。高背景问题:由于异常的高荧光背景而导致芯片模糊。高背景出现在整个芯片的杂交表面,也有出现在一个局部区域。高背景局限于杂交区,说明标记的探针粘着于玻片表面,在洗片步骤时没有被冲洗掉,导致这一结果有两种可能性, 是氨基基团封闭不足,或是在杂交中标记的探针非特异吸附。局部信号微弱问题:特异性信号减弱,可能是探针的信号被芯片表面的高背景所掩盖。也有可能是因为这-一区域探针层太薄而引起的。芯片和盖玻片表面的气泡和不匀质也可能引起这种结果。 经过对照,认为在杂交前预先系统地扫描芯片,可以避免覆盖整个芯片的明显高背景。仔细选择和放置玻片与盖玻片可减少局部信号微弱这个问题的出现。 建立多级质量控制有利于提高图像品质。氨基修饰玻片应质检,小量修饰好的玻片进行点样并测试整个后处理和杂交过程。玻片点样后,对小量点样玻片进行后处理,后处理是否成功可用靶标固定率检测方法来判定。所有阵列在杂交前扫描一次,弃去那些有明显污点的玻片,并弃去那些杂交前背景就高的阵列。 ( Brazma A, Hingamp P, Quackenbush J, Sherlock G, Spellman P, Stoeckert C,Aach J , Ansorge W , Ball C A , Ca u ston HC, Gaasterland T, G l enisson P,Holstege F C, K im IF, M arkowitz V, Matese JC, Parkinson H, Robinson A, S arkans U, Schulze-Kremer S, St e wart J, Ta y lor R, Vi l o J, V in gron M. 2001. M i nimum in f ormation ab o ut a mi c roarray experiment (MIAME)-toward s t andards for m i croarray data[J]. N a t G e net29(4):365-367. ) ( Hao, K, W a ng X. 200 4 . Incorporating individual error rate into associationtest of u nmatched case-control design[J]. Hum H e red, 58( 3 -4):1 5 4-163. ) ( Kang, SJ, G ordon D , B r own AM , Ott J, F inch SJ, Ka n g SJ. 2004. Pacific symposium on biocomputing. Tradeoff between no-call r e duction in g enotyping e r ror r a te and l o ss of sample si z e for genetic case/controlassociation studies[J]. Pac Symp Biocomput, 1 1 6-127. ) ( P etkov, P M , Ca s sell M A, S a rgent E E , Donnelly CJ, R o b inson P, Cr e w V,Asquith S, Haar RV , W iles M V. 2004. D evelopment o f a S NPgenotyping panel f o r g e netic m o nitoring of th e la b oratory mo u se[J]. ) ( Genomics, 83(5):902-911. ) ( Potter M, Wa x JS . 19 8 1. Ge n etics of sus c eptibility to p r istane-inducedplasmacytomas i n B ALB/cAnN: red u ced sus c eptibility in B A LB/cJwith a brief description o f pristane-induced arthritis[J]. J Immunol, 127(4): 1 591-1595. ) ( Rosenzweig BA, Pine P S, D omon O E , Morris SM, Chen JJ , Sistare F D . 2004. D ye-bias c orrection i n d ual-labeled cDNA m i croarray gene expression measurements: Genomics and risk assessment: mini-monog- r aph[J]. Environ Health Perspect,112(4):480-487. ) ( Sambrook, E F Fritsch J, M aniatis T. 1989. Molecular Cloning: a Laboratory M anual[M]. 2nd Ed. New York: Cold Spring Harbor Press. ) ( Tintle NL, A hn K,Mendell NR, Gordon D, Fin c h SJ. 2005. Ch a racteristicsofreplicated single-nucleotide polymorphism genotypes from C OGA: A ffymetrix and center for inherited disease research[J]. BMC Genet, 6 (Suppl):15. ) ( Yangg YH, Speeedd T. 2002. Design i ssues for cDNA m i croarrayexperiments[J]. Nat Rev Genet, 3(8):579-588. ) Information of Dr. ZHANG Zhi-qiang, a member in the 7 Editorial Board of ZR ZHANG Zhi-qiang was born Shanghai in 1963. Zhi-qiang waseducated at Fudan University in China (BSc in zoology in1985). Hecompleted his PhD in entomology at Cornell University (Ithaca, NewYork, USA) in 1993 and worked as an insect ecologist at Oregon StateUniversity, Corvallis, Oregon, USA from1992 to 1994. During1994-1999, he was an acarologist for the CABI International Institute ofEntomology based in the Department of Entomology, The NaturalHistory Museum, London, UK. He also served as a Technical Officer forthe Secretariat of the BioNET-International (The Global Network for Taxonomy) during 1998一1999. Since 1999, he 张智强博士 has been a researcher and curator (Acari) at the New Zealand Arthropod Collection, Landcare Research, Auckland, New Zealand. Zhi-qiang is an honorary research fellow at the Department of Entomology, The Natural History Museum (London,UK), an adjunct professor at Fudan University (Shanghai, China) and Hebei Normal University (Shijiazhuang, China),an honorary professor at Fujian Academy of Agricultural Sciences (Fuzhou, China), and a guest professor at FujianAgricultural and Forestry University (Fuzhou, China). Zhi-qiang is the president of the Systematic & AppliedAcarology Society and a member of the International Executive Committee of the International Congress of Acarology.He is the editor/founder of several international journals of zoology, including Zootaxa. Since 2006, he has been aCommissioner of the International Commission on Zoological Nomenclature. Zhi-qiang Zhang's research is centred around the systematics, diversity and ecology of the Acari. He hascollaborated with colleagues in China on several biocontrol projects. Zhi-qiang has published over 200 papers, 102 ofthese were cited for a total of 482 times in SCIE (as of 24 Apr 2007).
确定





还剩5页未读,是否继续阅读?
海德创业(北京)生物科技有限公司为您提供《双色荧光杂交芯片在近交系小鼠遗传监测中的应用》,该方案主要用于其他中--检测,参考标准--,《双色荧光杂交芯片在近交系小鼠遗传监测中的应用》用到的仪器有
相关方案
更多
该厂商其他方案
更多








