方案详情
文
Agilent 2200 TapeStation 系统可对新一代测序 (NGS) 工作流程中的样品进行全程分析,从最初的起始材料一直到测序前的最终 DNA 文库质量控制 (QC)。本应用简报遵循 SureSelect 文库制备实验方案,展示了基因组 DNA ScreenTape、D1000 ScreenTape 及高灵敏度 D1000 ScreenTape 分析方法对该工作流程中样品的分析性能及适用性。数据证明了 2200 TapeStation 系统是可靠的文库分子量测定及定量分析 QC 平台。数据还证明了这些 DNA ScreenTape 分析方法与 Agilent 2100 生物分析仪的相应分析方法具有可比性,并在其性能指标内。
方案详情

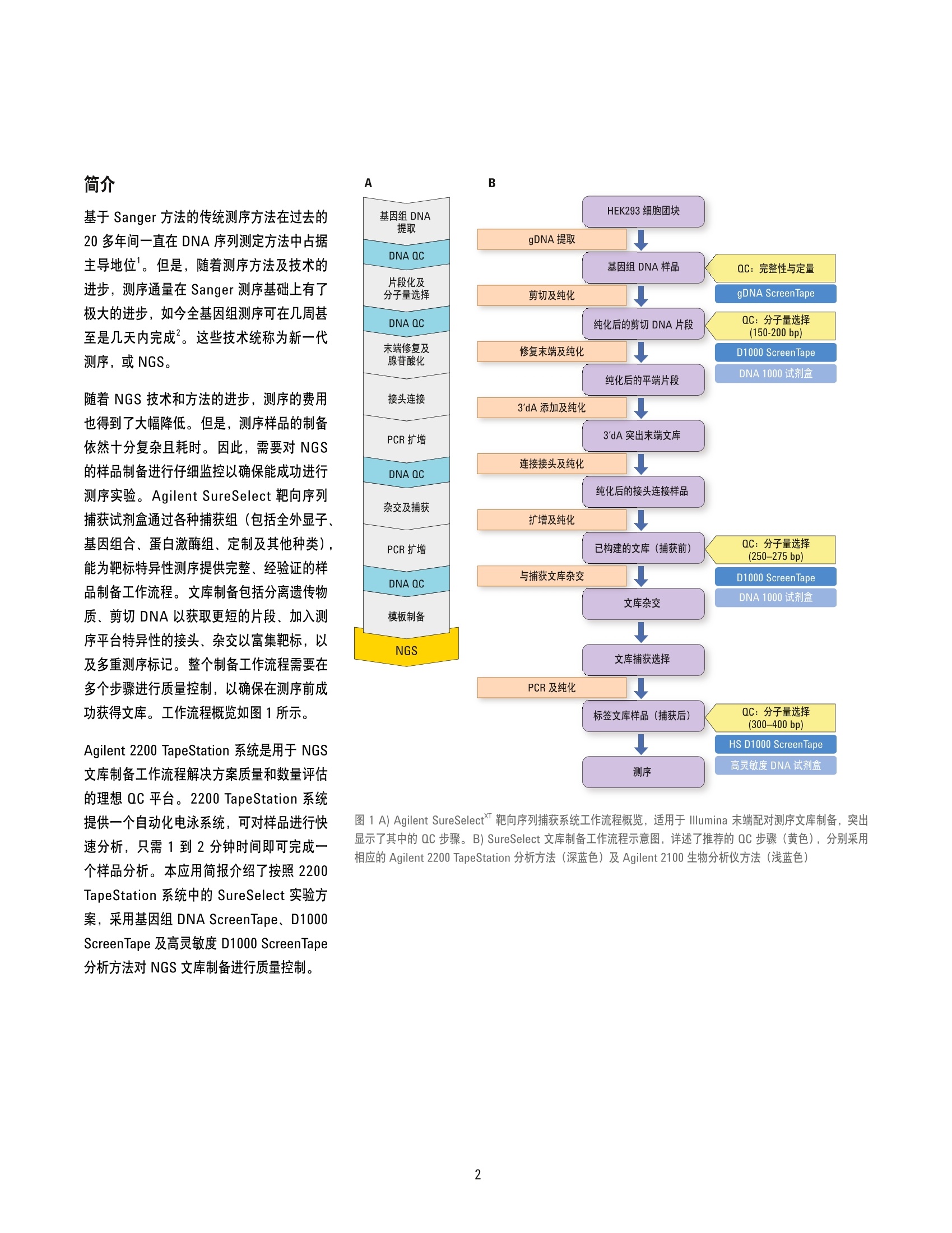
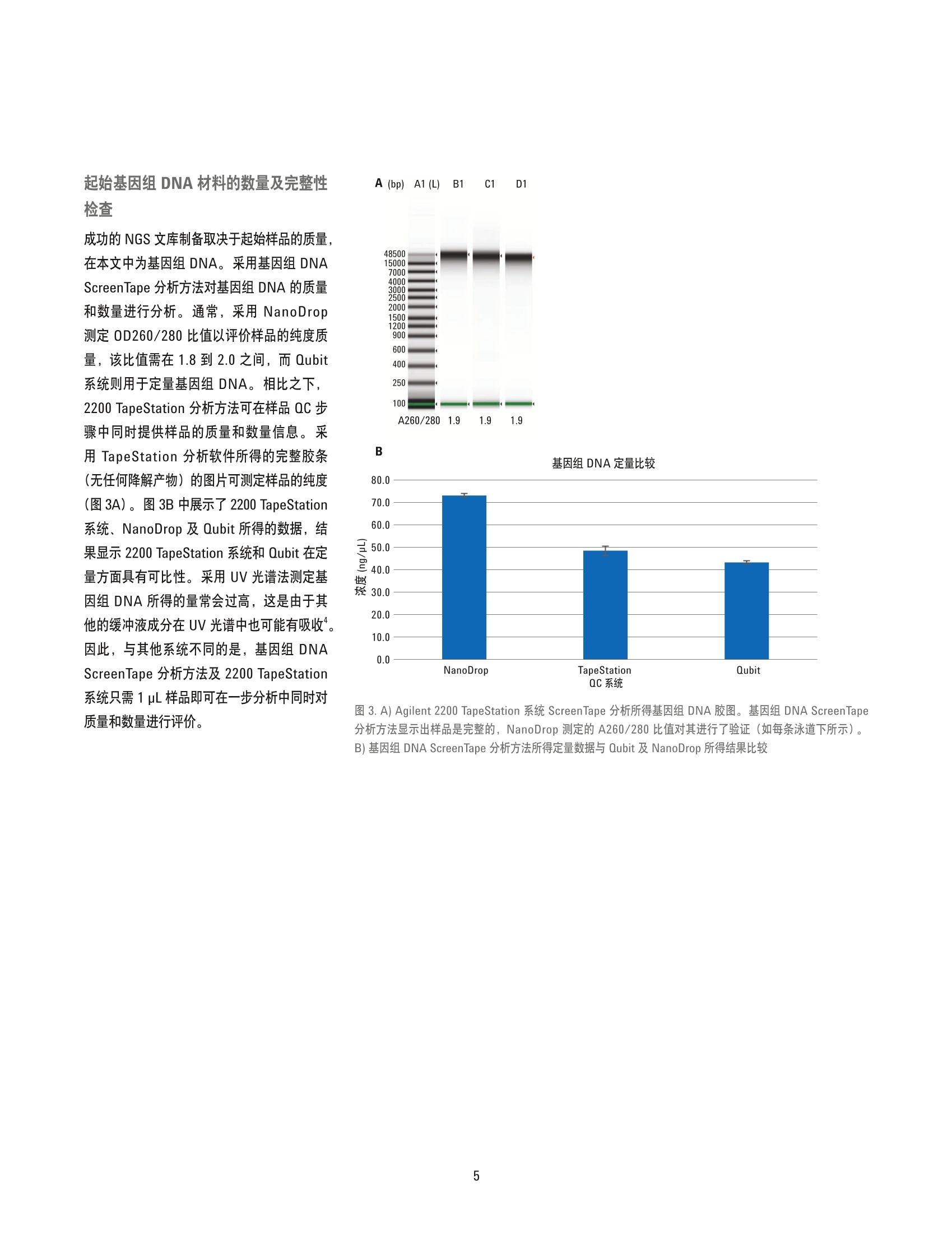
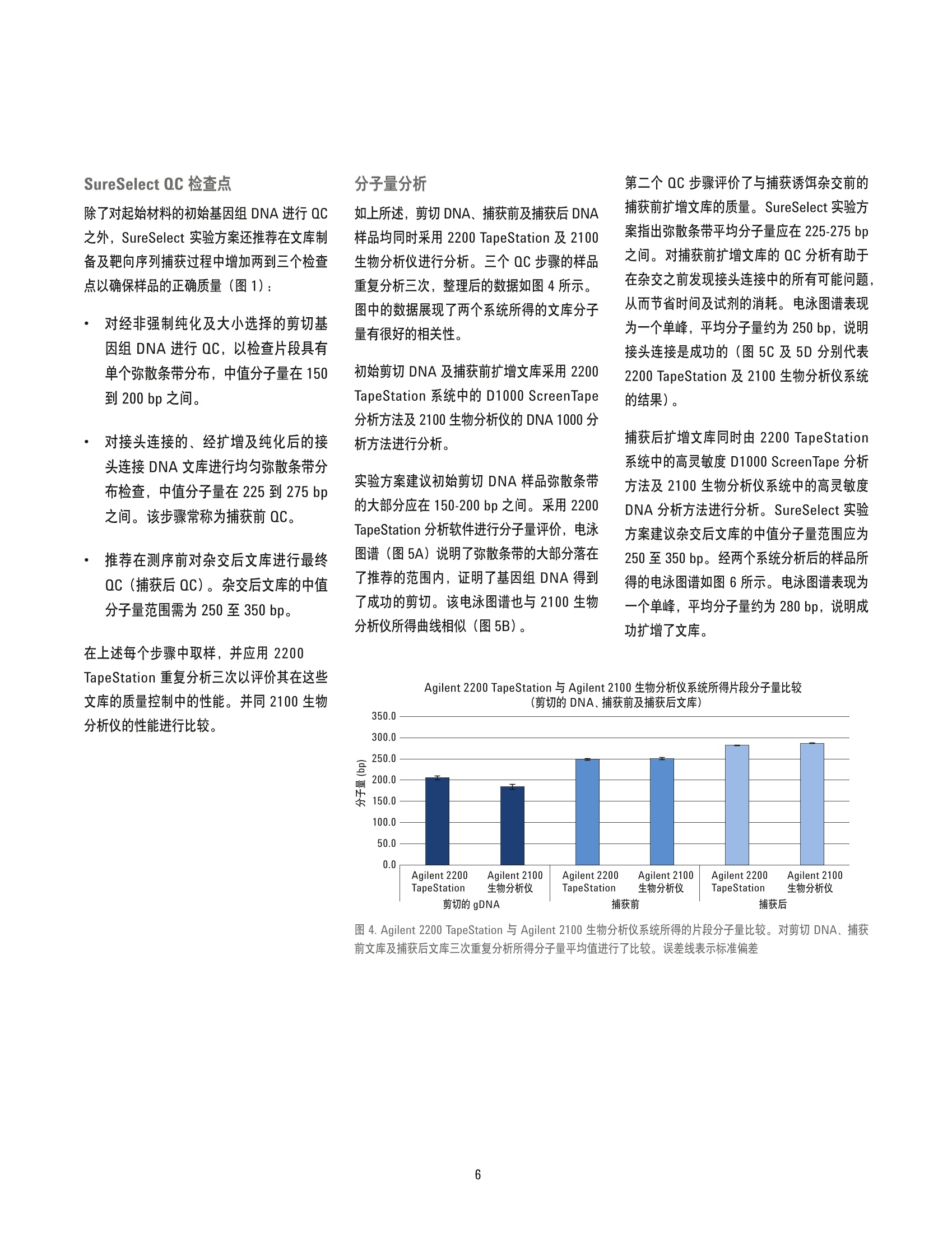
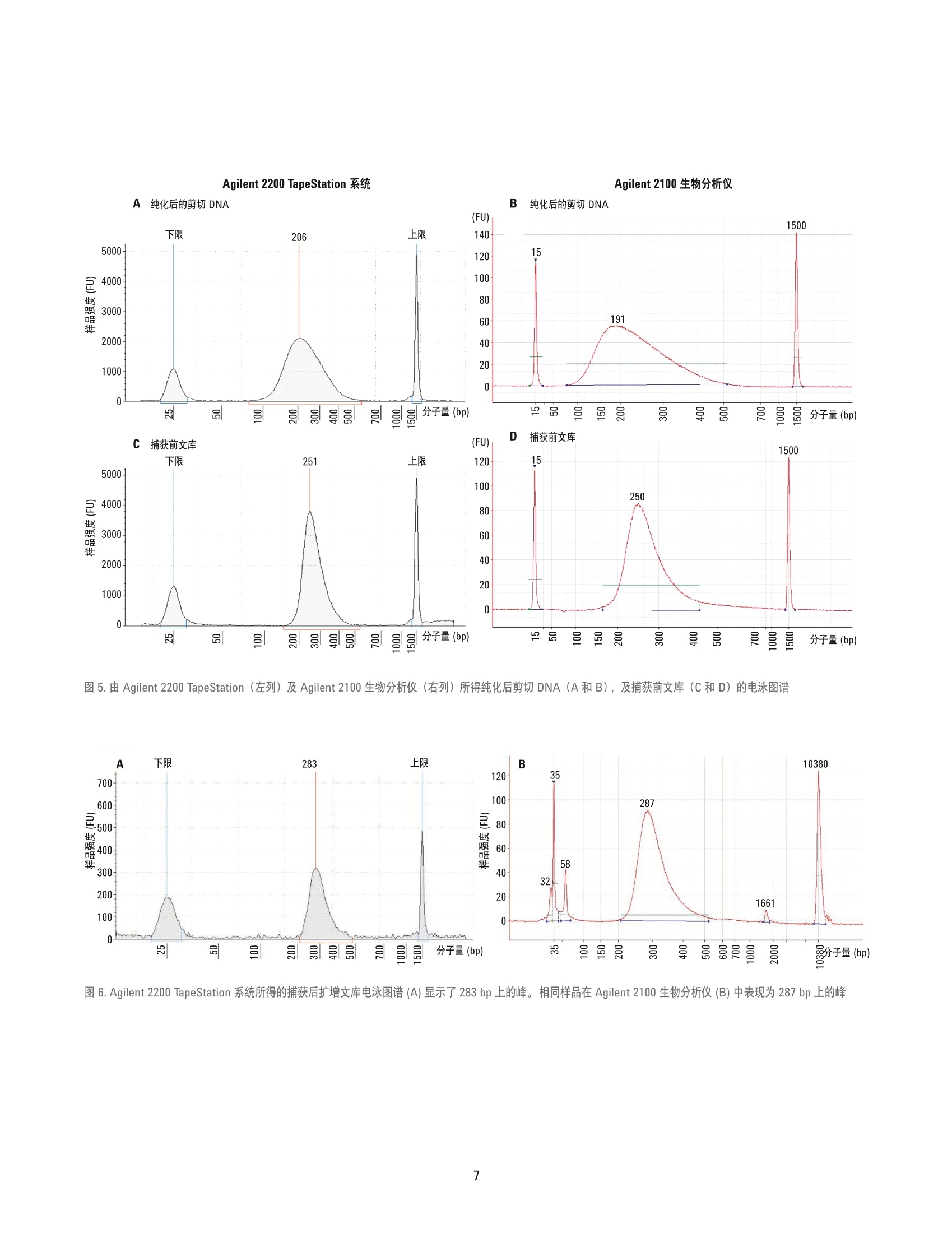
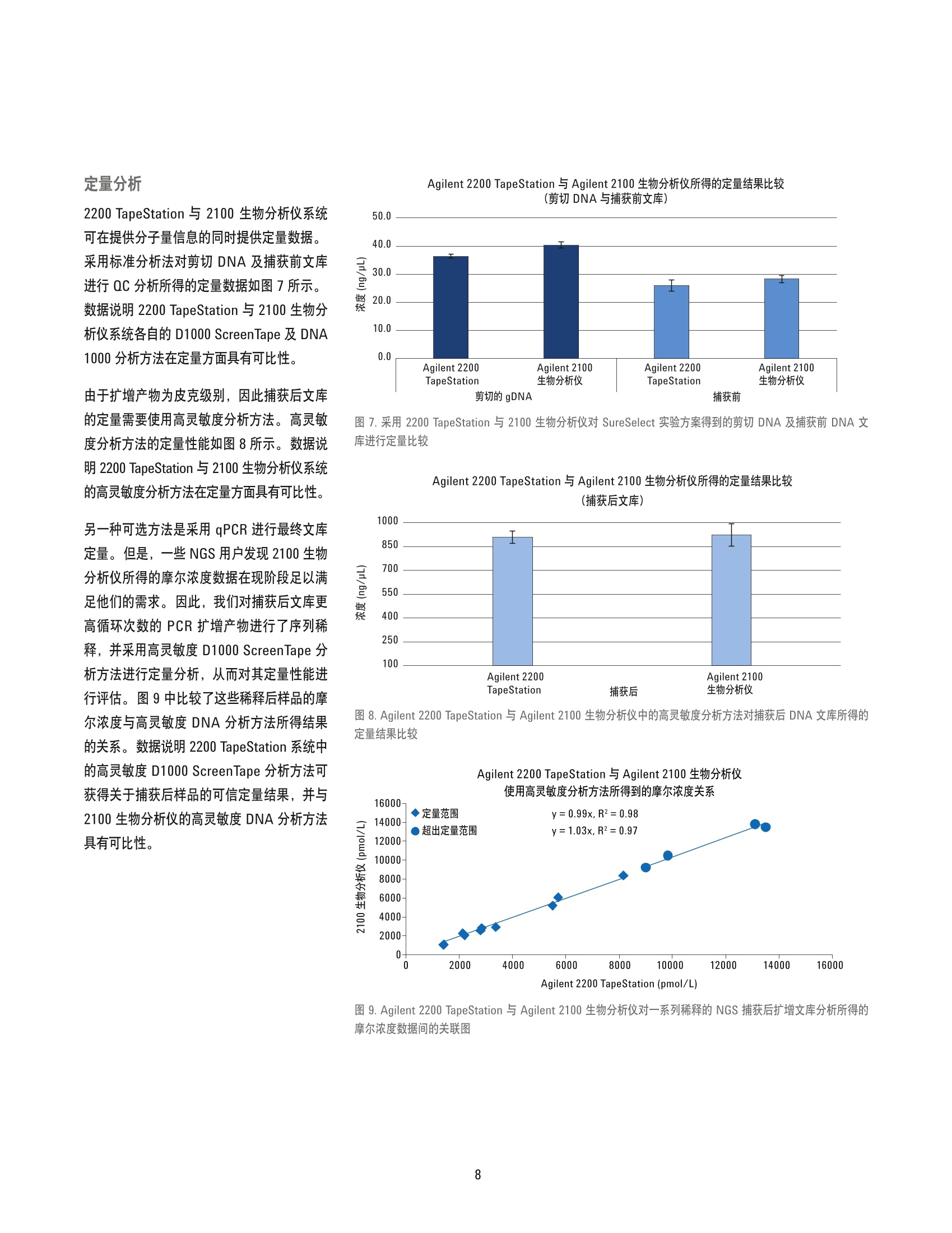
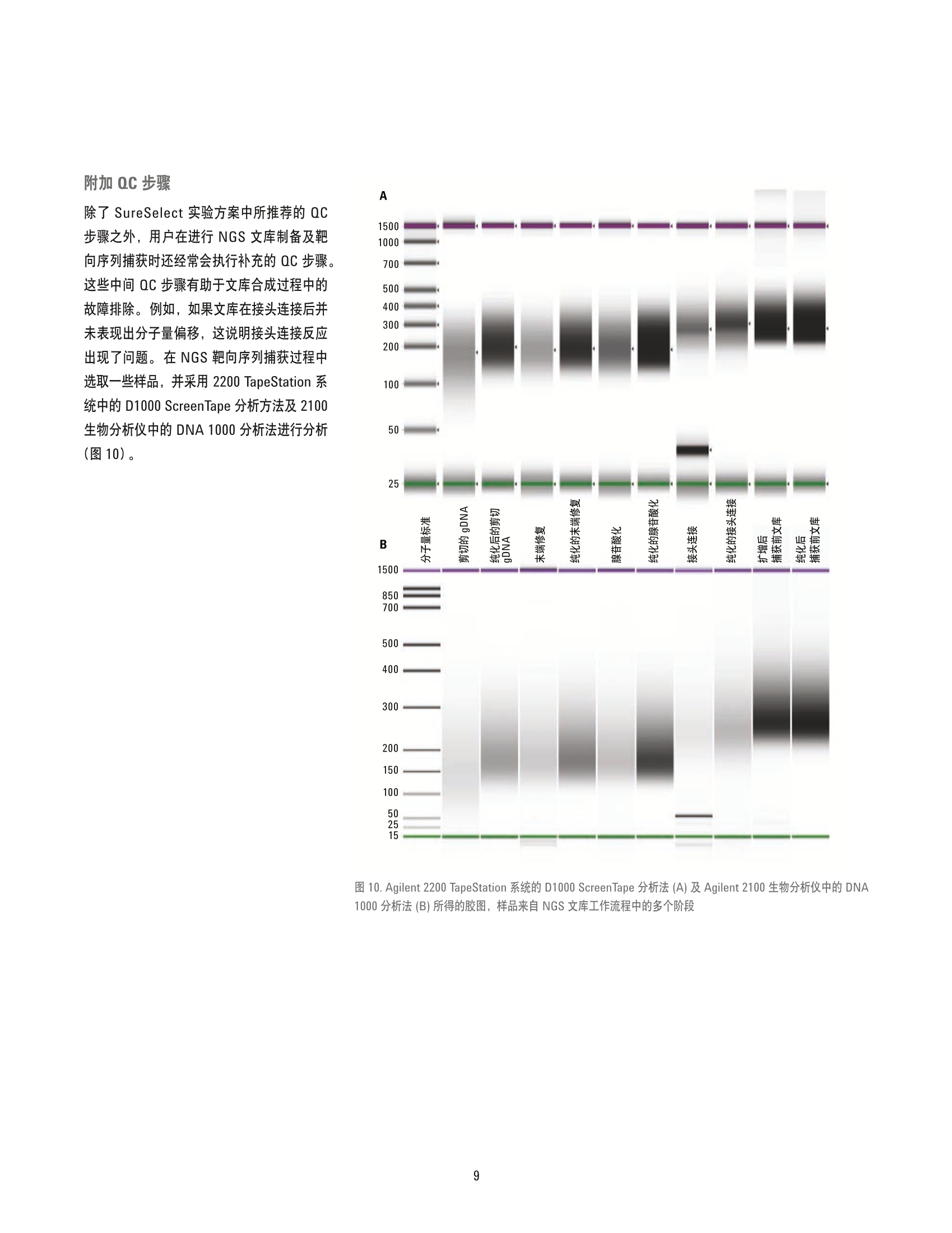
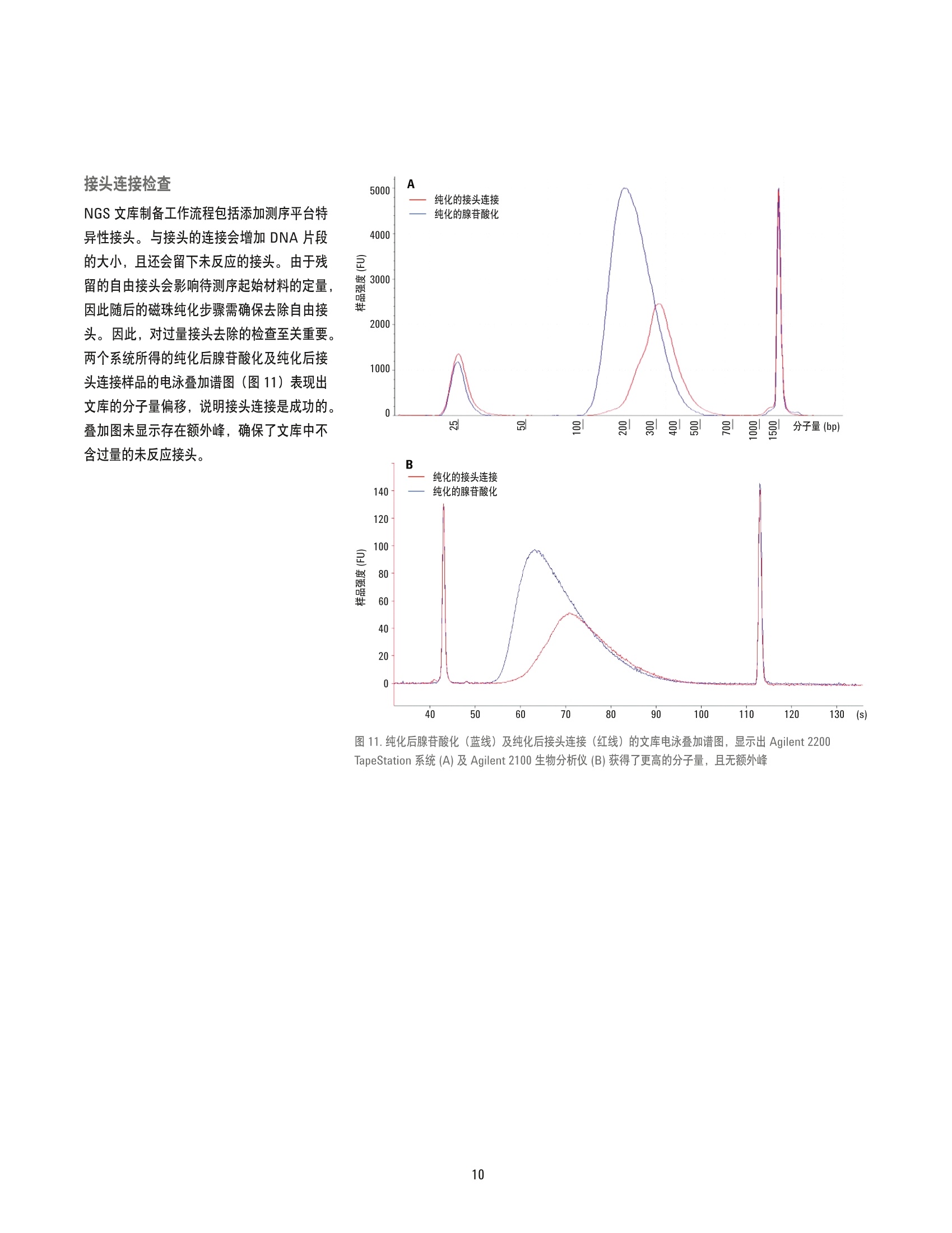
捕获后文库 利用 Agilent 2200 TapeStation 系统为新一代测序文库制备及 SureSelect 靶向序列捕获进行全程样品质量控制 应用简报核酸分析 作者 摘要 Arunkumar Padmanaban 安捷伦科技有限公司 印度班加罗尔 Agilent 2200 TapeStation 系统可对新一代测序 (NGS) 工作流程中的样品进行全程分析,从最初的起始材料料直到测序前的最终 DNA 文库质量控制(QC)。本应用简报遵循SureSelect 文库制备实验方案,展示了基因示 DNA ScreenTape、 D1000 ScreenTape及高灵敏度 D1000 ScreenTape 分析方法对该工作流程中样品的分析性能及适用性。数据证正了 2200 TapeStation 系统是可靠的文库分子量测定及定量分析 QC平台。数据还证明了这些 DNA ScreenTape 分析方法与 Agilent 2100 生物分析仪的相应分析方法具有可比性,并在其性能指标内。 Agilent Technologies 简介 基于 Sanger 方法的传统测序方法在过去的20多年间一直在 DNA序列测定方法中占据主导地位。但是,随着测序方法及技术的进步,测序通量在 Sanger 测序基础上有了极大的进步,如今全基因组测序可在几周甚至是几天内完成。这些技术统称为新一代测序, 或 NGS。 随着 NGS 技术和方法的进步,测序的费用也得到了大幅降低。但是,测序样品的制备依然十分复杂且耗时。因此,需要对 NGS的样品制备进行仔细监控以确保能成功进行测序实验。 Agilent SureSelect 靶向序列捕获试剂盒通过各种捕获组(包括全外显子、基因组合、蛋白激酶组、定制及其他种类),能为靶标特异性测序提供完整、经验证的样品制备工作流程。文库制备包括分离遗传物质、剪切 DNA以获取更短的片段、加入测序平台特异性的接头、杂交以富集靶标,以及多重测序标记。整个制备工作流程需要在多个步骤进行质量控制,以确保在测序前成功获得文库。工作流程概览如图1所示。 Agilent 2200 TapeStation 系统是用于 NGS文库制备工作流程解决方案质量和数量评估的理想 QC 平台。2200 TapeStation 系统提供一个自动化电泳系统,可对样品进行快速分析,只需1到2分钟时间即可完成一个样品分析。本应用简报介绍了按照2200TapeStation 系统中的 SureSelect 实验方案,采用基因组 DNA ScreenTape、D1000ScreenTape 及高灵敏度 D1000 ScreenTape分析方法对 NGS 文库制备进行质量控制。 A 图1 A) Agilent SureSelect靶向序列捕获系统工作流程概览,适用于 Illumina 末端配对测序文库制备,突出显示了其中的 QC 步骤。B) SureSelect 文库制备工作流程示意图,详述了推荐的 QC步骤(黄色),分别采用相应的 Agilent 2200 TapeStation分析方法(深蓝色) 及 Agilent 2100 生物分析仪方法(浅蓝色) 材料与方法 QC分析购自 ATCC 的 HEK293细胞系用含有10%FBS 及1%青霉素/链霉素(Pen/Strep)的MEM培养基在 37°℃、含5% CO,培养箱内培养。收细胞后,采用安捷伦 DNA 提取试剂盒 (200600)提取基因组 DNA。 Agilent2200 TapeStation 系统 (G2964AA)、基因组 DNA ScreenTape 及试剂(部件号5067-5365及5067-5366)、D1000 ScreenTape及试剂(部件号5067-5582及5067-5583)、高灵敏度 D1000 ScreenTape 及试剂(部件号5067-5584及5067-5585)、2100生物分析仪 (G2939AA)、 DNA 1000 (部件号5067-1504 及5067-1505)及高灵敏度 DNA试剂盒(部件号5067-4626及5067-4627)均来自安捷伦科技有限公司。应用 Qubit 1.0荧光光及 NanoDrop 1000 (赛默飞世尔)进行比较QC分析。并采用 S220自动聚焦声波样本处理仪 (Covaris公司)进行基因组DNA剪切, 该仪器由 Genotypic 科技友情提供。 NGS 文库制备 对于标准的3pg 起始 DNA, 我们采用适用于 Illumina 配对末端测序文库(版本1.6)的 Agilent SureSelect^靶向序列捕获系统实验方案中的指南,制备适用于Illumina HiSeq 及 MiSeq 多重测序平台的NGS文库。 SureSelect 试剂盒 (G9611A)及SureSelect"人全外显子 V4+UTR (5190-4636)捕获文库来自安捷伦科技公司,并遵照生产商的实验方案使用。Agencourt AMPureXP试剂盒(贝克曼库尔特基因组学公司)及 Dynabeads MyOne 链霉亲和素 T1 (LifeTechnologies) 分别购自对应的生产商,并根据各自的指南进行使用。通常,用于 NGS 文库制备的 SureSelect 实验方案第一步为提取基因组 DNA。根据推荐的剪切设置,应用Covaris 超声仪对来自 HEK293 细胞团块的3 ug 基因组 DNA进行剪切,然后进行纯化。对纯化后的片段在3'端进行末端修复及腺苷酸化,并采用磁珠进一步纯化。然后在片段的3'端连接上 Illumina 平台特异性接头,并 通过纯化去除过量的未连接接头。将连接上接头的片段与生物素化的 RNA 文库(捕获诱饵) 在65℃下杂交16小时以富集靶标。接着扩增所捕获的文库,并插入标签进行多重测序。 所有样品在2200 TapeStation 及2100 生物分析仪中均重复分析三次。 结果与讨论 ( 现行的 NGS技术通常需要获取待测序的短片段 DNA 文库。本文中详述的 Illumina 测序实验方案实例显示, DNA 的起始片段大小推荐在150-200基基对之间 。 如图1A和图1B中所示,需要若干步骤以完全制备DNA用于测序。QC 测试是该工作流程中的关键步骤,可确保成功获取文库。在 NGS平台测序前, 2200 TapeStation 系统可检查起始基因组 DNA的完整性,并评价多个纯化步骤,直至最终 QC步骤的样品质量。 ) A 基因组DNA 纯化后的剪切 DNA 捕获前文库 图2. A: 由 Agilent 2200 TapeStation 系统所得的基因组 DNA、剪切 DNA、捕获前及捕获后文库的QC步骤胶图,分别采用基因组 DNA、 D1000 及高灵敏度 D1000ScreenTape 分析。B: 由Agilent 2100生物分析仪所得的剪切 DNA、捕获前及最终文库的QC步骤胶样图,分别采用 DNA 1000 及高灵敏度 DNA 分析方法。 Agilent2100生物分析仪系统不适合用于基因组DNA分析 起始基因组 DNA材料的数量及完整性检查 成功的 NGS 文库制备取决于起始样品的质量,在本文中为基因组 DNA。采用基因组 DNAScreenTape 分析方法对基因组 DNA 的质量和数量进行分析。通常,采用 NanoDrop测定 OD260/280比值以评价样品的纯度质量,该比值需在1.8到2.0之间, 而 Qubit系统则用于定量基因组 DNA。相比之下,2200 TapeStation 分析方法可在样品 QC步骤中同时提供样品的质量和数量信息。采用 TapeStation 分析软件所得的完整胶条(无任何降解产物)的图片可测定样品的纯度(图3A)。图 3B中展示了2200 TapeStation系统、NanoDrop 及 Qubit 所得的数据,结果显示2200 TapeStation 系统和 Qubit 在定量方面具有可比性。采用 UV 光谱法测定基因组 DNA 所得的量常会过高,这是由于其他的缓冲液成分在 UV光谱中也可能有吸收"。因此,与其他系统不同的是,基因组 DNAScreenTape 分析方法及2200 TapeStation系统只需1pL样品即可在一步分析中同时对质量和数量进行评价。 B 基因组 DNA 定量比较 图3. A) Agilent 2200 TapeStation 系统 ScreenTape 分析所得基因组 DNA 冬图。基因组 DNA ScreenTape分析方法显示出样品是完整的, NanoDrop 测定的 A260/280 比值对其进行了验证(如每条泳道下所示)。 B)基因组 DNA ScreenTape 分析方法所得定量数据与 Qubit 及 NanoDrop 所得结果比较 除了对起始材料的初始基因组 DNA 进行 QC之外, SureSelect 实验方案还推荐在文库制备及靶向序列捕获过程中增加两到三个检查点以确保样品的正确质量(图1): 对经非强制纯化及大小选择的剪切基因组 DNA 进行QC,以检查片段具有单个弥散条带分布,中值分子量在150到200 bp之间。 对接头连接的、经扩增及纯化后的接头连接 DNA 文库进行均匀弥散条带分布检查,中值分子量在225到275 bp之间。该步骤常称为捕获前 QC。 推荐在测序前对杂交后文库进行最终QC (捕获后QC)。杂交后文库的中值分子量范围需为 250至350 bp。 在上述每个步骤中取样,并应用2200 TapeStation 重复分析三次以评价其在这些文库的质量控制中的性能。并同2100生物分析仪的性能进行比较。 分子量分析 如上所述,剪切 DNA、捕获前及捕获后 DNA样品均同时采用 2200 TapeStation 及 2100生物分析仪进行分析。三个 QC步骤的样品重复分析三次,整理后的数据如图4所示。图中的数据展现了两个系统所得的文库分子量有很好的相关性。 初始剪切 DNA 及捕获前扩增文库采用2200TapeStation 系统中的 D1000 ScreenTape分析方法及2100生物分析仪的 DNA 1000分析方法进行分析。 实验方案建议初始剪切 DNA 样品弥散条带的大部分应在 150-200 bp之间。采用2200TapeStation 分析软件进行分子量评价,电泳图谱(图5A)说明了弥散条带的大部分落在了推荐的范围内,证明了基因组 DNA 得到了成功的剪切。该电泳图图也也2100生物分析仪所得曲线相似(图5B)。 第二个 QC 步骤评价了与捕获诱饵杂交前的捕获前扩增文库的质量。SureSelect 实验方案指出弥散条带平均分子量应在 225-275 bp之间。对捕获前扩增文库的QC分析有助于在杂交之前发现接头连接中的所有可能问题,从而节省时间及试剂的消耗。电泳图谱表现为一个单峰,平均分子量约为250bp,说明接头连接是成功的(图5C及5D分别代表2200 TapeStation 及 2100生物分析仪系统的结果)。 捕获后扩增文库同时由2200 TapeStation系统中的高灵敏度 D1000 ScreenTape分析方法及2100生物分析仪系统中的高灵敏度DNA 分析方法进行分析。SureSelect 实验方案建议杂交后文库的中值分子量范围应为250 至350bp。经两个系统分析后的样品所得的电泳图谱如图6所示。电泳图谱表现为一个单峰,平均分子量约为280 bp, 说明成功扩增了文库。 Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪系统所得片段分子量比较 图 4. Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪系统所得的片段分子量比较。对剪切 DNA、捕获前文库及捕获后文库三次重复分析所得分子量平均值进行了比较。误差线表示标准偏差 Agilent 2200 TapeStation 系统 Agilent 2100生物分析仪 图5.由Agilent 2200 TapeStation(左列)及 Agilent 2100 生物分析仪(右列)所得纯化后剪切 DNA (A和B),及捕获前文库(C和D)的电泳图谱 图 6. Agilent 2200 TapeStation 系统所得的捕获后扩增文库电泳图谱(A)显示了283 bp 上的峰。相同样品在 Agilent 2100 生物分析仪(B)中表现为 287 bp 上的峰 7 2200 TapeStation 与 2100 生物分析仪系统可在提供分子量信息的同时提供定量数据。采用标准分析法对剪切 DNA 及捕获前文库进行QC分析所得的定量数据如图7所示。数据说明 2200 TapeStation 与2100生物分析仪系统各自的 D1000 ScreenTape 及 DNA1000分析方法在定量方面具有可比性。 由于扩增产物为皮克级别,因此捕获后文库的定量需要使用高灵敏度分析方法。高灵敏度分析方法的定量性能如图8所示。数据说明2200 TapeStation 与2100生物分析仪系统的高灵敏度分析方法在定量方面具有可比性。 另一种可选方法是采用 qPCR 进行最终文库定量。但是,,一些 NGS 用户发现2100生物分析仪所得的摩尔浓度数据在现阶段足以满足他们的需求。因此,我们对捕获后文库更高循环次数的 PCR 扩增产物进行了序列稀释,并采用高灵敏度 D1000 ScreenTape 分析方法进行定量分析,从而对其定量性能进行评估。图9中比较了这些稀释后样品的摩尔浓度与高灵敏度 DNA分析方法所得结果的关系。数据说明 2200 TapeStation 系统中的高灵敏度 D1000 ScreenTape 分析方法可获得关于捕获后样品的可信定量结果,并与2100 生物分析仪的高灵敏度 DNA分析方法具有可比性。 Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪所得的定量结果比较(剪切 DNA与捕获前文库) 图7.采用2200 TapeStation 与2100生物分析仪对 SureSelect 实验方案得到丁剪切 DNA 及捕获前 DNA 文库进行定量比较 Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪所得的定量结果比较(捕获后文库) 图 8. Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪中的高灵敏度分析方法对捕获后 DNA 文库所得的定量结果比较 Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪 图 9.Agilent 2200 TapeStation 与 Agilent 2100 生物分析仪对一系列稀释的 NGS 捕获后扩增文库分析所得的摩尔浓度数据间的关联图 附加 QC 步骤 除了 SureSelect 实验方案中所推荐的 QC步骤之外,用户在进行NGS 文库制备及靶向序列捕获时还经常会执行补充的QC 步骤。这些中间 QC步骤有助于文库合成过程中的故障排除。例如,如果文库在接头连接后并未表现出分子量偏移,这说明接头连接反应出现了问题。在 NGS 靶向序列捕获过程中选取一些样品,并采用 2200 TapeStation 系统中的 D1000 ScreenTape 分析方法及2100生物分析仪中的DNA 1000分析法进行分析(图10)。 图 10. Agilent 2200 TapeStation 系统的 D1000 ScreenTape 分析法 (A) 及 Agilent 2100 生物分析仪中的 DNA1000分析法(B)所得的胶图,样品来自 NGS 文库工作流程中的多个阶段 接头连接检查 NGS 文库制备工作流程包括添加测序平台特异性接头。与接头的连接会增加 DNA片段的大小,且还会留下未反应的接头。由于残留的自由接头会影响待测序起始材料的定量,因此随后的磁珠纯化步骤需确保去除自由接头。因此,对过量接头去除的检查至关重要。两个系统所得的纯化后腺苷酸化及纯化后接头连接样品的电泳叠加谱图(图11)表现出文库的分子量偏移,说明接头连接是成功的。叠加图未显示存在额外峰,确保了文库中不含过量的未反应接头。 (s) 图11.纯化后腺苷酸化(蓝线)及纯化后纯头连接(红线)的文库电泳叠加谱图, 显叠出 Agilent 2200TapeStation 系统 (A) 及 Agilent 2100 生物分析仪(B)获得了更高的分子量,且无额外峰 杂交后 PCR 扩增优化 对捕获后文库的 PCR 扩增,通常需要优化其扩增循环的次数。扩增不足会导致进行测序的 DNA数量不足。过分扩增则会导致重复的增加,将会引起测序覆盖率的偏移。捕获后文库分别采用5、8和10次PCR循环进行扩增,并采用高灵敏度 D1000 ScreenTape分析方法进行分析。样品的电泳叠加谱图(图12)显示了 PCR 循环次数对所产生的 DNA 数量的影响。从本研究可以看出,由于分析时间短,样品需求量小,2200TapeStation 系统可有效用于优化 PCR循环次数。 图 12. 采用 Agilent 2200 TapeStation 系统分析经过5、8和10次PCR循环扩增(分别为蓝线、绿线及红线)得到的浓度逐渐增加的经扩增的捕获后文库 DNA 通过结合 SureSelect 实验方案,本应用简报说明了 Agilent 2200 TapeStation 系统结合基因组 DNA、D1000 及高灵敏度 D1000ScreenTape 分析方法是分析 NGS文库制备工作流程所得样品的理想选择。 基因组 DNA Screen Tape 分析方法不仅能评估基因组 DNA起始材料的质量,还能对数量进行评估,使用单个测试就能替代原先两个独立的测试。 SureSelect 靶向序列捕获工作流程所得的 NGS 样品的分子量可通过联合D1000 ScreenTape分析方法及2200TapeStation 系统进行可靠测定。所测得的分子量与 Agilent 2100 生物分木仪中类似分析法所得结果相匹配。 最终文库的定量可由 2200 TapeStation系统中的高灵敏度 D1000 ScreenTape分析法完成,结果与2100生物分析仪的高灵敏度 DNA分析方法所得结果一致。 ( 1.0 O. Morozova, M.A. Marra "Applications of next-generation sequencing technologies i n functional genomics"Genomics, Volume 92, Issue 5, November 2008, p. 255-264,ISSN 0888-7543. http://dx.doi.org/10.1016/j.ygeno.2008.07.001. ) 2.““An Introduction to Next-GenerationSequencing Technology"11http://res.illumina.com/documents/products/illumina_sequencing_introduction.pdf ( 3. S ureSelectXT Target E nrichmentSystem for Illumina Paired-EndSequencing Library - Illumina HiSeq and MiSeq Multiplexed Sequencing Platforms, Protocol 1.6(用于 Illumina配对末端测序文库的 SureSelectXT靶向序列捕获系统系 IluminaHiSeq 及 MiSeq 多重测序平台 , 实验方案1.6), http://www.chem. agilent.com/library/usermanuals/ Public/G7530-90000 SureSelect IlluminaXTMultiplexed_1.6.pdf ) ( 4. M .O'Neill, J. M cPartlin, K. Arthure, S. R iedel, and Nd McMillan, "Comparison of the T LDA with theNanodrop and the reference Qubit system" J. Physics: Conference Series Volume 307, Issue 1,2011, http://iopscience.iop.org/1742- 6596/307/1/012047. ) 摘要Agilent 2200 TapeStation 系统可对新一代测序 (NGS) 工作流程中的样品进行全程分析,从最初的起始材料一直到测序前的最终 DNA 文库质量控制 (QC)。本应用简报遵循 SureSelect 文库制备实验方案,展示了基因组 DNA ScreenTape、D1000 ScreenTape 及高灵敏度 D1000 ScreenTape 分析方法对该工作流程中样品的分析性能及适用性。数据证明了 2200 TapeStation 系统是可靠的文库分子量测定及定量分析 QC 平台。数据还证明了这些 DNA ScreenTape 分析方法与 Agilent 2100 生物分析仪的相应分析方法具有可比性,并在其性能指标内。简介基于 Sanger 方法的传统测序方法在过去的 20 多年间一直在 DNA 序列测定方法中占据主导地位。但是,随着测序方法及技术的进步,测序通量在 Sanger 测序基础上有了极大的进步,如今全基因组测序可在几周甚至是几天内完成2。这些技术统称为新一代测序,或 NGS。随着 NGS 技术和方法的进步,测序的费用也得到了大幅降低。但是,测序样品的制备依然十分复杂且耗时。因此,需要对 NGS 的样品制备进行仔细监控以确保能成功进行测序实验。Agilent SureSelect 靶向序列捕获试剂盒通过各种捕获组(包括全外显子、基因组合、蛋白激酶组、定制及其他种类),能为靶标特异性测序提供完整、经验证的样品制备工作流程。文库制备包括分离遗传物质、剪切 DNA 以获取更短的片段、加入测序平台特异性的接头、杂交以富集靶标,以及多重测序标记。整个制备工作流程需要在多个步骤进行质量控制,以确保在测序前成功获得文库。工作流程概览如图 1 所示。Agilent 2200 TapeStation 系统是用于 NGS 文库制备工作流程解决方案质量和数量评估的理想 QC 平台。2200 TapeStation 系统提供一个自动化电泳系统,可对样品进行快速分析,只需 1 到 2 分钟时间即可完成一个样品分析。本应用简报介绍了按照 2200 TapeStation 系统中的 SureSelect 实验方案,采用基因组 DNA ScreenTape、D1000 ScreenTape 及高灵敏度 D1000 ScreenTape 分析方法对 NGS 文库制备进行质量控制。结论通过结合 SureSelect 实验方案,本应用简报说明了 Agilent 2200 TapeStation 系统结合基因组 DNA、D1000 及高灵敏度 D1000 ScreenTape 分析方法是分析 NGS 文库制备工作流程所得样品的理想选择。• 基因组 DNA ScreenTape 分析方法不仅能评估基因组 DNA 起始材料的质量,还能对数量进行评估,使用单个测试就能替代原先两个独立的测试。• SureSelect 靶向序列捕获工作流程所得的 NGS 样品的分子量可通过联合 D1000 ScreenTape 分析方法及 2200 TapeStation 系统进行可靠测定。所测得的分子量与 Agilent 2100 生物分析仪中类似分析法所得结果相匹配。• 最终文库的定量可由 2200 TapeStation 系统中的高灵敏度 D1000 ScreenTape 分析法完成,结果与 2100 生物分析仪的高灵敏度 DNA 分析方法所得结果一致。
确定





还剩10页未读,是否继续阅读?
安捷伦科技(中国)有限公司为您提供《生物样品中DNA 测序检测方案(生物芯片)》,该方案主要用于其他中DNA 测序检测,参考标准--,《生物样品中DNA 测序检测方案(生物芯片)》用到的仪器有Agilent 2100 生物分析仪
推荐专场
相关方案
更多
该厂商其他方案
更多