推荐厂家
暂无
暂无
 留言咨询
留言咨询
 留言咨询
留言咨询
 400-860-5168转4423
400-860-5168转4423
 留言咨询
留言咨询

 400-611-9236
400-611-9236
 留言咨询
留言咨询
 400-629-8889
400-629-8889
 留言咨询
留言咨询

 400-875-8210
400-875-8210
 留言咨询
留言咨询

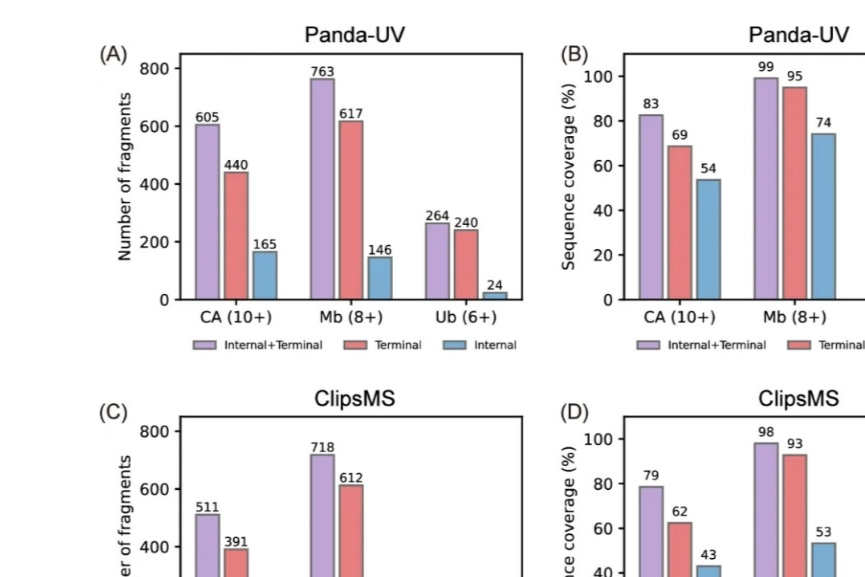




求常见毒物的碎片离子表
质谱图中碎片离子为何表观是棒图,而实际是峰形,比如在计算分辨率的时候,谢谢
你在使用质谱分析过程中,无论是在相关文献还是在仪器原理或使用说明中都往往会提到“离子碎片”这个概念,请问您对粒子碎片这个概念怎么理解?你认为离子碎片到底应该是怎样的?它是怎么形成的?质谱对它是如何辨识的?


