







中山大学药学院教授,博士生导师。主要从事生物质谱新技术的开发及应用。


大家好,本周为大家分享一篇发表在Journal of Proteome Research上的文章,Template-Assisted De Novo Sequencing of SARS-CoV‑2 and Influenza Monoclonal Antibodies by Mass Spectrometry [1],文章的通讯作者是德克萨斯大学生物医学研究支持中心的Maria D. Person。
抗体在对病毒和其他病原微生物的适应性免疫反应中起着重要作用。对病毒中和抗体的详细表征对于开发能够有效预防病毒疾病的新疫苗和单克隆抗体疗法至关重要。目前,仅从蛋白质中获得抗体的完整氨基酸序列仍然具有挑战性,且许多用于氨基酸从头测序的软件对计算、硬件及操作人员知识和经验的要求较高。另一种蛋白基因组学方法是使用来自DNA序列的数据库作为起点,以数据库序列和实验数据之间的最佳匹配作为模板。这种方法在分析抗体时特别有用,因为大部分抗体序列(不包括CDR3区域)通常与种系序列非常相似。此外,准确区分Ile和Leu在抗体CDR区测序时尤为重要,而大多数蛋白质从头测序软件都没有解决这个问题。Supernovo是从种系序列模板开始的测序程序之一,该模板可以利用ETD和EThcD片段数据来自动区分Ile/Leu。在这项研究中,作者利用SARS-CoV-1人类抗体来开发模板辅助从头测序的方案,对两株SARS-CoV-2人类单克隆抗体进行了测定,最终优化的方案使用四种蛋白酶、双片段化方法、优化补充激活归一化碰撞能量和Supernovo获得99%准确的氨基酸序列,并以该方案对25株流感人单抗进行了测序。
一、模板辅助从头测序方法的建立
Supernovo的工作原理是,首先将MS/MS数据与种系数据库中的序列进行匹配,在初始种系序列选择之后,使用通配符搜索来确定接合(J)区域,以与可变区和恒定区候选最佳结合,然后进行in silico重组以生成模板。接下来,限制的从头测序和Byonic通配符搜索通过在迭代过程中改变单个氨基酸来改进模板,实现实验数据最佳匹配。最初由种系序列确定的Ile和Leu残基通过蛋白酶切割特异性和w离子的存在进行修正。利用多种蛋白酶从基因组数据库中选择种系支架序列,并通过通配符搜索获得从头测序所需的各种肽。胰蛋白酶和糜蛋白酶是根据其不同的切割特异性选择的,并选择特异性较低的弹性蛋白酶和胃蛋白酶提供额外的切割位点。由于Supernovo利用来自特异性和非特异性裂解肽的所有信息来构建序列,因此具有许多重叠的裂解位点对于序列测定至关重要。
作者首先用序列已知的重组单抗CR3022制定和优化方案。CR3022用胰蛋白酶、糜蛋白酶、弹性蛋白酶和胃蛋白酶酶切,用HCD和EThcD LC-MS/MS分析酶切溶液。实验显示使用EThcD可以在不针对选定的单个z离子的情况下全局碎裂肽混合物,并且产生足够的w离子来区分Ile/Leu。b、c、y、z和w离子的产率取决于SA-NCE, 35%的SA-NCE值产生了最高数量的高置信度Ile/Leu分配,并被选择用于最终方案。图1显示了两种含有Leu和Ile的重链肽,其中检测到w离子z-43(Leu)和z-29(Ile),从而允许Supernovo区分Ile和Leu。虽然在碎裂谱中可以检测到w离子,但使用单一SA-NCE值的宽带碎裂,其丰度较低,这是该方法的主要限制。将多重断裂能量相结合,可以提高对w离子的检测,而使用额外的蛋白酶增加切割位点的数量,有助于分离位置相近的Ile/Leu。
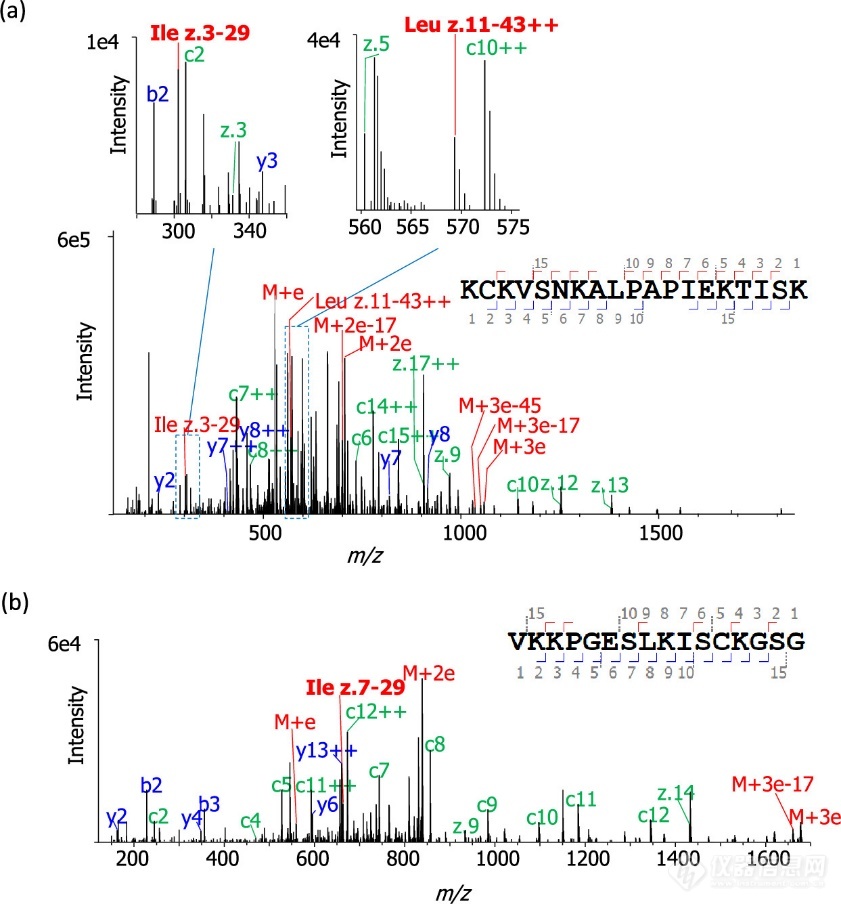
图1 使用EThcD对Ile/Leu进行MS/MS分配
图2显示了用胰蛋白酶、糜蛋白酶、胃蛋白酶和弹性蛋白酶酶切的重链Fab区域在HCD碎裂下的序列覆盖图。由于四种蛋白酶的不同切割特异性以及Supernovo对非特异性切割肽的匹配,可以观察到肽在许多氨基酸残基上的切割。除脯氨酸外,大多数残基的MS/MS中都观察到对应碎片离子。图3显示了DNA确定的参考CR3022序列(DNA sq)、通过将IMGT数据库中的序列片段与MS数据(SNO-gm)进行数据库匹配而识别的种系模板,以及通过对抗体可变区Fab的MS数据(SNO-MS)进行Supernovo分析而生成的最终从头序列之间的比较。所有CDR区域均实现100%正确的氨基酸序列(灰色高亮)覆盖,具有正确的Ile/Leu分配。Supernovo的错误分配(蓝色高亮)主要发生在HC和LC上的N-末端残基以及LC上的C-末端序列。与DNA衍生序列不匹配的外来N和 C-末端残基均与初始种系支架存在偏差,如图3中重链末端的洋红高亮所示。末端氨基酸具有较少的裂解和片段数据,并且可能具有混杂的修饰。半胱氨酸残基可导致附近氨基酸的错误分配,因为半胱氨酸硫的氧化修饰。总的来说,99%的单克隆抗体序列是通过这种方法正确测定的。结合HCD和EThcD数据,68个Ile/Leu中有67个被正确分配。
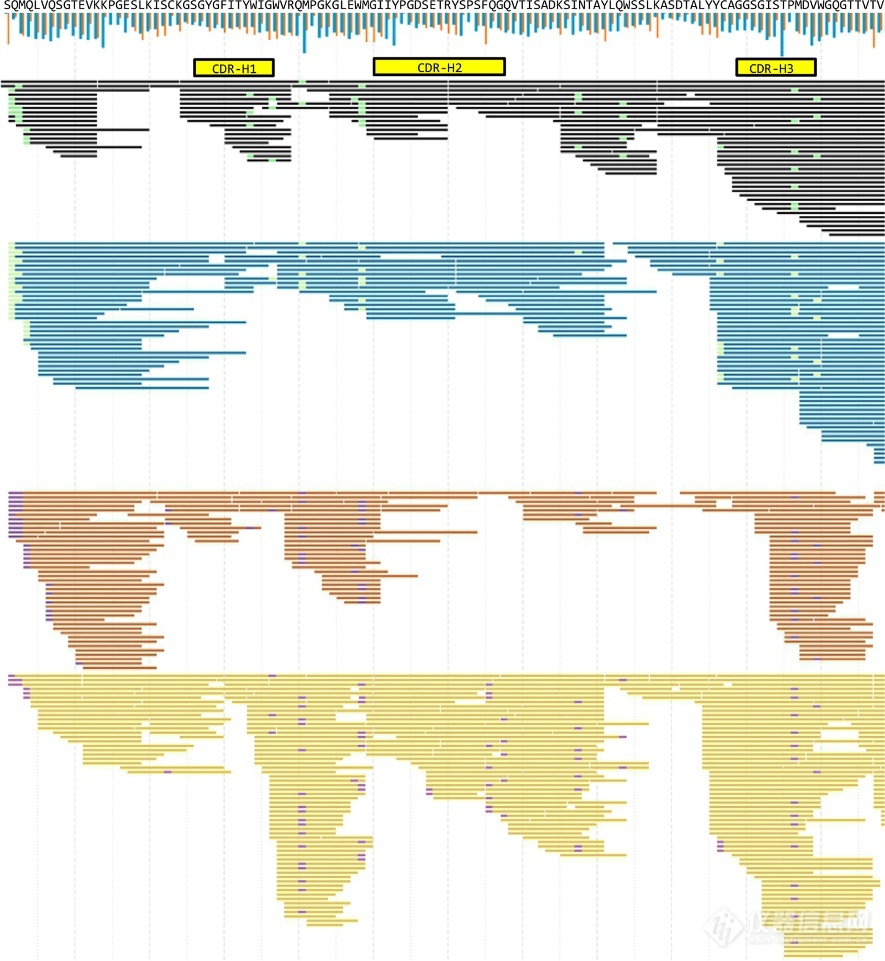
图2 用胰蛋白酶(黑色)、糜蛋白酶(蓝色)、弹性蛋白酶(红色)和胃蛋白酶(黄色)酶切并用HCD裂解的CR3022重链Fab的肽覆盖图。

图3 对CR3022 DNA衍生序列(DNA sq)、MS Supernovo种系模板(SNO-gm)和Fab区域的实验结果(SNO-MS)进行比对
接着,作者使用两种具有DNA衍生序列的SARS-CoV-2抗体CM66和CM67来测试该方案的最终版本(即四蛋白酶酶切,HCD和EThcD活化,SA-NCE 35%)。表1总结了该方法对已知序列的三种抗体CR3022、CM66和CM67的测试结果。CDR区氨基酸在HC和LC中的分配均正确,只有两个Ile错误分配给Leu。在高通量方法中,产生足够强度的诊断性w离子仍然不一致,但当与蛋白水解切割偏好和种系模板结合使用时,该方法是有效的,至少提供97%的正确Ile/Leu分配。因此,四酶酶切双活化法被确定足以对抗体进行高度准确的序列测定。

表1 受试单抗的氨基酸序列和Ile/Leu分配置信水平以及正确匹配的总结
二、基于质谱的25种流感抗体分析产生高置信度序列
作者用最终确定的方案分析了另外25种序列信息不确定的抗体,以评估该方法在更大规模的实验下是否能产生与SARS-CoV-1和2抗体相同水平的高置信度结果,并最终确定生成的序列是否可用于产生克隆和表达功能性抗体的基因。对于25株流感单克隆抗体,平均97%的序列和76%的Ile/Leu分配可在高置信度下确定。大多数置信度较低的氨基酸分配位于N端和C端,因此不太可能影响抗体结合。与半胱氨酸残基相邻的序列也有较高的中低置信度分配和偏离种系序列的发生率。在25株单克隆抗体中的24株中,两条链上三个CDR区域的所有残基都得到了高度可信的鉴定。由于该方法不总是在可检测水平上产生可区分的w离子,CDR区域具有一些中低置信度Ile/Leu分配。因此,当大规模应用于各种IgG抗体时,该方案提供了高置信度氨基酸序列。获得的流感单克隆抗体序列随后用于通过克隆和在HEK293细胞中表达产生抗体。
三、流感病毒HA特异性人单克隆抗体的生物学活性及序列分析
在克隆、表达和纯化后,通过ELISA评估原始25种流感病毒和相应重组表达的HA特异性单克隆抗体的生物活性。图4显示了25种单克隆抗体与原始和重组单克隆抗体的HA抗原的结合曲线。通过评估单克隆抗体对相应HA抗原的结合活性,证实了单克隆抗体对H1N1、H3N2和B型流感病毒HA的特异性。如图4所示,流感特异性单克隆抗体表现出与季节性和大流行性流感病毒株的重组HA(rHA)的差异结合,与原始抗体显示的结合活性相当。
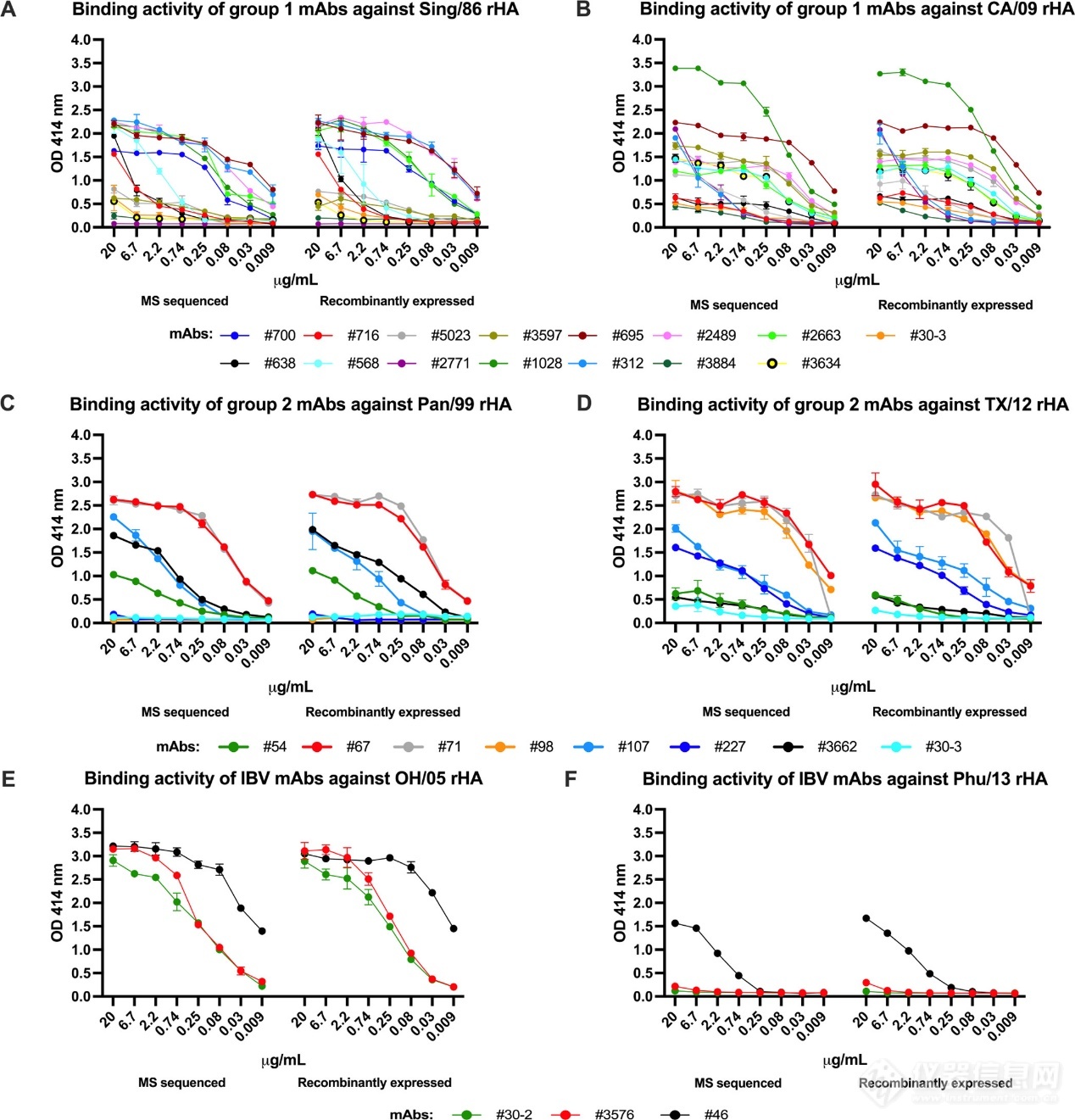
图4 原始MS测序(左曲线)和重组表达(右曲线)流感HA特异性单克隆抗体的ELISA结合曲线
总的来说,作者通过对25个未知序列单克隆抗体进行测序,确认所采用的方法在不同单克隆抗体上产生的残基测定置信度与已知序列或商业来源的单克隆抗体相同。蛋白质组学方法可以用来检查基于DNA的方法,这些方法被认为更准确,或者可以用来清除样本处理和标记中的错误。通过ELISA结合试验观察并验证了CDR序列的多样性。这些分析证实,当克隆和重组表达时,质谱模板辅助从头测序产生的高置信度序列会产生与特异性流感病毒rHA抗原结合的重组单克隆抗体,从而对该方法进行生物学验证。
撰稿:夏淑君编辑:李惠琳
文章引用: Template-Assisted De Novo Sequencing of SARS-CoV-2 and Influenza Monoclonal Antibodies by Mass Spectrometry.
[来源:仪器信息网] 未经授权不得转载

2022.05.12
生态环境中心刘倩等成果:单颗粒质谱追踪大气颗粒物的体内暴露途经
2024.07.03

增长的前沿,也是我们的舞台——2024夏季达沃斯论坛归来后的一些思考
2024.07.03

2024.07.02

2024.07.01

专家解读|GB 2763.1-2022中农药残留限量配套检测方法修订情况
2024.06.28
版权与免责声明:
① 凡本网注明"来源:仪器信息网"的所有作品,版权均属于仪器信息网,未经本网授权不得转载、摘编或利用其它方式使用。已获本网授权的作品,应在授权范围内使用,并注明"来源:仪器信息网"。违者本网将追究相关法律责任。
② 本网凡注明"来源:xxx(非本网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责,且不承担此类作品侵权行为的直接责任及连带责任。如其他媒体、网站或个人从本网下载使用,必须保留本网注明的"稿件来源",并自负版权等法律责任。
③ 如涉及作品内容、版权等问题,请在作品发表之日起两周内与本网联系,否则视为默认仪器信息网有权转载。
![]() 谢谢您的赞赏,您的鼓励是我前进的动力~
谢谢您的赞赏,您的鼓励是我前进的动力~
打赏失败了~
评论成功+4积分
评论成功,积分获取达到限制
![]() 投票成功~
投票成功~
投票失败了~