naica®微滴芯片数字PCR系统精准检测不同西瓜种质之间CITST2基因拷贝数的差异
导读西瓜 (Citrullus lanatus, 2n=22)是世界第三大水果,是世界各地种植的重要经济作物,也是一种受欢迎的新鲜水果,含有糖、番茄红素和瓜氨酸等有益人体健康的化合物。研究人员通过杂交开发了大量西瓜品种以满足消费者的偏好。但长期栽培和对果实品质性状的筛选,不同地理区域采集的栽培西瓜显示出较低的遗传多样性,因此需要更多的遗传种质资源用于可持续生产西瓜的创新品种。参考基因组对于性状和基因发现是必不可少的。西瓜基因组测序工作始于十几年前。除西瓜基因组初稿外,自2019年以来已经发布了三个高质量的西瓜参考基因组。然而,每个基因组仍然不完整,存在许多空白。高质量的参考基因组与从相同遗传库产生的突变体数据相结合,将有助于发现和分离遗传和育种所需的突变体。无缺口参考基因组是基因组组装的最终目标,为识别“暗物质”区域中的独特基因和结构变异带来了新的机会。北京大学高等农业科学研究所科研人员在Molecular Plant(最新JCR分区Q1,影响因子21.9496)上发表了题为《A telomere-to-telomere gap-free reference genome of watermelon and its mutation library provide important resources for gene discovery and breeding》的文章。研究者使用西瓜优良近交系G42组装了一个T2T(telomere-to-telomere)无缺口参考基因组,弥补了当前可用参考基因组中所有剩余的组装缺口。通过基因组信息比对识别了甜西瓜种质中一个包含ClTST2(液泡膜糖转运蛋白)基因的17.5 kb串联重复序列(sv04611)。该研究应用naica®微滴芯片数字PCR系统对西瓜CITST2基因的拷贝数进行检测,证实了不同西瓜种质之间CITST2基因拷贝数存在差异,甜西瓜种质中CITST2基因的拷贝数增加可能是造成糖含量增加的原因。应用亮点:▶ 使用naica®微滴芯片数字PCR系统对甜西瓜和不甜西瓜种质之间CITST2基因拷贝数的差异进行准确定量。▶ 甜西瓜和不甜西瓜种质中CITST2基因分别为两个拷贝和单个拷贝。▶ CITST2基因的拷贝数变异可能会改变西瓜糖含量。G42基因组组装比其他参考基因组组装具有更高的完整性和准确性,为更准确地描述基因结构变异(SVs)提供了更多的数据支撑。数字PCR技术已被证明可以灵敏可靠地检测拷贝数变异的核酸绝对定量工具。该研究采用naica®微滴芯片数字PCR系统精确定量不同西瓜种质之间CITST2基因拷贝数的差异,验证了无缺口参考基因组识别SVs的准确性。研究成果:本研究成功组装出G42 西瓜的T2T无缺口参考参考基因组,包括所有22个端粒和11个着丝粒的信息。利用无缺口参考基因组数据,研究者识别了甜西瓜种质(97103和G42)sv04986结构中一个包含ClTST2基因的17.5 kb串联重复序列(sv04611)。这是不甜西瓜种质(PI 595203和PI 296341-FR)基因组中不存在的。ClTST2基因编码一种定位于液泡的糖转运蛋白,其表达与西瓜果肉中的糖积累呈正相关。▲图1. (A)sv04986 结构在甜西瓜(97103和 G42)和不甜西瓜(PI 595203和PI 296341-FR)种质中含有 ClTST2 基因。甜西瓜种质中存在一个17.5kb的串联重复序列sv04611,包含2bp的CA插入突变。红色箭头代表引物 ClTST2-R8/ClTST2-F3的位置。(B)使用ClTST2-R8/ ClTST2 -F3 引物扩增四个西瓜种质DNA 样本的结果。随后作者使用naica®微滴芯片数字PCR系统对甜西瓜和非甜西瓜种质之间CITST2基因拷贝数的差异进行了准确定量。FAM通道仅能检测到存在CA插入突变的区域。HEX通道用于检测ClTST2基因。CY5通道检测的是西瓜中的单拷贝内参基因Actin。当CITST2基因为双拷贝时,FAM/HEX/CY5拷贝数比约为1:2:1。当CITST2基因为单拷贝时,FAM/HEX/CY5拷贝数比约为0:1:1。数据显示两个不甜西瓜的种质仅包含单拷贝CITST2基因,而甜西瓜种质在包含两个拷贝CITST2基因。▲表1.利用dPCR技术估算甜和不甜西瓜种质中CITST2基因拷贝数期刊介绍:Molecular Plant (《分子植物》)是由中科院上海生命科学研究院植物生理生态研究所(IPPE)与中国植物生理与植物分子生物学学会(CSPP)主办,中科院上海生命科学信息中心生命科学期刊社承办的学术期刊,创刊于2008年。2022最新JCR分区Q1,影响因子21.9496。
应用实例
2022.09.27
还敢熬夜吗!Echo显微镜揭秘生物钟变化带来的影响
导读大家或许听到过这样一首歌:零点睡六点起,ICU里喝小米;一点睡六点起;阎王夸我好身体;两点睡觉六点起,骨灰盒子长方体;三点睡觉六点起,墓碑和我绑一起。熬夜如今已成为现代年轻人不可或缺的一种常态,明知熬夜伤身体,但管不住自己啊!生物钟是一种进化保守的转录-翻译调控网络,与身体代谢调节密切相关。从多数积累的实验模型和流行病学研究中的证据表明,生物钟调节机制的中断会导致肥胖和胰岛素含量偏高。匹兹堡大学某研究团队在期刊Molecular And Cellular Endocrinology发表了一篇名为《Chronic circadian shift leads to adipose tissue inflammation and fibrosis》的文章,该团队之前的研究发现,重要的时钟转录激活因子Bmal1是脂肪形成的关键调控因子,在此基础上,该研究团队使用环境光照诱导时钟节律的中断,再用Echo显微镜去观察小鼠长达6个月的慢性时钟失调导致的相关细胞形态变化以及局部组织炎症和纤维化情况,结合转录组学分析,该研究初步确定了慢性时钟节律失调会引起显著的脂肪组织功能障碍,这可能也是昼夜节律失调导致了胰岛素抗性高的基础。文章相关结果:在本研究中,我们测试了环境光照移位诱导的时钟干扰是否会影响脂肪组织生长。我们设计了一个轮班计划,如图1A所示。这种轮班方案旨在引起类似于环境昼夜失调的中枢和外周昼夜时钟不同步,我们之前证明了类似的5周方案导致β细胞时钟失调。两组分别在正常周期的第二天上午8点(ZT3)和下午5点(ZT12)采集样本,如图所示(图1A)。这些时间点近似对应于ZT3和ZT12时附睾白色脂肪组织(eWAT)和腹股沟皮下白色脂肪组织(iWAT)中Bmal1的蛋白水平峰值。经过6个月治疗后,对照组和轮班组小鼠的体重都增加了50%以上,而且两组之间的体重没有差异(图1B)。令人惊讶的是,对内脏脂肪库的组织学分析显示,轮班组小鼠的脂肪细胞明显增大(图1C)。对eWAT脂肪细胞大小分布的定量分析显示,其大小明显向较大的脂肪细胞转移(图1D)。在iWAT中(图1E),同样可以观察到脂肪细胞肥大,其大小分布明显向较大的脂肪细胞倾斜(图1F)。此外,我们发现典型的冠状结构由凋亡脂肪细胞周围的巨噬细胞组成,在时间移位组的小鼠中都很丰富,而在对照组中很少出现。▲图1组织学分析显示,在轮班队列的小鼠脂肪库中存在大量的冠状结构,表明存在巨噬细胞浸润的现象,这是脂肪组织炎症的特征。与这一观察结果相一致的是,RNA-seq分析也发现参与炎症反应的基因表达显著增强。在内脏脂肪中,相关代谢通路在ZT3时表现出正常的低表达,在ZT12时表达升高,表现出日表达谱(图5A)。慢性转移导致该通路在ZT3上的表达显著升高,这与ZT12非常相似,但失去了日表达模式。该通路在轮班组中也被诱导表达(图5B)。对光照时间移位处理的小鼠进行详细的组织学检查,显示其巨噬细胞积累具有丰富的冠状结构特征,而在对照组中检测到最小的冠状结构(图5C)。利用巨噬细胞特异性表面标记物F4/80抗体进行免疫荧光染色,并用Echo显微镜鉴定了凋亡脂肪细胞周围的形成冠状结构的巨噬细胞。▲图5现代生活方式中睡眠及活动周期与内源性时钟周期的频繁失调会导致昼夜节律不同步,这就可能导致代谢性疾病的发生,总而言之,昼夜作息节律失调容易导致肥胖和糖尿病。在文献中我们可以清晰看到脂肪组织炎症情况,同时可以很清楚地观察到巨噬细胞的蛋白表达,那么不得不夸一夸咱们用的显微镜,很显然Echo显微镜在此发挥的作用功不可没。Echo Revolve Gen2正倒置一体电动荧光显微镜独特的正倒置一体设计,既可观看切片,又可观察培养瓶中的活细胞,同时配置了高能LED光立方荧光系统,极大程度降低荧光淬灭的速度,双相机设置既兼顾灵敏度又兼顾色彩还原,DHR功能抑制噪声减少模糊,Z-Stacking可轻松逐层拍摄提高景深,配置iPad系统,实现极简操控,易学易用,心动的话赶紧行动起来!原文:https://doi.org/10.1016/j.mce.2020.111110Revolve Gen 2正倒置一体电动荧光显微镜新一代Revolve Gen 2正倒置一体电动荧光显微镜,拥有流行的触屏操控方式,配备智能荧光成像系统,将Z-Stacking全景深成像和DHR数字处理功能有机联合,提升分辨率告别照片模糊,为您打造全新的成像体验。
应用实例
2022.09.21
【网络研讨会】数字PCR技术在废水样本中疾病监测的应用
废水中病毒载量监测自COVID-19新冠疫情以来,凭其低成本和无偏差优势,被世界各地广泛用于监测疫情和预测疾病动态,但是建立统一的废水分析方法极具挑战。样品处理方法和基于PCR的检测方法,在持续地改进和优化。本次在线研讨会邀请了来自EAWAG瑞士联邦供水、废水处理与水体保护研究所Timothy R. Julian博士、法国Stilla数字PCR应用专家Martin Becker博士,和Promega公司高级市场经理Brigitta Saul博士,将讨论直接捕获、浓缩和纯化方法结合数字PCR技术持续监测废水中的新冠病毒SARS-CoV-2载量,同时监测新冠病毒变异毒株。同时还将分享:改进方法生成的新数据与历史数据如何进行趋势分析?研讨会主题:数字PCR技术在废水样本中疾病监测的应用研讨会时间:2022年9月22日 11:00PM(北京时间)主讲人:Timothy R. Julian博士,EAWAG(瑞士联邦供水、废水处理与研究所)水体保护环境微生物专家Martin Becker博士,Stilla Technologies应用专家Brigitta Saul博士,Promega高级市场经理主讲内容• 数字PCR技术监测新冠病毒SARS-CoV-2载量和重点关注的变异毒株• 废水中病毒监测方法学变化对病毒载量的影响• 病毒载量如何转化为流行病学数据的解读注册报名:扫描二维码注册报名欢迎注册报名,注册成功后将收到参会链接,请注意查收,准时加入。【关于Stilla Technologies】法国Stilla Technologies是总部位于巴黎的欧洲生物创新技术公司,具有跨学科专业知识的全球团队,利用先进的微流体化学,分子生物学和计算机科学等技术,拥有80多项全球专利,致力于提供突破性且灵活的naica®系统来加速下一代基因检测的开发。为全球的研究人员和临床医生提供高精度的遗传分析解决方案来改善健康状况。【关于深蓝云】北京深蓝云生物科技有限公司作为法国Stilla Technologies公司在中国的数字PCR技术示范与服务中心,在北京和苏州建有标准PCR实验室,致力于为用户提供新型生命科学研究仪器和分析产品以及优化的整体应用解决方案。深蓝云生物配备着专业的技术支持和应用支持,依托生命科学产品和解决方案,专注为用户提供分析产品和完善的售前咨询和售后服务。naica®六通道微滴芯片数字PCR系统法国Stilla Technologies公司naica®六通道微滴芯片数字PCR系统,源于Crystal微滴芯片式数字PCR技术,自动化微滴生成和扩增,每个样本孔可实现6荧光通道的检测,智能化识别微滴并进行质控,3小时内即可获得至少6个靶标基因的绝对拷贝数浓度。
新品
2022.09.19
新品上市 :RUBY芯片
加样即运行一体化微流控芯片Ruby上市为实现自动化上样提供可能,配合naica®微滴芯片数字PCR系统,为您提供首个“加样即运行”的数字PCR工作流程,无论是手动操作,还是自动化移液工作站,均实现一步移液步骤即完成样本加载。极简工作流程,不需要其他耗材,有效减少从样本到结果过程的可变因素。• 16个样本/芯片,48个样本/运行• 一体化微流控芯片• 结果高度稳定,提高实验效率• 兼容手动和自动化移液设备• 兼容naica®微滴芯片数字PCR系统 了解更多信息我们的应用专家整理了技术说明,详细描述Ruby芯片的产品特性。如您感兴趣请发送电子邮件至info@cycloudbio.com与我们取得联系。
新品
2022.09.15
Echo Revolve显微镜在结核病的治疗途径与药物学研究中的应用
前言结核病(TB)仍然是单一传染源的首要死亡原因。2017年,估计出现了超过55万例耐多药/利福平结核病(MDR/RR-TB)患者,强调需要新的治疗策略。利奈唑胺(LZD)是一种治疗耐药革兰氏阳性感染的有效抗生素,也是结核病的有效治疗方法。然而,长期使用LZD可导致LZD相关的宿主毒性,最常见的是骨髓抑制。LZD毒性可能由IL-1介导,IL-1是结核分枝杆菌感染期间早期免疫的重要炎症途径。然而,IL-1可导致结核病进展后期的病理学和疾病严重性。由于IL-1可能有助于LZD毒性,并确实影响结核病病理学,因此本实验将这一途径作为潜在的宿主导向治疗(HDT)的靶点。本实验假设LZD的疗效可以通过调节IL-1途径来增强,以减少骨髓毒性和结核相关炎症。本文使用两种结核病动物模型进行研究,一种是结核病易感小鼠模型和临床相关猕猴。在本研究中应用Echo Revolve正倒置一体荧光显微镜首先对小鼠肺组织的病变情况进行观察,以及肺组织病变面积的测量及可视化呈现;其次观察猕猴胸骨骨髓组织切片,比较细胞数量和差异性变化。荧光显微镜观察鼠抗CD3e、鼠抗Ly-6G抗体在肺组织中的特异性表达情况。通过苏木精-伊红(HE)染色对WT或Nos2-/-小鼠的单个肺叶进行组织病理学分析。αIL-1R1治疗降低了肺脏的平均细菌负荷(图1)。αIL-1R1治疗没有加重任何一种小鼠株的疾病。▲ 图1 苏木精-伊红(HE)染色对WT或Nos2-/-小鼠单个肺叶的组织病理学分析结果作者使用鼠抗CD3e、鼠抗Ly-6G抗体孵育,在Echo Revolve 4荧光显微镜DAPI FITC和RFP3个通道下分别观察继发性荧光和自发性荧光,将3个通道的图像叠加,可观察到图像中的紫色为抗体在肺组织中特异性表达,见图2。由于对Mtb感染和LZD治疗产生的IL-1β在很大程度上取决于NLRP3炎症体,本文还研究了一种方案,其中在感染后56天和77天之间施用LZD和小分子NLRP3抑制剂(MCC950)。如αIL-1R1所观察到的,与LZD单独相比,添加MCC950减少了肺中的PMN数量,并且没有显著改变细菌杀灭(图2)。▲ 图2 不同治疗组的肺部组织免疫荧光图像用DAPI染色的细胞核(蓝色)、用鼠抗CD3ε染色的T细胞(绿色)和用鼠抗Ly-6G染色的中性粒细胞(红色)为了证实我们的观察结果,病理学家评估了猕猴骨髓组织切片。结果发现不同治疗组,如TB组、LZD组和LZD+IL-1Rn组在细胞数量(髓系:红系比率)或异常方面没有明显差异(图3)。▲图3 猕猴尸检时获得的胸骨骨髓的代表性HE图像图片中的左侧图像为20X拍摄结果,右侧图像为40X拍摄结果研究亮点:▶ 本文使用Echo Revolve正倒置一体荧光显微镜眀场清晰观察到小鼠肺组织的病灶情况,还清晰观察到猕猴胸骨骨髓细胞分布状况以及进行不同治疗组间细胞数量比较;眀场可一键快速切换到荧光成像,荧光观察对不同治疗组的肺部组织中的抗体特异性表达进行清晰观察。▶ 阐明了小鼠和猕猴之间骨髓抑制的差异突出了在人类实验之前评估多个模型中HDT的重要性;在两种模型中,IL-1Rn给药对宿主的影响,以及对Mtb和LZD功效的反应,可以通过显微镜观察两种模型细胞中的病灶变化,细胞数量以及差异性变化来鉴定IL-1与LZD联合抑制是否对结核病治疗有效。本文研究数据为IL-1Rn作为结核病的潜在治疗提供了临床前数据。原文:doi: 10.3389/fimmu.2020.00891Revolve Gen 2正倒置一体电动荧光显微镜新一代Revolve Gen 2正倒置一体电动荧光显微镜,拥有流行的触屏操控方式,配备智能荧光成像系统,将Z-Stacking全景深成像和DHR数字处理功能有机联合,提升分辨率告别照片模糊,为您打造全新的成像体验。
应用实例
2022.09.14
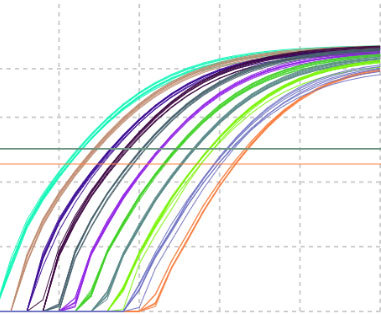
Taq酶你选对了吗?
导读目前国内市场上销售的热启动Taq酶的品牌很多,但真正高质量的热启动Taq酶并不多,面对这么多的热启动Taq酶产品,我们应如何选择?首先,选择扩增效率高的热启动Taq酶耐受性好的Taq酶反应体系经优化后,扩增效率在95%以上,即使是在目标基因含量较低时也能够获得满意的扩增,在模板量较高时,也不易中毒,指数扩增期较长。耐受性能较差的Taq酶,即使反应体系经多次优化,扩增效率仍达不到90%,扩增曲线“S”型不明显,斜率较小,曲线低平。模板量较低时扩增不出来,较高时扩增效果也不理想。所以PCR扩增效率与Taq酶的性能密切相关,选择高扩增效率的DNA聚合酶对PCR、qPCR能否成功至关重要。其次,选择酶动力强的热启动Taq酶Taq酶的酶动力与扩增效率有关。一般热启动Taq酶的酶动力越强,PCR扩增的指数增长期越长,‘S型’曲线更加典型,荧光信号值更高,而且更适合做多重PCR检测。酶动力弱的品牌DNA聚合酶一般只能支持2重反应,在做3重反应时,扩增曲线低矮,荧光信号值较低,无典型扩增曲线,结果很难判定。最后,选择灵敏度高的热启动Taq酶一般说,DNA聚合酶的扩增效率高,灵敏度就高,但也有不一致的情况。如果要扩增的样本目标基因丰度较低,建议对Taq酶的扩增灵敏度进行检测。Taq酶的扩增灵敏度进行检测,常见的检测方法是将目标基因质粒片段进行10倍或5倍梯度稀释,在较低的几个稀释度进行PCR检测,选择检测灵敏度较高的热启动Taq酶。由此可见,研究者需根据自己的实验要求和经费情况进行选择,最好做一个梯度稀释扩增实验,以检测热启动Taq酶的扩增效率和灵敏度。CieloTM实时荧光定量PCR系统Harness of the power of qPCR▌12列温度梯度设置:多温度梯度设置,快速优化条件,确定合适退火温度。▌良好的S型扩增曲线。▌熔解曲线单峰,无杂峰。除此之外, Cielo™实时荧光定量PCR系统融合了高品质Peltier温度模块和基于光纤传输的光学检测系统,可为您的科学研究就提供高精准、高灵敏可靠结果。
应用实例
2022.09.05
naica®微滴芯片数字PCR系统精准量化胰岛素编码基因DNA甲基化水平
导读在过去的几十年中,糖尿病的发病率在全球范围内显著增长。除了不健康的生活方式外,环境污染物被认为是糖尿病发生的危险因素。多环芳烃 (PAH)是一类含有2-7个芳环的有机化合物,由自然和人类活动产生并广泛存在的污染物。流行病学研究表明,PAHs水平与成人和儿童的肥胖和二型糖尿病相关。厦门大学生命科学学院细胞应激生物学国家重点实验室的研究人员在Ecotoxicology and Environmental Safety上发表了题为《Prenatal exposure to a mixture of PAHs causes the dysfunction of islet cells in adult male mice: Association with type 1 diabetes mellitus》的文章。文中应用naica®微滴芯片数字PCR系统对胰岛素编码基因DNA甲基化水平进行量化,揭示了产前暴露于多环芳烃混合物对成年雄性小鼠胰岛细胞功能的不良影响。应用亮点:▶ 使用naica®微滴芯片数字PCR系统对胰岛素编码基因启动子甲基化水平进行量化。▶ 在产前暴露于500µg/kg PAHs的小鼠中,胰岛素编码基因启动子的甲基化水平显著升高。▶ 产前暴露于PAHs可能促进I型糖尿病的发病。作者使用8种PAHs的混合物进行了实验,以研究产前PAHs对成年期胰岛细胞功能和质量的影响,同时试图阐明 I型糖尿病发病的环境原因。他们分离了成年雄性小鼠的胰岛,对胰岛素编码基因的启动子DNA甲基化水平进行分析。研究成果:▲图1. 产前暴露于多环芳烃对成年雄性小鼠胰岛素编码基因甲基化水平的影响。(A) 数字PCR结果代表性一维图。(B)胰岛素编码基因启动子甲基化水平。(每个处理三只母鼠, 每只母鼠取一个雄性后代) 。在本研究中,子宫内暴露于500µg/kg PAHs的小鼠胰岛中胰岛素编码基因启动子中的DNA甲基化水平显著增加,同时胰岛素编码基因转录显著下调。▲图2. 不同PAHs浓度对胰岛素编码基因转录水平的影响原文链接如下:https://www.sciencedirect.com/science/article/pii/S0147651322005358期刊介绍:Ecotoxicology and Environmental Safety 1977创刊,隶属于爱思唯尔出版集团。是一份多学科交叉期刊,主要研究环境污染对包括人类健康在内的生物体的暴露和影响。最新影响因子为7.129。naica®六通道数字PCR系统法国Stilla Technologies公司naica®六通道数字PCR系统,源于Crystal微滴芯片式数字PCR技术,自动化微滴生成和扩增,每个样本孔可实现6荧光通道的检测,智能化识别微滴并进行质控,3小时内即可获得至少6个靶标基因的绝对拷贝数浓度。
应用实例
2022.08.18
qPCR体系优化和常见问题分析
前言聚合酶链式反应(PCR)是用于扩增特定DNA片段的分子生物学实验技术。实时荧光定量PCR(以下简称qPCR)作为第二代PCR技术,自1996年推出以来,已经广泛应用于基因表达分析、病原微生物检测、动植物育种等许多研究领域,为了获得最理想的检测结果,qPCR从样本采集、核酸提取、cDNA合成到上机检测的流程有许多可以优化的参数。qPCR实验的工作流程首先需要确定研究的目的,根据实验设计规划好实验分组、重复次数等细节。接下来分为样本准备和引物探针验证两个重要的步骤。样本准备主要是核酸提取逆转录等步骤,引物探针需要去测试特异性和效率。接下来需要使用qPCR仪来对样品中的目的核酸进行扩增qPCR结束后根据实验目的对目的核酸进行相对或者绝对定量。接下来讲的qPCR体系优化会围绕着这个流程展开。1.样本的采集与处理首先,提前做好功课,了解样本的不同分型,或者了解详细的细胞分群。如果条件允许尽可能覆盖所有的组织类型或者细胞类型。其次,尽可能增加样本数量,也就是生物学重复,从而更客观地反映生物变异程度。另外,qPCR实验也需要有技术重复来降低误差。采样是需要严格规划的过程,比如材料的时效性、珍贵程度等,都要纳入考量范围。样品要尽量新鲜,取样尽可能快速。戴手套操作,防止污染。如果不马上提取核酸,需要-80°C保存,并尽快处理。2.核酸的提取和检测模板的质量直接影响到检测性能。核酸提取需要有效地将RNA或DNA从其他混合物中分离。RNA样本中的污染物——基因组DNA、DNA结合蛋白、酚类化合物或在提取RNA过程中引入的外源杂质(如手套中的粉末)——都已被证明会抑制下游实验,如逆转录和PCR扩增。核酸提取需要使用无菌无酶的试剂耗材,避免RNase或DNase污染,并对内源RNAse或DNAse进行有效抑制;多糖多酚样品要考虑多糖多酚杂质的有效去除。低温保存防止RNA或DNA降解。降解或不纯的RNA会限制逆转录反应的效率,降低产量。部分降解的RNA可能不能给出准确的基因表达结果。对于基因的定量,必须使用高质量的RNA,这意味着需要非常仔细地检查RNA的浓度和质量。可采用高分辨率琼脂糖凝胶检测核酸质量和分光光度法(A260/A280=1.8和A260/A230=2.0)检测核酸纯度和浓度。3.cDNA合成RNA 质量对 cDNA 合成结果会产生重要影响。并且RNA 很脆弱,容易降解。为了保证 RNA 的完整性,我们需要非常注意,比如在冰上操作,用 RNase-free 的枪头和离心管,减少操作时间等。在反应体系中加入 RNase 抑制剂也能有效防止 RNA 降解。如何评价样品中的杂质对逆转录的影响呢?可以梯度稀释后绘制标准曲线,如果低浓度的样品点数值偏大比较明显,基本可以判定杂质影响显著。不同厂家的反转录试剂会有差异,对RNA中的杂质耐受程度也不同。逆转录酶在整个反转录体系中具有关键性影响。除了活性以外,逆转录酶的热稳定性同样很重要,在较高温度下进行逆转录,能够减少 RNA 的二级结构,增加逆转录的效率。除了掌握 RNA 的完整性之外,反转录之前还需要对 RNA 浓度进行测定。一般反转录试剂盒会对上样量有要求,建议 total RNA 上样量小于 5 μg。超过这个范围,会使反转录产物产生偏好性 (表达丰度高的基因优先被反转录) 而造成定量结果不准确。逆转录出来的cDNA可以直接放在4°C保存,若长期不用,可分装,然后-20°C保存。4.qPCR方法的建立① 定量方法绝对定量:检测起始模板数的精确拷贝数,需要标准品构建标准曲线。标准品可以是纯化的基因组DNA、质粒DNA或者体外转录RNA(cDNA),其作用是生成标准曲线,建立Ct值与浓度之间的线性关系。标准品与待测样品的PCR效率一致,且接近100%,与样品的性质尽可能接近,与样品相同的扩增条件(PCR体系、耗材、同一次扩增),大于或等于5个梯度稀释的标准品。相对定量:在一个样本中,目的基因相对于内参基因的量的变化。内参基因选择建议筛选不少于三个内参基因来归一化RT-qPCR数据。目的是消除外部样品偏差,例如总RNA含量,RNA稳定性,酶效率或样品装载量的变化。对候选的内参基因进行qPCR 实验,得出Ct平均值以及 Ct值的标准偏差,选择SD最小的基因作为实验内参。可通过geNorm 、 BestKeeper 、 NormFinder、RefGenes 等工具来评估您的内参基因。② 荧光标记方法染料法:利用能与DNA双链结合的染料来实现,如SYBR Green I。该染料在游离状态下呈现微弱的荧光,一旦与双链DNA的双螺旋小沟结合,其绿色荧光增强约1000倍。因此其总的荧光强度与双链DNA含量成正比,利用这一关系可以反映生成的PCR产物的量。TaqMan荧光探针:是一种寡核苷酸探针,荧光基团连接在探针的5'末端,而淬灭剂则在3'末端。PCR扩增时在加入一对引物的同时加入一个特异性的荧光探针,探针完整时,报告基团发射的荧光信号被淬灭基团吸收; PCR扩增时, Tag酶的5'-3'外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号,每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步。常用的荧光基团是FAM,TET,VIC,HEX。引物探针设计可以参考Gene π网站:https://www.gene-pi.com/item/primers-and-probes-2/③ 引物扩增效率验证标准曲线是评估PCR扩增效率最可靠和稳定的一种方法,该方法涉及到制作一系列的样品来控制目标模板的相对数量。最常用的是10倍梯度稀释样品,采用标准qPCR程序进行扩增获得Cq值,最后根据各样品浓度及相应的Cq值绘制标准曲线,得到线性方程Cq= -klgX0+b,扩增效率E=10(-1/k)-1。利用qPCR进行定量分析时,要求扩增效率范围在90%-110%(3.6>k>3.1)。④ 反应体系优化▶ 根据仪器类型,选择合适的耗材和qPCR试剂。▶ 每对引物先进行预实验,确定特异性以及最适浓度。▶ 配置不同的PCR反应体系,选择每个组分合适的浓度。▶ 设置温度梯度测试引物最合适的退火温度。▶ 实验设置NTC、NRT、 NEG和POS等对照组,来监控实验体系或污染。实时荧光定量PCR常见问题分析1.可疑的扩增曲线真正的扩增曲线,有特征的形状:首先背景信号,然后是三个增长阶段(指数增长期、线性增长期和平台期)。如果不是同时具有特征性的三个增长阶段,没有典型的指数增长期,那就不存在扩增。平台期很低也是常见的异常扩增曲线。可能是模板的浓度太低。通常如果模板的起始浓度太低, 反应体系中会形成大量的引物二聚体。大量引物二聚体的形成使得引物很快消耗完,从而造成扩增曲线的平台期很低。这种情况可通过调整引物和模板的比例。2.异常的荧光信号NTC出现荧光信号---引物二聚体形成或气溶胶污染,查看熔解曲线是否为单一峰。3.扩增效率过高或过低过低的扩增效率(▶ 移液器校准不良或移液技术差。▶ 不正确的稀释导致标准曲线出现错误。▶ 引物设计不好或扩增子具有二级结构。▶ 标准曲线动态范围太小。▶ Taq酶无活性或活性降低。▶ 样品抑制。过高的扩增效率(> 110%)可能存在的原因:▶ 移液器校准不良或移液技术差。▶ 不正确的稀释导致标准曲线出现错误。▶ 引物二聚体或非特异性扩增。▶ 标准曲线动态范围太小。▶ 基因组DNA污染。4.重复性差为精确定量,对每个样品都要做重复实验,复孔之间的Ct值不应超过0.5,标准偏差不大于0.2,这样,实验结果就有很好的精确度。造成重复性差的原因:▶ 加样误差(操作或者加样器导致)。▶ 没有将试剂和样品充分混匀。▶ 低拷贝的目的片段→泊松分布。▶ 基线阈值设定不合理。Cielo™实时荧光定量PCR系统Harness of the power of qPCR☑ 数据可靠性:连续1000次实验后,结果高度一致。☑ 应用灵活性:提供多种qPCR应用分析。☑ 流程智能化:中英文用户界面,触控操作,可多机联用。☑ 在线便捷性:主机可独立运行qPCR程序,数据可USB、Wi-Fi等网络传输。
应用实例
2022.08.15
什么是活细胞成像,怎样才能得到一张好的活细胞成像图
• 什么是活细胞成像? 活细胞成像(live cell imaging)统称为捕捉活的、活动状态的细胞图像的技术,这些细胞图像可以是单个静态图像,也可以是延时系列图像。相应地,活细胞成像的应用可以分为两大类:❶ 细胞在自然状态下的图像记录。❷ 实时观察和记录细胞、组织或整个生物体的动态过程。• 观察分析活细胞时面临的挑战 ▷ 在相对较短的时间内采集大量信息。▷ 要保持细胞保存在可调节培养环境气体浓度和温度(在很多情况下)的培养室中。▷ 激发光源会损害活细胞。▷ 细胞焦面漂移,无法聚焦。▷ 需要使用配备有软件或硬件控制自动对焦的成像仪器来避免这种情况。Revolution全自动显微镜成像系统Revolution全自动显微镜成像系统部件高度集成内置,节省空间,避免繁琐调试及维护;触屏式操控观察工作站,界面直观简洁,易于学习,方便使用。Revolution全自动显微镜成像系统的光源采用高能LED光源,自动荧光切换把光毒降到最低。▌智能化全自动多功能系统:▶ TimeLapse延时摄影:可以根据设定在特定时间内完成特定间隔时间和特定的拍照张数。▶ 独有的Hyperscan快速成像:30帧高速成像,可以在几秒钟内完成上百张照片的采集。▶ Multi-well Point孔板导航成像:不限定孔位大小,只需输入参数就可以自动完成多孔或单孔采集。▶ Focus Map自定义多点聚焦:可以自动完成不同层面的自动聚焦。▶ Z-Stacking多层扫描大景深成像:完成多层面大景深成像。▶ DHR智能实时数字化降噪:实时完成反卷积计算,得到清晰图像。▌ECHO INCUBATOR为活细胞观察提供一个稳定而灵活的培养环境ECHO INCUBATOR采用紧凑的一体式设计,方便用户快速安装和拆卸。箱体结构透明和大型前置开门设计,可为用户提供清晰的观察视野并方便操作样本。采用无风扇对流加热和循环热空气方案,在消除振动的同时并可防止外部灰尘进入您的样品和仪器光学元件。提供稳定的细胞生长环境,确保适合的细胞培养条件,使细胞处于最佳生长状态。
企业动态
2022.08.09
普通PCR引物 VS qPCR 引物
导读说起引物,大家都再熟悉不过了。引物,在核苷酸聚合作用的起始时,可以刺激合成另一种大分子,并与反应物以共价键形式连接的序列。在核酸化学中,引物是一段短的单链RNA或DNA片段,可结合在核酸链上与之互补的区域,其功能是作为核苷酸聚合作用的起始点,核酸聚合酶可由其3′端开始合成新的核酸链。无论是做普通PCR还是荧光定量PCR,设计合适的引物是非常关键的一步。普通PCR和荧光定量PCR的检测方式具有较大的差别。荧光定量PCR实时监测与DNA结合的荧光染料激发的荧光,普通PCR通过检测插入DNA中核酸染料的量来测定PCR最终产物量。荧光定量PCR可以通过扩增曲线进行精确定量,普通PCR则是通过电泳的方式进行定性分析。那么在普通PCR和荧光定量PCR的引物设计方面,有什么相同点和不同点呢?今天小编就给大家汇总一下。相同处:▶ 序列的查找是一致的。▶ 序列选取应在基因的保守区段。▶ 选取合适的扩增片段大小。▶ 避免引物自身或与引物之间形成4个或4个以上连续配对。▶ 避免引物自身形成环状发卡结构。▶ Tm值在55-65℃,GC含量在40%-60%。▶ 引物之间的Tm相差避免超过2℃。▶ 引物的3’端避免出现3个或3个以上连续相同的碱基。不同处:荧光定量PCR 引物▶ PCR产物长度:要求在300bp以内,一般首选80-150bp之间。▶ 引物特异性:对引物要求高,尽量减少引物二聚体的产生,熔解曲线要求单一产物。▶ 扩增效率:荧光定量 PCR的目的是进行定量或者相对定量,扩增效率应在90%—110%之间。▶ 多对目的基因同时扩增,文献上查来的引物条件会不同,需要设计尽可能条件一致的引物。▶ 目的基因含量比较低时,需要设计灵敏度比较高的引物。普通PCR 引物▶ 普通PCR的扩增长度,根据实验的要求不同,一般是从150bp到几千bp不等。▶ 相对于荧光定量PCR,普通PCR对二级结构要求较低。▶ 对扩增效率要求不高。▶ 可以选用梯度PCR仪,选择不同退火温度,对引物退火温度要求不需要一致。Cielo™实时荧光定量PCR系统Harness of the power of qPCR☑ 数据可靠性:连续1000次实验后,结果高度一致。☑ 应用灵活性:提供多种qPCR应用分析。☑ 流程智能化:中英文用户界面,触控操作,可多机联用。☑ 在线便捷性:主机可独立运行qPCR程序,数据可USB、Wi-Fi等网络传输。
应用实例
2022.08.04
单个大肠杆菌检测新思路| naica®全自动微滴芯片数字PCR系统提供最强支撑
导读尽管各国卫生系统发展迅速,但由病原菌引起的传染病仍然是人类健康的主要威胁之一。据报道,全世界每年有220多万人死于水传播大肠杆菌病原体。尽管大多数大肠菌群是无害的,但某些大肠杆菌的存在可能会导致甚至威胁到人类健康,例如,大肠杆菌O157:H7和其他产志贺毒素的大肠杆菌菌株(非O157 STEC)是食源性疾病的常见因素,可能对健康造成严重后果,尤其是对幼儿。因此,检测大肠杆菌对于生物医学应用以及食品、水和空气质量监测非常重要。大连理工大学环境科学与技术学院,工业生态学与环境工程教育部重点实验室的科学家,开发出基于naica®全自动微滴芯片数字PCR系统的单细菌检测方法,该方法可以在1.5小时内以单细胞灵敏度选择性检测临床尿液样本中的大肠杆菌 。该方法发表在《Analytical Methods》,题为“Single bacteria detection by droplet DNAzyme-coupled rolling circle amplification”。应用亮点:▶ dDRCA系统,能够快速、选择性地检测具有单细胞敏感性的大肠杆菌 ,dDRCA系统的检测灵敏度比之前报道的PAD高1000倍。▶ 证明了dDRCA系统在尿路感染诊断中的潜在临床适用性,dDRCA能够在不到1.5小时内,从20份临床尿液样本中成功识别出5名UTI患者,而传统的基于培养的方法需要数小时。文中采用naica️®全自动微滴芯片数字PCR系统液滴微流控技术,快速精准的检测到复杂样本和高背景样本中的致病大肠杆菌。通常扩增需要约4小时进行定量大肠杆菌检测。在这项研究中,我们描述了DNA酶偶联滚圈扩增(RCA),这是一种高效的等温酶DNA复制过程,可在naica️®全自动微滴芯片数字PCR系统上进行,以建立液滴DNA酶偶联RCA(表示为dDRCA)系统。我们进一步证明,该系统能够在1.5小时内以单细胞敏感性选择性检测临床尿液样本中的大肠杆菌。▲图2(a)通过琼脂糖凝胶电泳分析RCA产物(RP)。(b) 反应混合物在指示反应条件下的荧光响应:+RFD-EC1/+ RDS/+ E. coli (blue line); RFD-EC1/+ RDS/+ E. coli (green line); RFD-EC1/+ RDS/-E. coli (red line); + RFD-EC1/+ RDS/-E. coli (pink line)。(c) Naica Prism3阅读器图像(左)、CLSM图像(中)和荧光显微镜图像(右)为微晶芯片液滴的大小。比例尺:1.4 mm(左)和100 mm(中、右)。指示反应条件下dDRCA系统的荧光图像和荧光滴数:(d)+RFD-EC1/+ RDS/+ E. coli;(e)-RFD-EC1/+ RDS/+ E. coli; (f) -RFD-EC1/+ RDS/ E. coli; (g) + RFD-EC1/+ RDS/ E. coli.使用Naica Prism3阅读器对生成的荧光液滴进行成像和分析。dDRCA系统能够在75分钟内选择性计数具有单细胞敏感性的大肠杆菌,包括20分钟的细胞裂解时间、12分钟的液滴生成时间和43分钟的液滴反应时间。通过比较其他三种常见细菌,包括枯草芽孢杆菌(B.subtilis)、酸性乳片球菌(P.acidilactici)和唐菖蒲伯克霍尔德菌(B.gladioli)存在时的信号反应,也检查了dDRCA检测大肠杆菌的选择性。当用缓冲液或尿液中的这些意外靶点测试每个dDRCA系统时,未观察到明显的荧光液滴(图4c和S4†)。▲图4(a)不同大肠杆菌浓度(每毫升细胞数)下dDRCA反应的荧光图像。比例尺:1.4 mm。(b) 不同浓度下计数的液滴数与大肠杆菌之间的关系。提供了计数的液滴数量,插图显示了1–104大肠杆菌范围内的线性反应。误差条代表三个独立实验的标准偏差。(c) dDRCA的特异性。最终证明了dDRCA系统在尿路感染(UTI)诊断中的潜在临床适用性。分析了20份患者和健康献血者的临床尿样。整个操作程序包括:(1)细胞收集和裂解(25分钟);(2) 液滴生成(12分钟);(3) 液滴反应(43分钟)。如图5c所示,五个尿样,即ID 6、8、9、12和14,比其他尿样产生大量荧光液滴(>3000)。使用传统的大肠杆菌培养方法进一步确认了这些有无大肠杆菌感染的样本(图5d)。因此,对UTI的诊断有很大的希望。▲图5(a)检测尿液样本中的大肠杆菌。(b) 显示尿液样本中计数数与大肠杆菌细胞在1-104范围内的线性相关性的曲线图。(c) 20份临床尿液样本中阳性液滴的数量。(d) CLED(胱氨酸、乳糖电解质缺乏)琼脂细菌尿液培养板。黄色菌落代表大肠杆菌在37℃的CLED琼脂中培养22小时后的生长。该系统能够在不到1.5小时的分析时间内,从20份临床尿液样本中成功识别出5名UTI患者,而传统的基于培养的方法需要数小时。原文:https://doi.org/10.1039/D2AY00656Anaica®六通道数字PCR系统法国Stilla Technologies公司naica®六通道数字PCR系统,源于Crystal微滴芯片式数字PCR技术,自动化微滴生成和扩增,每个样本孔可实现6荧光通道的检测,智能化识别微滴并进行质控,3小时内即可获得至少6个靶标基因的绝对拷贝数浓度。
应用实例
2022.08.02
荧光定量PCR实验,荧光标记怎么选?
荧光定量PCR技术是将常规的PCR和荧光检测技术相结合,利用荧光信号积累实时监测整个PCR进程,以此实现对初始模板的定量分析。荧光定量PCR技术作为当今生物学研究的重要手段之一,在基础科学、生物技术、医学研究、法医学、诊断学等多方面具有广泛的应用前景。说到荧光定量PCR技术,我们经常会问类似于“你的实验是染料法还是探针法”,“你用的探针是什么类型”等之类的问题,这其实是对荧光标记的选择。根据荧光标记的不同,可以将荧光定量PCR实验分为探针法和染料法两大类。染料法染料法利用能与DNA双链结合的染料来实现,如SYBR Green I。该染料在游离状态下呈现微弱的荧光,一旦与双链DNA的双螺旋小沟结合,其绿色荧光增强约1000倍。因此其总的荧光强度与双链DNA含量成正比,利用这一关系可以反映生成的PCR产物的量(图 1)。SYBR Green I的最大吸收约在497 nm,发射波长最大约在520 nm,与FAM荧光分子的光谱性质类似,因此在所有的荧光定量PCR设备中都是第一通道检测,即“FAM/SYBR Green I”通道。▲ 图1 染料法原理图理论上,所有能与双链DNA结合的染料都可以用于qPCR检测,如溴化乙锭EtBr,碘化丙啶PI,吖啶橙、Cy3等。而选择用于qPCR反应的染料通常会从信号强度、生物安全性、检测简便性和经济适用性这几个因素考虑。例如EtBr可能存在潜在的致癌性。综合考虑下来,SYBR Green I成了比较理想的选择。目前,使用Evagreen的实验者也越来越多,Evagreen作为一种新型的染料,其光谱性质与SYBR Green I类似,其优势在于:▶ 对PCR反应的抑制程度小。高浓度SYBR Green I会强烈抑制PCR反应,因此要控制其使用浓度,一定程度上降低了DNA检测的灵敏度。▶ 与DNA结合密度高。单位长度的DNA双链上Evagreen的密度更高,为饱和型染料,消除了SYBR Green I存在的“染料重分布”的缺陷,提高检测灵敏度的同时也可用于高分辨率溶解曲线HRM。▶ 化学性质更稳定,适合长期保存。TaqMan 探针TaqMan 探针基本可以满足60%以上的qPCR实验,如常规的基因表达和拷贝数变异CNV实验。TaqMan 荧光探针是一种寡核苷酸探针,荧光基团连接在探针的5'末端,而淬灭剂则在3'末端(图2)。PCR扩增时在加入一对引物的同时加入一个特异性的荧光探针,探针完整时,报告基团发射的荧光信号被淬灭基团吸收; PCR扩增时, Tag酶的5'-3'外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号,每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步。常用的荧光基团是FAM,TET,VIC,HEX。▲ 图2 Taqman探针MGB探针对于要分辨单碱基差异的SNP实验,采用对碱基错配容忍度更低的MGB探针,在淬灭基团后加入了DPI3基团(图3),从而提高了与靶标结合的亲和力;而且可以对靶点碱基进行化学修饰,如PeptideNucleic Acid和Locked Nucleic Acid。▲ 图3 MGB探针双杂交探针Taqman探针对探针的长度有比较严格的要求,双杂交探针则消除了这一“缺陷”。双杂交探针是由两条寡核苷酸单链组成,一条的3’端带有供体荧光分子,另一条的5’端带有受体荧光分子(图4)。游离状态下荧光供体会发出荧光,但当两条单链都匹配到模板链上时,就会发生荧光共振能量转移FRET,受体荧光分子就可以发出荧光,其荧光强度与生成的产物的量成正比。双杂交探针的优势有两点:摆脱了探针长度的限制,较长的探针可以提高与模板匹配的成功率;只有上下游两条探针都正确匹配后才能检测到受体荧光分子发出的荧光,特异性有所提高。▲ 图4 双杂交探针分子信标探针游离状态下,分子信标探针是一种茎环结构,其环状部分的15-30个碱基可以与靶标区域相结合匹配,下端的配对区域(左右一般各5-6个碱基)则由重复的GC组成,从而将5’的荧光分子和3’的淬灭基团紧紧聚在一起,荧光发生淬灭;当退火时,分子信标探针解开环并与模板靶标杂交,这样荧光分子和淬灭基团的物理距离就变大了,荧光淬灭的前提就打破了(图5)。除了MGB探针,分子信标探针也非常适合用于SNP检测。▲ 图5 分子信标探针除了上述几种类型的探针之外,还有尝试将引物和探针功能相结合的技术,如Amplifluor、LUX等,在设计难度上有所提高,但使用时更简便。各有其应用的侧重点和设计上的利弊,在此不多赘述。那么在荧光定量PCR实验中,我们应该如何进行选择呢?在这里,给大家总结了一些两种方法的区别,供大家参考。表1 染料法和探针法的区别
应用实例
2022.07.22
再现奇迹,科学家打印一颗会跳动的心!
人造器官再现奇迹科学家打印出会跳动的心近期,美国卡耐基梅隆大学(CMU)的研究人员找到了解决方案。他们开发了一种叫做Freeform Reversible Embedding of Suspended Hydrogels(FRESH)技术,以胶原蛋白为生物墨水,3D生物打印了人类心脏的功能性部件(血管、瓣膜和心室搏动),并实现了前所未有的分辨率和保真度。相关研究结果发表在《Science》杂志上。在最新的研究中,Feinberg实验室开发的FRESH 3D生物打印方法允许胶原蛋白在支持凝胶中逐层沉积,使胶原蛋白有机会在从支持凝胶中取出之前固化。使用FRESH技术,打印完成后,通过将凝胶从室温加热至体温,即可将支持凝胶融化。这样,研究人员就可以在不破坏胶原蛋白或细胞打印结构的前提下移除支持凝胶。▲卡内基梅隆大学研究人员开发出一种3D生物打印胶原蛋白技术,可以制造人体心脏的全功能成分。( 图片来源:CMU )FRESH这种方法对于3D生物打印领域来说是非常令人兴奋的,因为它允许胶原支架打印大尺寸的人体器官。而且它不限于胶原蛋白,纤维蛋白、藻酸盐、透明质酸等多种软性生物材料均可作为生物墨水。通过FRESH技术进行3D生物打印,为组织工程提供了一个强大且适应性强的平台。更重要的是,研究人员还做了开源设计,这样任何人都可以构建并获得低成本、高性能的3D生物打印机。Feinberg表示,近期会进行例如因心脏病或肝脏受损而丧失功能的心脏修补工作。展望未来,从伤口修复到器官生物工程,FRESH在再生医学的许多方面都会有所应用。目前仍然存在的挑战是打印大型组织需要数十亿的细胞,如何实现制造规模以及遵循监管程序,以便能在动物和人类中进行测试。尽管任重道远,但我们距离实现3D生物打印全尺寸人类心脏的梦想又近了一步。您的首选3D打印技术综合解决方案供应商CELLINK在这领域中所扮演的角色使用FRESH 3D生物打印实现更复杂的几何形状FRESH 已迅速成为许多组织工程师首选的生物打印平台。FRESH 能够以更高的分辨率使用任何软凝胶生物材料进行生物打印,而不受几何复杂性的限制。FRESH 可以集成到标准生物打印工作流程中,并在 BIO X™ 等挤压式生物打印机上实现。FRESH 彻底改变了生物打印,使研究人员能够应对复杂组织结构和功能的紧迫挑战。例如,FRESH 消除了特定墨水打印优化这种繁琐的任务,让研究人员可以更专注于生物打印真正的 3D 支架和组织。在 BIO X™ 上进行 FRESH 3D 生物打印非常简单,只需将准备好的 LifeSupport™ 盘放在打印平台上,然后将打印针放入盘的中心即可开始制造复杂的几何形状。探索一些最有潜力的潜在研究方向:第一:类组织的复杂打印第二:形成血管化组织第三:多材料生物打印通过将 BIO X™ 生物打印机与 FRESH 相结合,研究人员可以迈入以往梦寐以求的打印复杂性和功能新领域。
应用实例
2022.07.20
深蓝云直播课 | 定量PCR体系优化及常见问题分析
聚合酶链式反应(PCR)是用于扩增特定DNA片段的分子生物学实验技术。实时荧光定量PCR作为第二代PCR技术,自1996年推出以来,已经广泛应用于基因表达分析、病原微生物检测、动植物育种等许多研究领域,为了获得最理想的检测结果,实时荧光定量PCR从样本采集、核酸提取、cDNA合成到上机检测的流程有许多可以优化的参数。直播时间:2022年7月7日下午3点直播亮点:1、荧光定量PCR的基本原理2、荧光定量PCR的体系优化3、荧光定量PCR常见问题分析主讲人:许雪琴,毕业于中国科学院大学,具有多年分子和细胞生物学实验经验。目前负责深蓝云生物科技荧光定量PCR、微滴芯片式数字PCR、Echo正倒置一体显微镜的技术支持工作。 扫描下方海报内二维码报名
企业动态
2022.07.01
ECHO REVOLVE显微镜在环境镉、细菌和宿主相互作用研究中的应用
前言环境镉的膳食摄入量高,生物半衰期长,直接或通过肠道微生物群损害生理功能,是一种严重的健康风险。然而,环境镉对微生物和宿主系统的毒性机制尚不清楚。中科院营养代谢与食品安全重点实验室和上海交通大学医学院的科学家在《Hazardous Materials》杂志联合发表了一篇名为《Cadmium accelerates bacterial oleic acid production to promote fat accumulation in Caenorhabditis elegans》的文章,本研究建立了三个线虫和大肠杆菌培养系统,以研究微生物在镉诱导的脂质毒性中的重要作用,阐明了镉通过细菌代谢物在体内诱导脂质积累的机制,并揭示了环境镉、微生物和宿主之间的相互作用。本研究使用ECHO REVOLVE正倒置一体荧光显微镜(RVL-100-G,Discover ECHO,US)检测VS29蠕虫的GFP荧光。激发和检测波长分别为470–495 nm和510–550 nm。使用图像处理和分析软件对荧光图像进行分析,计算其平均荧光密度。图A ECHO REVOLVE正倒置一体荧光显微镜拍摄转基因VS29蠕虫中GFP:dgat-2表达的代表性图像(左图)和相对定量(右图)(n≥ 15) 在成年期的第1、3、5和7天。比例尺:210µm。研究表明,镉暴露导致细菌代谢物的组成存在显著差异。油酸被确定为表达差异最大的代谢物。镉暴露显著增加了油酸的含量,表明镉具有特定的诱导效应。通过在正常和代谢失活系统中直接添加到秀丽隐杆线虫,进一步证实了油酸对体脂积累的影响。因此,研究发现镉诱导的复合差异(尤其是油酸的增加)而不是数量变化介导了镉对宿主的影响。除了油酸的产生外,还发现低剂量镉暴露可提高细菌中油酸合成相关基因(fabA、fabB、fabD、fabG、fabH、fabI、fabZ和accA)的表达。并且添加油酸增加了秀丽隐杆线虫的脂质合成和代谢相关基因(fat-5、fat-7、acs-11和sbp-1)的表达,这与镉暴露线虫中的现象相似。值得注意的是,与镉暴露不同,油酸对秀丽隐杆线虫体内脂质积累的影响是直接且独立于培养系统的,这表明升高的细菌油酸是环境镉促进宿主体内脂质积累的效应器。综上所述,本研究提出了一个模型来说明环境镉、细菌和线虫之间的相互作用。本研究首先集中于环境中低剂量镉引起的细菌代谢产物改变及其对镉暴露与宿主毒性之间关系的影响。本研究还探究了维生素D3在镉诱导的脂肪积累中的作用。补充维生素D3可显著降低低剂量镉加速的秀丽隐杆线虫体内的脂肪含量,表明补充维生素D3有可能防止环境镉暴露引起的脂肪异常积累。研究亮点:▶ 本研究发现在有活细菌系统中的线虫,镉提高了细菌代谢产物油酸的产生,并提高了其合成基因的表达。从而进一步促进了线虫脂肪代谢相关基因的表达和脂肪沉积。▶ 本研究发现了维生素D3的潜在保护作用,可以显著防止镉或油酸诱导的脂肪沉积,可以降低环境镉的脂质毒性,这些发现为镉引起的健康风险和毒理学机制提供了深入的见解。原文:https://doi.org/10.1016/j.jhazmat.2021.126723Revolve Gen 2正倒置一体电动荧光显微镜新一代Revolve Gen 2正倒置一体电动荧光显微镜,拥有流行的触屏操控方式,配备智能荧光成像系统,将Z-Stacking全景深成像和DHR数字处理功能有机联合,提升分辨率告别照片模糊,为您打造全新的成像体验。
应用实例
2022.06.24
qPCR实验——核酸提取那些事
前言MIQE是描述评估qPCR实验所需的最小信息,该指南是稿件投稿时需要同时提交的一个清单。通过作者提供相关的实验条件和实验特点,审稿人可以评价实验所用方法的可靠性。另外提供详细的实验所用试剂、序列和分析方法,其他研究人员可以利用这些信息进行重复实验。对于核酸提取MIQE有明确要求两点——实验组与对照组的定义及每一组的样本数量。无论采用手工法还是试剂盒,只要符合MIQE的要求并实现良好的可操作性和重现性,就是合理客观的技术方法。MIQE在该部分操作中的要求又分为两大块——样本描述和核酸提取方法。样本描述▶ 样本采集和处理的方法描述▶ 样本的来源描述(宏观解剖还是激光显微切割)▶ 样本是如何即时处理(保存条件及时间)▶ 冷冻样本的冷冻方法和时间核酸提取采取的提取方法:1)手工法:小批量经济型2)自动化仪器:大批量高效型按获取的物质可分为DNA和RNA:◆ DNA:适合拷贝数变异、SNP分析等实验◆ RNA:适合基因表达、长非编码RNA等实验图源:网络,侵删按提取方法分为化学法和机械破碎法:◆ 化学法:适合采用碱裂解法的样本,质粒提取、菌类的提取◆ 机械破碎法:比较坚硬有一定韧性的物质,动植物组织更推荐使用合适的商品化的核酸提取试剂盒,流程标准化程度更高,质量更容易得到保证,这样在方法描述时可以更简单一些,直接引用试剂盒的生产厂家、货号和名称即可。DNA酶或RNA酶的处理细节:基因表达实验中,RNA模板如果存在DNA污染,会严重干扰最后的结果。因此提取RNA时要加入DNA酶将DNA消化掉,工作浓度、处理温度和时间要加以说明,同时也要加入RNase抑制剂提供保护。使用柱纯化试剂盒提取RNA时也可以在柱上进行DNA消化。同样的,提取DNA时也要将RNA消化掉,尽管残留RNA对DNA为模板的qPCR实验影响小得多。 Cielo™实时荧光定量PCR系统☑ 数据可靠性:连续1000次实验后,结果高度一致。☑ 应用灵活性:提供多种qPCR应用分析。☑ 流程智能化:中英文用户界面,触控操作,可多机联用。☑ 在线便捷性:主机可独立运行qPCR程序,数据可USB、Wi-Fi等网络传输。
应用实例
2022.06.22
深蓝云直播 | 3D细胞培养及生物打印干货分享-维度革命
日常的细胞培养大多指的是在培养皿中进行的,也就是2D培养,虽然它在细胞生物学中的研究具有重要的作用,但在模拟体内环境具有一定的局限性。人体是处于动态变化的,因而出现了3D细胞培养,将具有三维结构的材料载体与各种细胞在体外共培养,可以使细胞在三维空间内生长迁移形成三维的细胞-载体复合体,更有利于支持细胞在模拟体内环境下的状态与趋势。常规的3D细胞培养缺少血管化、形成效率低,形态结构和功能方面也会存在很大差异。生物3D打印技术可以很好的解决上述问题,想了解更多生物打印技术,赶快来深蓝云直播间吧。直播时间:2022年6月16日下午3:00主要内容:1、3D和2D细胞培养区别介绍2、3D生物打印介绍3、3D生物打印应用主讲人:叶忠雪,毕业于东北农业大学,具有多年分子和细胞方面的经验,致力于定量PCR,凝胶成像系统,3D生物打印推广和应用工作,对于行业发展有前瞻性研究和见解。 扫描下方海报内二维码报名
企业动态
2022.06.13
qPCR实验中如何科学取材
规划一个qPCR实验时,最先碰到的问题就是研究目的和材料的选择。要想做好一个qPCR实验首先要根据目的选好材料。以目标为导向的取材研究目标的设立是非常重要的一个起始环节,所以对于样本取材要求做到两点,具体准确性和具体代表性。空间异质性个体的生物变异程度(也可以解读为生物多样性)往往是比较高的,这也正是以群体细胞为研究对象的数据往往遭受质疑,而单细胞水平的研究正开展得如火如荼的原因。如何使取到的样本更具代表性?首先,提前做好功课,了解样本的不同分型,或者了解详细的细胞分群。如果条件允许尽可能覆盖所有的组织类型或者细胞类型。其次,尽可能增加样本数量,也就是生物学重复,从而更客观地反映生物变异程度。一般建议生物学重复的数量至少是10。时间异质性生物个体的活动会受到生物节律的调控,细胞的转录水平也同样如此。由于转录的时间异质性,很多时候会导致qPCR的假阴性结果,如果采样时间不合适,会导致基因有表达或者表达量很低的错误结论。因此采样时首先要确认采样时间是否会对最后的结果造成影响,建议做一个比较全面的时间节点研究。样本数量前面建议增加生物学重复,但有时候超过10个的样本数量并不能保证最后能得到有显著表达量差异的结果(假如表达有显著差异是真实存在的),不到10个的样本数量在表达量差异很大的情况下也是足够的,这涉及到统计中的power calculation,很多网站提供在线计算,如http://www.univie.ac.at/ medstat等。Power calculation需要提供被比较的两个组表达量比值、标准差和设定的α和β水平,这样就可以计算出所需要的最少的样本数量。表达量比值和标准差等信息往往不能提前预知,需要查阅之前相似实验的数据,或者通过小规模的实验来获得粗略的信息。一般也可通过估算来得到所需的样本数量,提供一定的参考意义。* power一定的情况检测不同表达量差异所需的最少样本数量[3].采样是需要严格规划的过程,比如材料的时效性、珍贵程度等,都要纳入考量范围。本文讨论的几个因素对qPCR结果的影响是更直接的,需要倍加关注。Azure Cielo™实时荧光定量PCR系统来自美国Azure Biosystems公司,以创新服务于生命科学为愿景。Azure Cielo™实时荧光定量PCR系统融合了高品质Peltier温度模块和基于光纤传输的光学检测系统,为您的科学研究提供高精准、高灵敏的可靠结果。Azure Cielo 3/6检测通道,可根据实验需求灵活配置。同时配有10.3触控系统,主机本身可独立运行、连接、远程交互,让您随时随地知悉实验进程和及时获得实验结果。图源:部分图片来源于网络,侵删
应用实例
2022.06.10
Echo Revolve显微镜助力香棒虫草的生药学与数字化表征研究
前言香棒虫草主产于山西省,主要分布在山西南部中条山一带,民间常用它代替冬虫夏草作为滋补品使用。除了山西,香棒虫草在我国甘肃、云南、青海、广东、海南及国外斯里兰卡和欧洲也有分布。虽山西民间将香棒虫草作为冬虫夏草的替代品,但其未收载进药材标准,且药用历史较短。《中国真菌志》虽明确了其真菌的来源,但对其宿主来源和形态均未有详细的描述。鉴于此,本研究应用性状及显微鉴定法,对香棒虫草的虫体形态、头部特征、子座长出方式、环纹及分节、复毛区刚毛等特征进行详细研究和科学描述,同时与冬虫夏草进行比较,有助于香棒虫草资源的开发及其质量标准的制定,同时可以为冬虫夏草的市场监管和监督检验提供参考依据。本研究应用数码相机、体式显微镜与其数码成像系统对香棒虫草子座和虫体的外观性状特征进行观察和表征;通过冷冻切片和荧光染色,体式荧光显微镜与其数码成像系统、荧光显微镜工作站,对香棒虫草子座和虫体部位的横切面显微特征进行观察和表征;应用扫描电镜对表面及剖面的特征进行探究,并与冬虫夏草进行了生药学鉴别特征比较。作者采用calcofluor white stain试液染色后,在Echo Revolve荧光显微镜 DAPI、FITC和RFP 3个通道下分别观察继发性荧光及自发性荧光,将3个通道的图像叠加,可见虫体内部菌丝层与表皮分别呈紫红及黄色,动物组织与菌丝组织荧光差异明显,见图1。▲ 图1 香棒虫草虫体的横切面(标尺为该图片比例)A-calcofluor white stain染色,3通道叠加(A1-dapi通道;A2-fitc通道;A3-rfp通道);B-直接制片,白光下观察;C-乳酸酚棉蓝染色,白光下观察;D-calcofluor white stain染色,荧光下观察本研究系统阐明了香棒虫草头部上颚、胸足、腹节环节、尾部刚毛及体壁针状毛等性状特征,子座部位不同菌丝层荧光显微特征及虫体部位中虫体组织和菌丝组织荧光显微特征差异。香棒虫草与冬虫夏草相比,在虫体形态、腹足有无、气孔形态、子座长出部位等性状特征,以及体壁被毛、刚毛、毛片等显微特征中存在明显差异。通过对香棒虫草进行生药学研究,可为香棒虫草资源的开发与利用提供参考;通过与冬虫夏草的对比研究,可以避免混淆用药,为市场监管提供科学依据,也为虫草类药用品种数字化表征规范的建立奠定基础。 研究亮点: ▶ 首次采用calcoflouor white stain乳液进行荧光染色,子座与虫体及其不同组织间区别明显,证明该方法可对虫草类药材不同组织结构进行区分和表征。▶ 阐明了香棒虫草与冬虫夏草的区别性特征,可以通过性状和显微特征来区分冬虫夏草与香棒虫草,以防混用及掺伪的情况,也可为粉末和制剂的检验提供参考,同时也为其他混淆品的鉴别研究提供依据。文献原文:doi:10.11669/cpj.2022.06.006Revolve Gen 2正倒置一体电动荧光显微镜新一代Revolve正倒置一体电动荧光显微镜,拥有流行的触屏操控方式,配备智能荧光成像系统,将Z-Stacking全景深成像和DHR数字处理功能有机联合,提升分辨率告别照片模糊,为您打造全新的成像体验。Revolution则是Revolve的升级版,在保留了所有功能的同时,实现了多通道荧光的全切片扫描,20倍镜下3通道荧光,仅需45秒即可扫描完成,系统简洁,APP式样软件操作,任何一位从未接触过该系统的用户,均可快速学会操作,拍出高质量的图像!▶ 高速多通道全切片扫描▶ Apple App触屏操控,界面简洁,极易掌握▶ Apple Store 安装和更新▶ 移动端数据分享更加便捷高效▶ Retina视网膜屏幕高清显示
应用实例
2022.06.09
如何处理实时荧光定量PCR数据
实时荧光定量PCR数据中的基本概念:在整个扩增过程中会出现基线期,指数期,线性期和平台期。其中在基线期和指数增长期,扩增产物都是以指数级增长,但是在基线期我们没办法检测;而到了线性期和平台期之后,由于不同基因,或者不同条件下其扩增效率差别很大,因此没有办法计算模板的含量。因此,在指数增长期的CT值就成了计算模板含量的关键值。▲ 图一:线性图谱▲ 图二:对数图谱为什么知道CT值就能够得到最初的目标基因含量呢?如图三,表示一个基因单次扩增曲线。N为扩增产物的分子数,模板的分子数乘以1+扩增效率的n次方,n代表循环次数。也就是说,如果扩增效率为100%,产物的分子数就等于模板数乘以2的n次方。但是在线性增长期和平台期,扩增效率不可能是100%,因此PCR理论方程只在指数期成立,也就是图三绿色框的部分。▲ 图三数据处理:以下三张图分别表示我们在做荧光定量PCR时提到的三个名称:扩增曲线、标准曲线、溶解曲线。1、绝对定量:使用一系列稀释的已知浓度的标准品与未知样本同时进行测定,根据系列浓度标准品的CT值与起始模板量之间的线性比例关系。注意事项:☑ 标准品必须来源可靠,浓度已知。☑ 标准品要和待测样品同时在仪器中扩增。☑ 只能根据标准品覆盖的浓度范围进行待测样本浓度的推测。2、相对定量:是用来确定经过不同处理的样品之间的表达差异或目标在不同时相差的表达差异,也就是倍数差异。Azure Cielo™实时荧光定量PCR系统来自美国Azure Biosystems公司,以创新服务于生命科学为愿景。Azure Cielo™实时荧光定量PCR系统融合了高品质Peltier温度模块和基于光纤传输的光学检测系统,为您的科学研究提供高精准、高灵敏的可靠结果。Azure Cielo 3/6检测通道,可根据实验需求灵活配置。同时配有10.3触控系统,主机本身可独立运行、连接、远程交互,让您随时随地知悉实验进程和及时获得实验结果。
应用实例
2022.06.06
生物打印肝脏模型评价药物的肝脏毒性研究
背景介绍 药物性肝损伤(DILI)会影响肝脏代谢和解毒能力,但其根本机制仍有很多未知。为了准确和可再现地预测人的DILI,非常需要体外肝脏模型来替代昂贵和低通量的2D细胞培养系统、动物研究和芯片实验室模型。我们提出了一种新的“droplet in droplet”(DID)生物打印方法,该方法可以产生用于肝毒性研究的生理相关肝脏模型。这些模型,或称微型肝脏,是用BIO X微滴打印包裹在ⅰ型胶原中的肝(HepG2和LX2 肝星状细胞)和非肝(HUVEC 人脐静脉血管内皮细胞)细胞制成的。培养7天后,将微型肝脏暴露于急性和高剂量的对乙酰氨基酚或氟他胺,然后评估细胞活力、白蛋白分泌、丙氨酸氨基转移酶(ALT)活性和脂质积累的变化。微型肝脏ALT活性增加,白蛋白和脂质生成减少,表面这两种药物均有细胞毒性反应。这项研究的结果进一步验证了3D生物打印是一种可行的、可用于模拟肝组织和筛选特异性药物反应的中到高通量的解决方案。 材料和方法 细胞准备根据建议的方案培养两种肝细胞(HepG2和LX2)和一种非肝细胞(HUVEC)细胞系,并每3-4天传代一次。HepG2在含有谷氨酰胺的MEMα中生长,并补充1%丙酮酸钠(Gibco,Cat#11360070)和1%MEM非必需氨基酸溶液(Gibco,Cat-#11140050)。LX2细胞在IMDM(Gibco,Cat#12440053)中生长,HUVEC在EGM-2生长培养基(Lonza,Cat#CC-3156)中培养,并添加单体补充剂(Lonza,Cat#CC-4176)。所有培养基均添加10%的FBS(Gibco,16000044类)和1%的青霉素链霉素(Gibco,参考文献1509-70-063)。.生物墨水的制备和DID生物打印中和并制备3mg/mL浓度的Coll I bioink(CELLINK,SKU#IK4000002001)用于生物打印。以1:1:2(LX2:HUVEC:HepG2)的比例将5x106个细胞/毫升装入冷冻墨盒。在未经处理的96孔板(Thermo Fisher Scientific)中,使用BIO X(CELLINK,SKU#0000000 2222)上的液滴打印功能对微型肝脏进行生物打印。使用设置为8°C的温控打印头(TCPH,SKU#0000000 20346)将胶原液滴分配到设置为8°C–10°C的冷却打印床上。在第一轮液滴打印后,样品在37°C下培养3分钟,然后返回BIO X,使用相同参数进行第二轮液滴打印。在37°C条件下,将得到的封装液滴热交联20分钟,并为每个孔提供200微升混合培养基(25%IMDM+25%DMEM+50%MEM)。培养液每2-3天更新一次。药物处理和分析培养7天后,用不同浓度的APAP[0.1,0.5,1,5,10,25,50 mM](Abcam)或FLU[10,25,50,75,100,150,200µM](Selleckchem)处理微型肝脏72小时。采用比色溴甲酚绿(BCG)测定法(Sigma-Aldrich)、ALT活性测定法(BioVision)和活/死染色试剂盒(Invitrogen)分别检测白蛋白产生、肝损伤和细胞活力。所有分析均按照制造商的说明进行。 结论 胶原I中的细胞生长和球体形成胶原I中的细胞生长和球体形成在这项研究中,我们评估了Coll I bioink中的细胞生长、球体形成和迁移模式。到第2天,HepG2和LX2已紧密组装成小簇,HUVEC已拉长,形成同心网络(图1)。使用胶原蛋白作为支架可以在整个培养过程中进行细胞重组、球体极化和细胞增殖(数据未显示)。此外,根据图1,很明显,细胞在整个培养过程中渗透DILI模型,并可能在内部和外部液滴层之间迁移。生物打印微型肝脏的药物治疗和细胞毒性第10天的毒性评估结果表明,生物打印微型肝脏对APAP(图2A)和FLU(图2B)具有细胞毒性和剂量依赖性反应。这种肝功能下降表现为白蛋白分泌和脂质生成减少,ALT活性上调。同样明显的是,基于ALT活性的增加,两种药物的毒性剂量都会对细胞活力产生破坏性影响。后者在图3中尤为明显,其中活/死图像表明,在较高浓度的APAP或流感病毒下,细胞活力显著降低。药物治疗的动态细胞内反应研究了APAP和FLU如何调节细胞内脂肪含量。肝组织的ORO染色通常用于识别脂肪酸或药物引起的不同阶段纤维化或脂肪变性(Pingitore,2019)。在我们的研究中,经处理的微型肝脏的ORO染色显示,在高剂量药物处理的样本中,脂肪积累最小,而在未经处理或低剂量药物治疗的样本中,脂肪积累显著(图4A)。一种解释是APAP和FLU与脂质过氧化有关,其中毒性药物水平引起的氧化应激可能引发脂质降解和膜损伤(Behrends,2019)。图4B中未处理样品的详细观察提供了液滴模型中液滴的横截面图。这张图片显示了大量细胞向液滴外壳迁移并产生脂肪,可能表明存在营养和氧气梯度,并验证了细胞重组模式和胶原内的球体极化。▶ 作为2D细胞培养系统、动物研究和芯片实验室原型的可靠替代品,BIO X可作为中高通量工具,用于制作功能性3D生物打印肝脏模型,实现药物筛选和分析,并减轻药物消耗的成本。▶ CELLINK Coll I作为DID模型的支架,为模型提供了一个稳定、可调和高度相容的环境,且具有丰富的肝细胞重排和球体形成的结合位点。▶ 基于脂质过氧化、白蛋白分泌减少和ALT活性上调的证据,我们的研究结果表明,DID微型肝脏具有功能性,并且对APAP和FLU具有剂量依赖性和细胞毒性反应。▶ DID模型允许组织层之间的细胞间相互作用,并为研究不同硬度层之间的迁移模式提供了独特的机会。未来的毒性研究可以采用该模型复制纤维化的各个阶段,或研究药物治疗后肝脏组织的再生能力。参考文献:1.Behrends, V., Giskeødegård, G. F., Bravo-Santano, N., Letek, M., & Keun, H. C. Acetaminophen cytotoxicity in HepG2 cells isassociated with a decoupling of glycolysis from the TCA cycle, loss of NADPH production, and suppression of anabolism. Archivesof Toxicology. 2019; 93(2): 341–353. DOI: 10.1007/s00204-018-2371-0.2.Chen, M., Suzuki, A., Borlak, J., Andrade, R. J., & Lucena, M. I. Drug-induced liver injury: Interactions between drug properties andhost factors. Journal of Hepatology. 2015; 63: 503–514. DOI: 10.1016/j.jhep.2015.04.016.3.Pingitore, P., Sasidharan, K., Ekstrand, M., Prill, S., Lindén, D., & Romeo, S. Human multilineage 3D spheroids as a model of liversteatosis and fibrosis. International Journal of Molecular Sciences. 2019; 20(7): 1629.
应用实例
2022.06.01
CELLINK GMP 实验室 - 打印医学未来
• CELLINK的故事 •CELLINK 致力于将人体器官和组织生物制造提升到一个新的水平,符合 BICO 在生命科学中的憧憬:利用生物(与科技) 的融合为人类健康创造出崭新的医学未来。• 符合GMP标准 •CELLINK在位于Kallebäck公司成立了首个良好生产规范(GMP)实验室,为人类应用提供临床级生物墨水。我们的实验室遵守严格的FDA和欧盟质量和制药安全法规,这即将是我们踏入临床应用的第一个步骤。据市场报告,组织工程医疗产品是一个价值20亿美元的行业,根据规定,在开发TEMP过程中所使用的任何材料都必须符合GMP要求。随着该实验室的开放,CELLINK相信将能够成为生物材料的首要供应商。我们能够更好地为客户服务,并作为他们的合作伙伴,继续帮助他们推动组织工程和再生医学的发展。这将会是减少器官短缺现况的重要转折点。• 临床级生物墨水 •纯优质生物材料保持较低的批次间可变性可靠的重复结果• 如何联系我们 •深蓝云生物科技有限公司为CELLINK在中国的分销商,如果您有任何需要帮助或希望了解我们的产品和服务信息,可随时联系。电话:010-57256059
应用实例
2022.05.25
深蓝云直播课 | 数字PCR:核酸绝对定量技术原理及其在工业领域的应用
数字PCR技术具备不依赖于标准品、灵敏度高、抑制剂耐受力高以及可区分微小差异等技术特性,是能够直接对目标核酸进行绝对定量的有力检测工具。数字PCR技术在液态活检、产前诊断、病原体筛查、转基因动植物、遗传育种、环境监测方面都有广泛的应用。此外,在细胞治疗及基因治疗等先进治疗领域还能够实现对核酸标准品及质控品定值、病毒基因组(如rAAV VG)滴度测定、GOI拷贝数检测、残留检测以及外源污染物等的检测。新一代微滴芯片数字PCR技术,集微滴式和芯片式数字PCR优势于一体,能够实现自动微滴体积质控和智能可视化数据分析,一步操作,双倍效能,为核酸绝对定量提供可靠的检测工具。本期我们邀请法国Stilla数字PCR系统应用专家王琳琳老师,为大家介绍数字PCR的技术原理、技术优势以及前沿应用。直播时间:2022年5月26日下午3:00主要内容:1、数字PCR如何实现核酸绝对定量2、数字PCR的技术特点和应用优势3、数字PCR在工业CGT等领域的应用进展主讲人:王琳琳,法国Stilla naica微滴芯片数字PCR系统应用专家。毕业于四川大学,细胞生物学专业,从事数字PCR技术数年,在数字PCR技术原理、应用以及方法学验证方面具有丰富的经验。 扫描下方海报内二维码报名
企业动态
2022.05.23
比较 2D 培养和 3D 生物打印肿瘤模型中的药物反应
导读在癌症生物学中,肿瘤微环境(TME)是肿瘤细胞和免疫系统之间的一个关键。TME是细胞外基质(ECM)、免疫细胞、信号分子、血管和成纤维细胞,它们包裹肿瘤并影响癌症进展。TME的成分通过分泌小信号分子相互作用,影响肿瘤行为的各个方面,包括细胞增殖、侵袭、转移和抗肿瘤治疗的耐药性(Bremnes,2011)。因此,重建TME对抗癌研究至关重要,但一个主要的痛点是无法开发出可预测的3D肿瘤模型用于高通量药物评估。3D肿瘤模型应再现肿瘤间质内细胞间的相互作用,并克服2D细胞培养系统的局限性。在这里,3D生物打印为预测体内结果、建模TME和评估药物反应提供了一个有前景的解决方案。肿瘤转移和化疗耐药性威胁着肿瘤患者的生存。在癌症治疗领域,化疗是一种很有效的治疗方式,它利用小的抗癌分子攻击特定的生长途径并杀死癌细胞。在这些分子中,顺铂(CIS)和吉非替尼(GEF)是FDA批准的靶向DNA和EGFR通路的抗癌药物。简而言之,CIS通过抑制细胞分裂和 mRNA的产生导致细胞凋亡,而GEF干扰癌细胞中EGFR信号的上调。有趣的是,虽然CIS和GEF都被用于治疗致命的胰腺癌和乳腺癌,但它们也与体外假阴性或假阳性预测有关,这表明它们在2D和3D中对细胞的影响不同(Reynolds, 2017)。为了进一步解决这一差异,我们使用两种乳腺癌(MCF7, MDA MB 231)和两种胰腺癌(BxPC3, Panc-1)细胞系,比较了CIS和GEF对2D单层细胞和3D生物打印类肿瘤模型的作用。材料和方法生物墨水制备和生物打印根据CELLINK方案制备3 mg/mL Coll 1 (CELLINK, Ref #IK4000002001)和5% GelMA (CELLINK, Ref #IK3051020303)用于生物打印。共3ml Coll 1或GelMA与5 x 106 cells/100µL培养基(10:1)混合,分别装入透明和琥珀色墨盒(CELLINK, Ref #CSO010311502),以~ 3kpa进行液滴打印。使用温度控制的打印头(TCPH, SKU #000000020346)设置为8℃,气动打印头分别在8℃的打印床上对Coll 1和GelMA液滴进行生物打印。使用BIO X (CELLINK, SKU #000000022222)上的液滴打印功能,将每种生物墨水打印在未经处理的96孔板(Thermo Fisher Scientific, Cat #267427)上。打印完成后,Coll 1液滴在37℃下热交联20分钟,GelMA液滴在365 nm下紫外交联6秒。每孔加100µL培养基,每2 ~ 3天更换一次。2D单层培养为了进行2D比较,将每个细胞株接种在处理过的96孔板上(Thermo Fisher Scientific, Cat #167425)。优化各细胞培养48小时后的细胞密度,达到90%的一致性。Panc-1细胞接种1.2 × 104个细胞/孔,BxPC3细胞接种1.7 × 104个细胞/孔,MCF7细胞接种2.0 × 104个细胞/孔,MDA MB 231细胞接种2.0 × 104个细胞/孔。药物治疗与分析生物打印类肿瘤细胞和2D细胞分别用不同浓度的吉非替尼(LC Laboratories,#G-4408)或顺铂(Cayman Chemical Company)处理96小时和48小时。MTS Assay(Sigma-Aldrich)和LIVE/DEAD染色试剂盒(Invitrogen)用于评估2D和3D条件下的细胞活力。所有的检测都是按照制造商的说明进行的。图1:该测定的优点显示了抗肿瘤药物对所有4种细胞系的强大作用,并描述了每种细胞类型和ECM的细胞形态变化。比例尺:1000m或650m。绿色:LIVE,红色:DEAD肿瘤根据细胞类型和培养条件适应不同的形态(Nath, 2016)。在GelMA和Coll 1中培养7天后,癌细胞聚集形成各种形态的球体。如图1所示,MDA MB 231细胞形成同心星形网络,MCF7细胞形成圆形椭球,BxPC3细胞形成葡萄状椭球,Panc-1细胞形成团块状椭球。使用GelMA和Coll 1作为肿瘤支架,由于孔隙度、刚度和成分的不同,也影响了球状体的形成。有趣的是,2D培养的癌细胞缺乏所描述的形态,可能是因为它们缺乏支持细胞间相互作用、紧密连接、营养和氧梯度的ECM(数据未显示)。3D模型的缺氧效应缺氧是药物反应的另一个变量,这是3D模型和体内组织所特有的。Warburg效应将缺氧描述为癌细胞的一种生存模式,它们从生产氧气和ATP转换为上调EGFR和AKT信号以促进增殖。这种转换增加了毒性、酸度和3D模型中的废物堆积,从而产生了一个三环低氧梯度。图1显示了低氧梯度,其中靠近球体中心的细胞呈死亡状态(红色),边缘的细胞呈存活状态(绿色)。最外面的环是一层增殖细胞,中间的环是一层活细胞,最里面的环是坏死细胞的核心,这是由于废物堆积和缺氧造成的(Nath, 2016)。顺铂在2D和3D模型的疗效分别在第2天和第7天,将低到高剂量的CIS添加到2D单层细胞和3D生物打印类肿瘤细胞中。2D细胞处理治疗48小时,3D生物打印类肿瘤治疗96小时。MTS试验显示,2D单层对所有细胞株的细胞毒性均呈剂量依赖性,3D乳腺癌类肿瘤细胞也是如此(图2A)。有趣的是,BxPC3和Panc-1细胞株在3D中比在2D中显示更高的IC50。换句话说,这两种胰腺癌细胞株在3D生物打印类肿瘤中基本上不受CIS的影响。这里,一种解释是胰腺癌细胞对CIS浓度的增加表现出了耐药性(Wang, 2016;凯兰,2007;Sangster-Guity, 2011)。针对药物治疗,胰腺癌细胞可能已经诱导了他们的生存途径,上调衰老、DNA损伤反应信号转导和跨损伤DNA合成(Gomes, 2019年)。吉非替尼在2D和3D模型的疗效EGFR癌蛋白常在乳腺癌和胰腺癌细胞系中表达。因此,药物抑制EGFR通路可导致细胞周期阻滞、衰老或凋亡(Jacobi, 2017)。如图2B所示,在3D和2D中,吉非替尼显著降低了细胞活力。对于所有细胞类型,3D Coll 1和GelMA的IC50均低于2D培养的IC50,这表明GEF在3D生物打印类肿瘤细胞中比在2D培养中造成更多的死亡。2D细胞培养的局限性2D细胞培养系统不能模拟体内肿瘤的内在特性,包括自然屏障、低氧梯度和紧密的细胞-细胞连接,这些都减缓了药物扩散。此外,它们缺乏支持3D生长和癌蛋白上调的组织特异性环境和ECM (Reynolds, 2017)。图2A的另一项研究显示,3D胰腺癌细胞比2D单层细胞对CIS的抗性更强。很明显,2D研究对于胰腺癌的体内治疗是一种误导和不准确的预测。结论使用CELLINK GelMA和Coll 1作为类肿瘤支架,为球状形成和药物扩散提供了稳定的肿瘤微环境(TME)。用GelMA和Coll 1构建的不同杀伤曲线模型表明,细胞外基质(ECM)在药物反应中起关键作用。未来的研究需要确定哪种支架适合特定的肿瘤模型。我们的研究结果显示,在2D和3D肿瘤模型中,顺铂(CIS)和吉非替尼(GEF)治疗具有剂量依赖性和细胞特异性反应。乳腺癌和胰腺癌细胞株在3D条件下比2D条件下对GEF更敏感。同样,乳腺癌细胞株3D对CIS治疗的敏感性高于2D,而胰腺细胞株对CIS治疗的敏感性则相反,提示3D模型的耐药水平升高。3D生物打印类肿瘤模型用于药物筛选,可用于减少假阴性和假阳性预测。未来的研究可以使用BIO X来扩大类肿瘤的生产,用于高通量药物测试。
应用实例
2022.05.18
深蓝云直播 | 细胞培养的好帮手
养细胞离不开看细胞和数细胞,方便快速成本低的完成细胞观察和细胞计数是每个细胞培养者每天都要面对的问题。一般的细胞房面积小,所以仪器的小型化多功能化是必备条件。有没有一款仪器可以解决上述的所有问题呢?本次直播为大家带来来自美国Echo公司的解决方案,欢迎大家参加。直播时间:2022年5月19日下午3:00主要内容:1、细胞计数原理介绍2、使用显微镜如何进行明场拍照和细胞计数主讲人:李晓丹,显微镜高级应用专家,从事显微镜行业多年,在显微镜使用、应用和行业发展方面有丰富的经验和深入研究。 扫描下方海报内二维码报名
企业动态
2022.05.16
Drop-off-数字PCR方法快速检测和定量废水中新冠病毒及变种VOCs
废水中检测到新冠病毒和新冠变种,在世界各地的污水厂都有类似的报道。今年初,美国疾病控制预防中心 (CDC)发布的报告中,指出新冠病毒变种奥密克戎(Omicron)很有可能在美国首例确诊病例官方声明前一周已经存在于纽约市的废水中,报告同时指出,废水监测是一个可行的早期预警系统,可以帮助跟踪新冠病毒变种的传播。尽管对废水中的SARS-CoV-2 RNA测序的方法极具应用意义,可以从单个样本中追踪数百人到数千人的新冠变种VOCs,但在样本收集和分析方面存在时间滞后,测序和生物信息学管道通常需要至少3天的时间才能得到结果,为了能更快速地监测,科学家们开发了基于PCR的分析方法,以识别其特征突变(Vogels等人,2021;Bedotto等人,2021;Chaintoutis等人,2021)。例如,针对单核苷酸多态性的检测,如突刺蛋白中的N501Y和E484K,或特征缺失,如突刺Δ69-70和ORF1a Δ3675- 3677。瑞士科学家Lea Caduff等使用drop-off数字PCR方法开发快速、高通量的方法来检测和量化废水中新冠变种的两种缺失频率,开发统计方法监测特异性突变随时间的动态变化,从而量化新冠变种的传播速度,数据与临床样本测序结果一致。该方法基于naica®微滴芯片数字PCR系统,针对废水中新冠病毒特异性突变的数字PCR检测提供了近实时的SARS-CoV-2 新冠变种VOCs监测,并可能比临床测序更早地检测和推断新冠变种VOCs的传播速度,方法发表于文章“Inferring Transmission Fitness Advantage of SARS-CoV-2 Variants of Concern in Wastewater Using Digital PCR。DOI:https://doi.org/10.1101/2021.08.22.21262024。应用亮点:▶ 利用naica®微滴芯片数字系统,开发出drop-off方法检测废水中SARS-CoV-2变种的突变频率。▶ 通过对大量废水及临床样本的检测分析,发现废水中SARS-CoV-2变种的传播适应性与临床样品一致。▶ Drop-off RT-dPCR方法为社区内VOCs的快速检测和定量提供了机会,可用于基于SARS-CoV-2的废水流行病学的大规模检测调查。▶ 与临床或废水测序相比,drop-off RT-dPCR检测提供了一种在社区内跟踪突变的方法,成本更低,检测速度更快,而且可以用于筛选VOCs的其它突变。在COVID-19全球大流行期间,SARS-CoV-2令人担忧的遗传变异(VOCs)多次出现。VOCs的特征是传播性增强,毒性增强,或通过先前感染或接种疫苗获得的抗体的中和作用减弱。通常追踪VOCs的输入和传播依赖于测序,对临床样本进行全基因组测序。现在废水监测越来越多地被用于通过测序方法跟踪SARS-CoV-2变种的输入和传播。虽然废水中SARS-CoV-2 RNA测序是一种很有前途的方法,可以在一个样本中跟踪成百上千个人的VOCs,但样本收集和分析存在时间滞后,这与临床测试相关的滞后类似。在得到RNA样本后,测序和生物信息学分析通常需要至少3天的时间才能得到结果。为了更快速地识别VOCs,我们开发了基于PCR的快速、高通量的方法来检测和定量废水中B.1.1.7、B.1.351和P.1 VOCs的两种突变频率。我们进一步开发了一种统计方法来分析RT-dPCR检测数据的时间动态,以量化传播适应性,获得与从临床样本获得的相似的数据。针对废水中的SARS-CoV-2数字PCR检测提供了对VOCs的实时监测,并可以比临床测序更早地发现和推断传播适应性。在一个RT-dPCR检测中,使用两个非竞争性水解探针,针对同一扩增子上的保守区域,量化废水样品中的突变频率(“drop-off RT-dPCR检测”),可以同时定量野生型(不携带突变的菌株)和突变型(携带突变的菌株)(图1)。▲图1 基于两种不同探针的drop-off RT-dPCR检测方法概述:一种drop-off探针(Deletion Probe)和一种Reference探针(Universal Probe)。(A)Reference探针可同时与野生型和突变型结合,而drop-off探针只与野生型结合。(B)在dPCR 2D散点图中,野生型为双阳性液滴(棕色),而突变型为单阳性液滴(红色)。在2020年12月7日至2021年3月26日期间,对从废水处理厂Werdhölzli(服务于瑞士苏黎世约45万人)收集的32个原始进水(24小时流量比例复合)样本进行了drop-off RT-dPCR检测,并与在苏黎世收集并测序的2497个临床样本进行对比。使用drop-off RT-dPCR方法在废水中检测的突变的时间趋势与临床样本的测序数据一致(图2)。▲图2 与苏黎世临床样本相比,废水样本中 (A) spike Δ69-70 和 (B) ORF1a Δ3675-3677 的突变频率,以及苏黎世临床样本中 B.1.1.7 谱系的突变频率。拟合曲线对应于废水和临床数据缺失的三参数拟合和 B.1.1.7 临床数据的两参数拟合。阴影区域对应于 95% 的置信带。(C) spike Δ69-70 和 (D) ORF1a Δ3675-3677 的 SARS-CoV-2 RNA(灰色)浓度和缺失等位基因(蓝色)浓度,在废水样品中。(E) spike Δ69-70 和 (F) ORF1a Δ3675-3677,苏黎世测序的临床样本数量(灰色)和缺失等位基因(红色)的样本数量。(G, H) 苏黎世测序的临床样本数量(灰色)和B.1.1.7 谱系的样本数量(橙色)。使用dPCR技术跟踪废水中VOCs的特征突变(ORF1aΔ3675-3677和spikeΔ69-70)的时间趋势可以迅速告知变异传播性。来自废水的生长速率和传播适应性估计值与来自临床样本的估计值基本一致,95%置信区间重叠。由于dPCR样本分析比全基因组测序更快,一旦识别出特征突变,可以帮助我们早期洞察新变异的传播性。本研究可以帮助大家进行SARS-CoV-2变种的传播性研究。参考文献:1、Bedotto, Marielle, et al. 2021. “Implementation of an in-House Real-Time Reverse Transcription-PCR Assay for the Rapid Detection of the SARS-CoV-2 Marseille-4 Variant.” Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology 139 (June): 104814.2、Chaintoutis, et al. 2021. “A One-Step Real-Time RT-PCR Assay for Simultaneous Typing of SARS-CoV-2 Mutations Associated with the E484K and N501Y Spike Protein Amino-Acid Substitutions.” medRxiv. https://doi.org/10.1101/2021.05.31.21257367.3、Fernandez-Cassi, et al.. “Wastewater Monitoring Outperforms Case Numbers as a Tool to Track COVID-19 Incidence Dynamics When Test Positivity Rates Are High.” https://doi.org/10.1101/2021.03.25.21254344.4、Crits-Christoph, et al. 2021. “Genome Sequencing of Sewage Detects Regionally Prevalent SARS-CoV-2 Variants.” mBio 12 (1). https://doi.org/10.1128/mBio.02703-20.
应用实例
2022.05.13
PCR原理、PCR扩增影响因素及预防解决方案
PCR简介聚合酶链反应(polymerase chain reaction,PCR)是利用一段DNA为模板,在DNA聚合酶和核苷酸底物共同参与下,将该段DNA扩增至足够数量,以便进行结构和功能分析的一种反应。PCR扩增原理核酸降解是DNA/RNA分子中的碱基和戊糖间的氮糖苷键,或磷酸二酯键在物理因素、化学因素和生物因素等作用下发生水解,使DNA/RNA链发生断裂。▲ 图一:PCR原理反应示意图▲ 图二:PCR反应过程中温度变化图实时荧光定量PCR原理通过荧光染料或荧光标记的特异性探针,对PCR产物进行标记跟踪,实时监控反应过程,结合相应软件可以对结果进行分析,通过标准曲线对未知模板进行定量分析,计算待测样本的初始模板浓度。▶ 初始DNA浓度越高,荧光达到某一值(阈值)时所需要的循环数越少(Cq值)。▶ Log浓度与循环数成线性关系,根据样品扩增到阈值的循环数与已知起始拷贝数的标准品作出的标准曲线对比就可以计算出该样品的起始拷贝数。影响PCR扩增的因素▶ 模板间的交叉污染。▶ PCR试剂的污染。▶ PCR产物的污染。防止污染的预防操作❶ 永远要设置NTC(No Template Control)对照,一个不含有模板DNA但含有PCR体系中所有其他成分的对照。如果不能在污染的第一时间发现,会导致后续一系列的数据无法使用。❷ 准备PCR体系的移液器要专用,千万不能用吸取过PCR产物的移液器去准备PCR体系。❸ 打开离心管前先离心,开管动作要轻,以防管内液体溅出。❹ 最好在加完其他反应成分再加入模板。❺ 实验结束后及时清理台面。出现污染后的解决办法❶ 更换试剂:更换新的试剂和水,用确保无污染的移液器分装备用。❷ 清洁所有可能的污染源:实验台面,离心机,门把手等。❸ 实验过程更加小心,采用前面提到的各种防止污染的方法。CieloTM实时荧光定量PCR系统Harness of the power of qPCR☑ 数据可靠性:连续1000次实验后,结果高度一致。☑ 应用灵活性:提供多种qPCR应用分析。☑ 流程智能化:中英文用户界面,触控操作,可多机联用。☑ 在线便捷性:主机可独立运行qPCR程序,数据可USB、Wi-Fi等网络传输。
应用实例
2022.05.10
细胞培养好,实验 so easy
一提到细胞培养,这是多少实验者的噩梦,简直……说多了都是泪。小编经过软磨硬泡终于从师兄师姐那里讨到了细胞培养秘籍,今天就大方一回,分享给大家。★ 血清与培养基的选择一般实验血清的添加比例为10%,也可根据细胞状态和生长速率适当增加或减少添加比例。对于培养基,建议选择商品化的,比较成熟,稳定性也较好。★ 培养瓶的选择目前商品化的细胞培养瓶主要为瓶盖是否带气孔两种。其中不带透气孔细胞培养瓶拧紧后需适当回旋瓶盖,保证CO2能顺利进入细胞培养瓶。细胞培养过程中,对于适应能力强的肿瘤细胞,可在传代3-4次后更换新的细胞培养瓶。对于非肿瘤细胞系如293T等细胞,建议每次传代更换新的细胞培养瓶来保证细胞状态。★ 细胞传代传代时通常使用胰酶消化法。该方法又分为干消化法和湿消化法。▶ 湿消化法:待细胞变圆,成流沙状脱落时即可加入新鲜培养基终止消化,1000rpm离心5min,弃去上清,用新鲜培养基重悬传代。▶ 干消化法:胰酶浸润后弃去胰酶,待细胞变圆后加入新鲜培养基吹下后再进行细胞传代。不同细胞的细胞间连接强度不一样,消化时间不同;不同细胞的贴壁能力不同,消化时间不同;消化时应观察细胞形态,避免因过度消化影响细胞活性。★ 细胞冻存与复苏当细胞生长状况较好或者代数较早时,需及时大量的冻存。细胞冻存密度通常为1-3×106个/ml。通常4℃放置0.5h,-20℃放置2h,-80℃冰箱中过夜,取出冻存管,移入液氮容器内。细胞复苏38℃水浴不时摇动,在1分钟内使其完全融化,1000rpm离心5min,弃去上清,用新鲜培养基重悬移入培养瓶中。★ 污染防控细胞培养最基本的是做好污染防控,包括微生物污染的防控和细胞系间交叉污染的防控。实验中需保持良好的操作习惯,所用材料的位置摆放要顺手,污染源和已灭菌物品要分开放置。实验室也需定期清洁与消毒。ECHO强大的活细胞成像工作站INCUBATOR+REVOLUTION智能电动化显微镜
应用实例
2022.05.06
qPCR中CT值的那些事
导读Ct值是荧光定量PCR重要的结果呈现形式,它被用于计算基因表达量差异或者基因拷贝数。那么荧光定量的Ct值多大被认为是合理?如何保证Ct值的有效范围呢? Ct值以及Ct值的作用:Ct值概念:也称循环阈值,C代表Cycle,t代表threshold。其含义是:在PCR 循环过程中,荧光信号开始由本底进入指数增长阶段的拐点所对应的循环次数。Ct值的作用:1)Ct值的重现性,PCR循环在到达Ct值所在的循环数时,刚刚进入真正的指数扩增期(对数期),此时微小误差尚未放大,因此Ct值的重现性极好,即同一模板不同时间扩增或同一时间不同管内扩增,得到的Ct值是恒定的。注:模板为105k拷贝GAPDH,用GAPDH引物扩增。96个孔重复结果的Ct平均值为19.1,变异系数(Cv)=0.002.2)Ct值与起始模板的线性关系,由于Ct值与起始模板的对数存在线性关系,可利用标准曲线对未知样品进行定量测定,因此,实时荧光定量PCR是一种采用标准曲线定量的方法。注:样本进行10倍的倍比稀释后进行荧光定量检测得到的Ct值。 合理的Ct值范围:Ct值的范围为15-35,Ct值小于15,认为扩增在基线期范围内,未达到荧光阈值。理想情况下,Ct值与模板起始拷贝数的对数存在线性关系,也就是标准曲线。通过标准曲线,扩增效率为100%时,计算出基因单个拷贝数定量的Ct值在35左右,若大于35,理论上模板起始拷贝数小于1,可认为无意义。对于不同的基因Ct范围,因起始模板量中基因拷贝数和扩增效率不同,需做出该基因的标准曲线,计算出基因的线性检测范围。 影响Ct值的因素及解决办法:由扩增循环数和产物量之间的关系:扩增产物量=起始模板量×(1+En)循环个数,可以看出,在理想条件下,起始模板量和En(扩增效率)会对Ct值产生影响。模板质量或者扩增效率的差异会造成Ct值偏大或过小。CieloTM实时荧光定量PCR系统Harness of the power of qPCRCielo™实时荧光定量PCR系统可以为您提供3/6检测通道:☑ 高能LED光源:光源强度高,重复性好。☑ 帕尔贴温控系统:升降温速率快,可设置12列跨度为30℃的温度梯度。☑ 光纤信号传导+CMOS拍照:传输快,灵敏度高,无光损失和噪音干扰。☑ 无荧光串扰:检测更精准
应用实例
2022.04.27
naica®微滴芯片数字PCR系统量化新冠病毒用于COVID-19 住院患者临床监测指标
导读目前新型冠状病毒肺炎(COVID-19)已经在全球范围内呈大流行趋势,为全球社会带来重大损失。大约15-30% 的住院 COVID-19 患者出现急性呼吸窘迫综合症、全身性组织损伤和/或多器官衰竭,导致大约 45% 的病例死亡。因此非常需要生物标志物来量化组织损伤、预测临床结果并指导住院 COVID-19 患者的临床管理。近日来自巴黎公共援助医院微生物学系的科学家在《Clin Infect Dis.》发表了一篇文章,题目为“Circulating Ubiquitous RNA, A Highly Predictive and Prognostic Biomarker in Hospitalized Coronavirus Disease 2019 (COVID-19) Patients”。文章中运用naica®微滴芯片数字PCR对SARS-CoV-2 RNAemia和核糖核酸酶P (RNase P) RNAemia进行定量分析,结果表明这两种标志物可以用于COVID-19住院患者的临床监测和管理。应用亮点:▶ 利用naica®微滴芯片数字PCR对SARS-CoV-2 RNAemia和核糖核酸酶P (RNase P) RNAemia进行检测。▶ 患者入院时SARS-CoV-2 RNAemia和RNase P均与临床严重程度和有创机械通气(IMV)有相关性。▶ 入院时血浆RNase P RNA浓度与生存率有相关性。首先作者利用naica®微滴芯片数字PCR对139例COVID-19患者入院时血浆中SARS-CoV-2 RNAemia和核糖核酸酶P (RNase P) RNAemia进行定量分析。随后采用统计学公式对两种标志物在COVID-19患者入院时的临床严重程度方面的价值进行评估,并将它们与患者治疗期间的临床结果进行关联。结果表明:患者入院时SARS-CoV-2 RNAemia与临床严重程度和有创机械通气(IMV)有相关性,与患者入院时CT扫描显示的肺部严重程度没有相关性(图1)。▲图1. A,139例临床严重程度不同的COVID-19患者的SARS-CoV-2 RNAemia浓度。B,根据IMV状态分析139例COVID-19患者的SARS-CoV-2 RNAemia浓度。C, SARS-CoV-2 RNAemia浓度(log copy/mL)与肺部严重程度的相关性。血浆RNase P浓度与临床严重程度等级和有创机械通气状态高度相关,RNase P RNAemia与患者入院时CT扫描显示的肺部严重程度之间没有相关性(图2)。▲图2. A,139例COVID-19患者根据临床严重程度的血浆RNase P浓度。B,根据IMV状态分析139例COVID-19患者血浆RNase P浓度。C,RNase P RNAemia浓度(log copy/mL)与肺严重程度的相关性。患者入院时血浆RNase P RNA浓度与治疗期间的死亡显著相关,而SARS-CoV-2 RNAemia值并不能预测死亡率(图3)。▲图3. RNase P RNAemia的总生存率(log copy/mL)因此,在住院的COVID-19患者中,SARS-CoV-2 RNAemia和普遍存在且具有特异性的人类细胞内RNA标记物RNase P可以作为生物标志物,指导COVID-19住院患者的管理,提供准确的预后。期刊介绍:Clin Infect Dis杂志是美国感染病协会(IDSA)的官方出版物,主要刊登感染病学各方面的原创前沿研究和一些综述性文章,致力于将研究结果应用于临床,直到临床医生对感染性疾病的诊治。CID还定期发布感染性疾病最新治疗指南。CID杂志影响因子为8.195,在所有感染病专业期刊中排名第一。
应用实例
2022.04.26