









转座在生物体基因重塑过程中具有关键性的作用。IS200/IS605 和 IS607 家族的插入序列(insertion sequences, ISs)是最简单的可移动遗传元素之一,仅包含其转座和调节所需的基因。2021年9月9日,张锋团队在Science杂志上发表了一篇题为 The widespread IS200/605 transposon family encodes diverse programmable RNA-guided endonucleases 的文章(详见BioArt报道:Science | 张锋团队再发文:源于IS200/605转座子家族的多种核酸酶或可成为基因编辑新工具)【1】,重建了CRISPR-Cas9系统的进化起源,发现了3种高度丰富但功能未知的转座子编码的可编程RNA引导核酸酶:IscB、IsrB和TnpB,并对IscB的蛋白功能进行了详细探究。该研究还发现TnpB可能是CRISPR-Cas9/Cas12核酸酶的祖先,推测TnpB也具备RNA引导的核酸酶活性。
近日,来自立陶宛维尔纽斯大学的Virginijus Siksnys团队(CRISPR开创者之一,通过研究CRISPR-Cas9 生化性质,得出了Cas9酶可以定点切割DNA的结论,特别致敬CRISPR幕后的英雄们——最全的一份CRISPR英雄谱)在Nature杂志上在线发表了题为Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease的研究论文。研究人员通过一系列生化实验证实CRISPR-cas核酸酶的祖先TnpB是可重编程的 RNA 引导的功能性核酸酶。TnpB通过reRNA(right element RNA,长非编码RNA,来源于ISDra2转座子中的RE元件)引导去切割靠近TTGAT转座相关模块(Transposon associated motif, TAM)5’端的DNA,并可以切割人类基因组DNA。

如图1所示,IS200/IS605家族的Deinococcus radiodurans ISDra2中包含tnpA和tnpB基因,以及位于两侧的LE(Left element)和RE(Right element)。在纯化TnpB的过程中,作者发现有许多RNA也被一同纯化。对TnpB结合的RNA进行small RNA测序(sRNA-seq)分析,发现它们大多是长度约为150nt的长非编码RNA,来源于ISDra2中的RE序列,作者将这些RNA称为reRNAs(right element RNA)。reRNA 3’端的16nt来源于IS200/IS605转座子的侧翼DNA序列,其余序列与TnpB基因的3’端和RE序列匹配,说明TnpB可以与转座子3’端来源的reRNA形成RNP复合物。
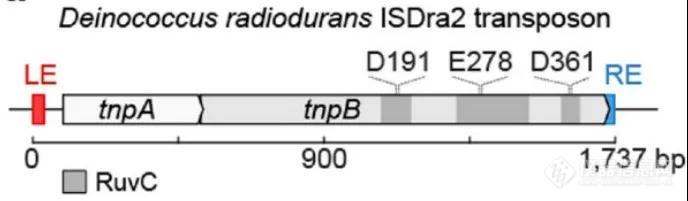
图1. D. radiodurans ISDra2位点
PAM(protospacer adjacent motif)序列是Cas9或Cas12核酸酶启动DNA切割所必须的,那么TnpB发挥作用可能也需要类似的序列。通过PAM鉴定实验【2】,作者观察到在目标基因5’端上游富集了大量的TTGAT序列,并称之为Transposon Associated Motif (TAM)。体外DNA切割实验证实TnpB具备RNA引导的靶向dsDNA的核酸酶活性。进一步分析发现,将TnpB序列中的RuvC-like活性位点突变后,TnpB失去了切割能力,说明RuvC模块与TnpB的活性相关。研究人员发现实现TnpB的DNA切割功能需要同时满足两个条件:(1)TAM序列;(2)与靶基因匹配的位于reRNA 3’端的序列。随后,作者对切割产物进行了测序分析,结果显示,TnpB采取的是一种交错切割模式,在NTS(non-target strand)的多个位置和 TS (target strand) 的单个位置进行切割,最终产生5’-悬挂端(overhangs)(图2)。
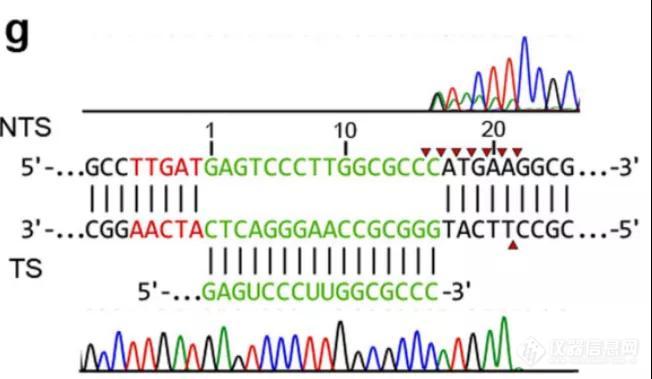
图2. TnpB-reRNA复合物切割双链DNA的实验设计及流程
最后,作者探究了TnpB在细胞内切割目标dsDNA的能力。首先,在E.coli中进行的质粒干扰实验表明TnpB可以在原核生物体内切割dsDNA。紧接着,作者想知道TnpB是否可以应用于切割人类基因组。将编码TnpB和reRNA的工具质粒转染至HEK293T细胞中,72小时后,提取基因组DNA(gDNA)测序分析目标切割位点中的序列插入和删除(insertions and deletions,indels)情况(图3)。实验结果显示,TnpB在两个测试靶点(AGBL1-2和EMX1-1)中引入突变的频率为10%-20%,这与之前报道过的CRISPR-Cas9和Cas12的效率类似【3-7】。进一步分析发现,切割位点引入的删除突变占主导地位,与Cas12切割谱中观察到的现象类似【5,7】。这些结果说明RNA引导的TnpB核酸酶可以切割真核生物的基因组DNA。
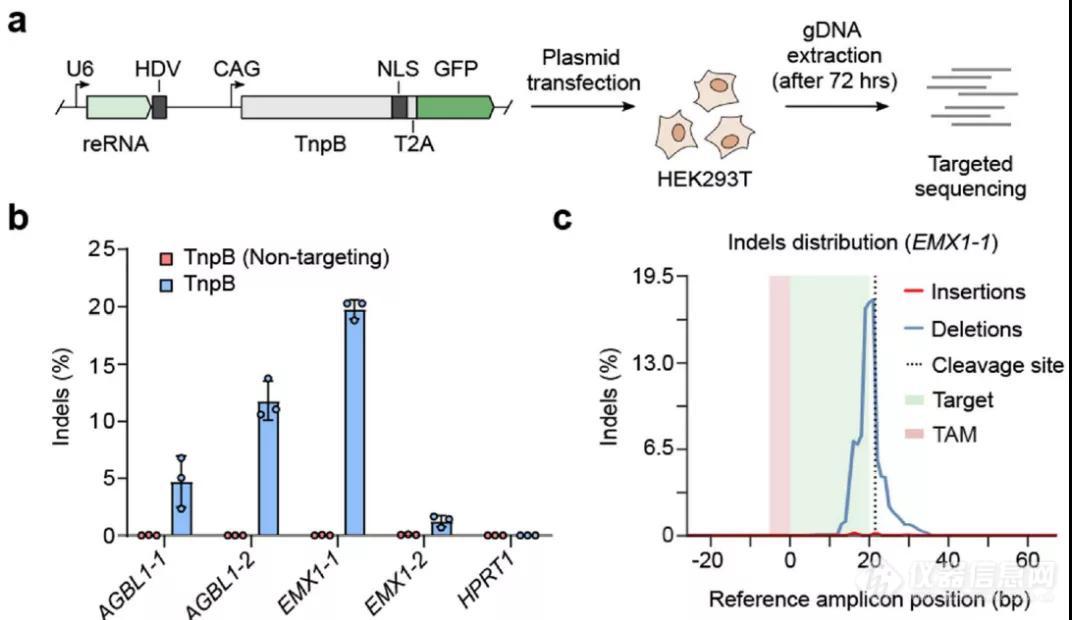
图3. 利用TnpB编辑人类基因组
综上所述,该研究从ISDra2系统中鉴定了一个新的具有dsDNA切割功能的核酸酶TnpB,其在原核和真核细胞中均能有效切割dsDNA,具有编辑人类基因组的巨大潜力。在进化树上,虽然TnpB与微型 Cas12f核酸酶的关系最为紧密,但作者认为两者之间依旧存在重大区别:(1)TnpB与Cas12f使用的guide RNA不同;(2)TnpB是单体,仅需要一个reRNA;而Cas12f 核酸酶是二聚体,需要结合一个拷贝的crRNA-tracrRNA duplex;(3)TnpB需要TAM序列,Cas12f需要PAM序列,两种序列截然不同。
原文链接:
https://doi.org/10.1038/s41586-021-04058-1
[来源: BioArt]
 手持拉曼光谱仪检测血红细胞
手持拉曼光谱仪检测血红细胞
文献速递丨naica®微滴芯片数字PCR系统与CRISPR基因编辑技术强强联合
2021.09.07
2021.10.21

1420万!龙湖现代免疫实验室单克隆细胞筛选系统等仪器设备采购项目
2024.07.29



版权与免责声明:
① 凡本网注明"来源:仪器信息网"的所有作品,版权均属于仪器信息网,未经本网授权不得转载、摘编或利用其它方式使用。已获本网授权的作品,应在授权范围内使用,并注明"来源:仪器信息网"。违者本网将追究相关法律责任。
② 本网凡注明"来源:xxx(非本网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责,且不承担此类作品侵权行为的直接责任及连带责任。如其他媒体、网站或个人从本网下载使用,必须保留本网注明的"稿件来源",并自负版权等法律责任。
③ 如涉及作品内容、版权等问题,请在作品发表之日起两周内与本网联系,否则视为默认仪器信息网有权转载。
![]() 谢谢您的赞赏,您的鼓励是我前进的动力~
谢谢您的赞赏,您的鼓励是我前进的动力~
打赏失败了~
评论成功+4积分
评论成功,积分获取达到限制
![]() 投票成功~
投票成功~
投票失败了~