










近期,浙江大学化学系方群教授团队在国际权威期刊《Nature Communications》上发表了题为“Pick-up single-cell proteomic analysis for quantifying up to 3000 proteins in a Mammalian cell”的研究成果,创新性地提出了PiSPA(Pick-up Single-cell Proteomic Analysis)技术用于单细胞蛋白组分析。该工作流程利用纳升级微流控液滴操控机器人,可以实现单细胞精准捕获、样本前处理以及自动进样,利用布鲁克tims TOF Pro质谱仪在单个哺乳动物细胞内实现了超过3000种蛋白的定量深度。作者通过此项技术对三种不同的哺乳动物细胞(HeLa、A549和U2OS细胞)进行单细胞蛋白质组学研究,以及研究了HeLa细胞在迁移过程中的细胞异质性,均展现了超高的定量分析深度。

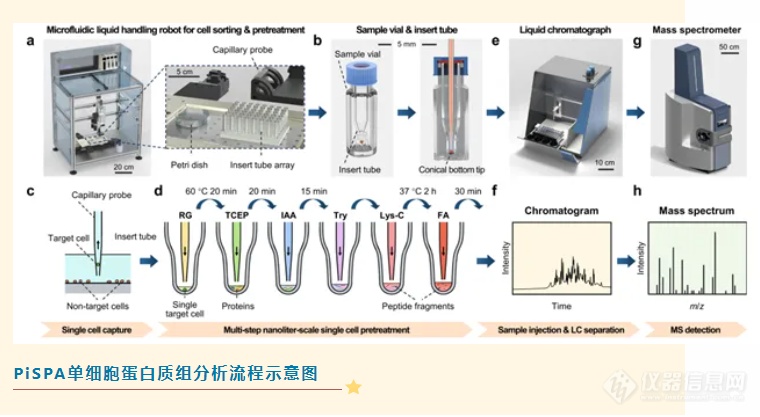
PiSPA平台的单细胞捕获过程是基于SODA(Sequential Operation Droplet Array)技术开发的微流控液体处理系统,可在“点取式”操作模式下实现自动化纳升级单细胞分选,并且能够基于细胞的明场形态特征或是标记的荧光信号灵活地选择单细胞,具有很高的捕获成功率和指向性。此外,研究人员将商品化的锥形底部内插管改造成阵列化的纳升级微反应器,兼容后续的液相色谱自动进样,可大大提高整个工作流程的可操作性、可靠性和成功率。PiSPA平台可自动完成单细胞捕获、样品前处理、色谱分离、质谱检测等一系列操作,最大程度降低样品的损失。
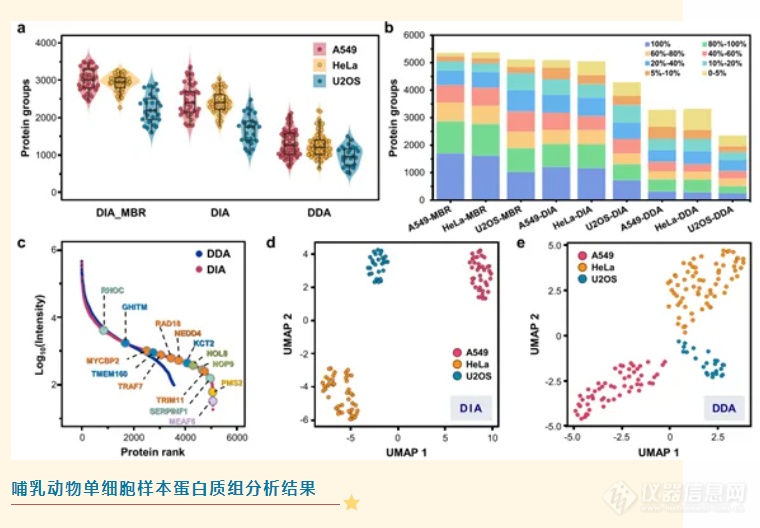
研究人员利用PiSPA工作流程,对多种哺乳动物单细胞实现了深度覆盖的定量分析。采用优化的胰蛋白酶/蛋白比例和色谱梯度,分别通过DDA和DIA扫描模式对A549、HeLa和U2OS三种细胞进行单细胞蛋白组分析,所有质谱数据均使用布鲁克4D蛋白质组学平台——timsTOF Pro进行采集。布鲁克独特的捕集离子淌度技术(TIMS)带了额外一维离子淌度信息,可大大降低样品分析复杂度,极大提高峰容量和分析物鉴定可靠性,最新一代平行累积连续碎裂技术(PASEF®)可以实现极高的二级扫描速度和灵敏度,只需要很少量样本就可以达到组学鉴定新深度。在本研究中,timsTOF Pro更是展示了探索单细胞蛋白质组学的能力,在DIA扫描模式下,利用DIA-NN(MBR算法)进行library-free数据检索,可在A549、HeLa和U2OS三种细胞的单细胞样本中,分别平均定量到3008、2926和2259种蛋白质,展现了PiSPA平台在单细胞蛋白组定量分析中的覆盖深度;此外,有2869、2772和1889种蛋白质在至少80%的A549、HeLa和U2OS单细胞样本中被重复定量到,说明了PiSPA平台有很高的重复性和可靠性。
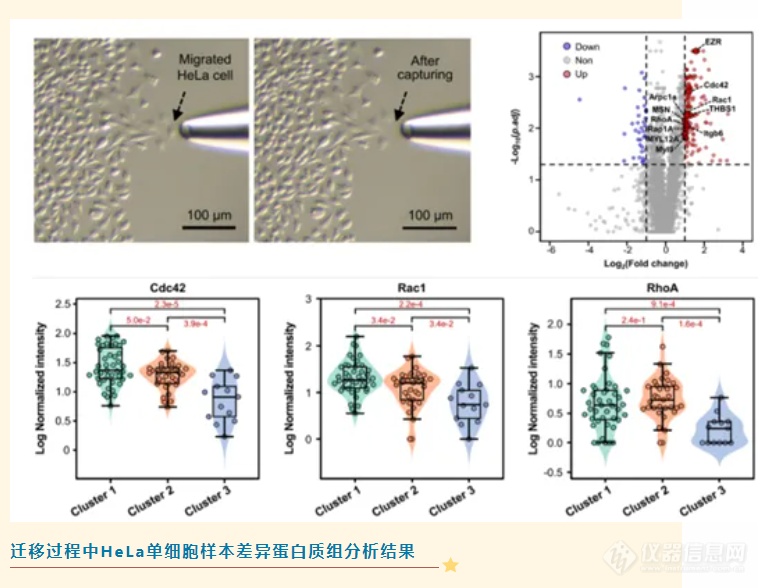
为了证明PiSPA平台的实际应用价值,研究人员分析了迁移过程中HeLa单细胞的蛋白质组表达情况。通过细胞划痕实验,选取有明显迁移(n=46)和未发生迁移(n=43)的HeLa单细胞进行定量分析。采用DIA扫描模式,分别平均鉴定到了2544和2893种蛋白质,后续生物信息学分析发现Cdc42、Rac1和RhoA等蛋白在发生迁移的HeLa细胞中发生显著上调,揭示了迁移过程中的焦点粘附和肌动蛋白骨架调节通路发生激活,说明了PiSPA可以作为细胞迁移研究和抗癌药物靶点研究的有效工具。
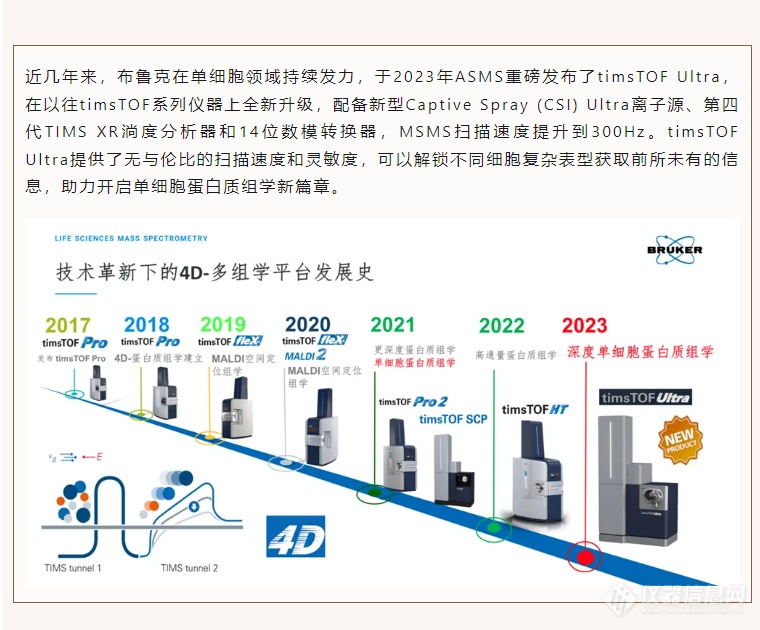
PiSPA工作流程包含了高精度的液体操控、单细胞的精确前处理,结合布鲁克先进的液相色谱-捕集离子淌度谱-四极杆飞行时间质谱仪(LC-TIMS-QTOF MS)进行蛋白质组分析,突破了传统质谱分析在单细胞蛋白组研究领域的技术瓶颈,实现在单个哺乳动物细胞中定量超过3000种蛋白,重新定义了单细胞蛋白质组学分析。这项成果也再次向我们证明了单细胞蛋白质组学在疾病诊断和预防、药物开发、癌症基因组学等精准医学研究中的应用潜力。
1. Wang Y, Guan ZY, Shi SW, et al. Pick-up single-cell proteomic analysis for quantifying up to 3000 proteins in a Mammalian cell. Nat Commun. 2024;15(1):1279.
2. Meier F, Brunner AD, Koch S, et al. Online Parallel Accumulation-Serial Fragmentation (PASEF) with a Novel Trapped Ion Mobility Mass Spectrometer. Mol Cell Proteomics. 2018;17(12):2534-2545.
3. Meier F, Brunner AD, Frank M, et al. diaPASEF: parallel accumulation-serial fragmentation combined with data-independent acquisition. Nat Methods. 2020;17(12):1229-1236.
[来源:布鲁克·道尔顿(Bruker Daltonics)]
2023.09.15

2023.03.13

2023.01.10

2022.12.01
2022.12.01
布鲁克 MALDI HiPLEX-IHC 真正的空间多组学表征解决方案
2022.09.09
版权与免责声明:
① 凡本网注明"来源:仪器信息网"的所有作品,版权均属于仪器信息网,未经本网授权不得转载、摘编或利用其它方式使用。已获本网授权的作品,应在授权范围内使用,并注明"来源:仪器信息网"。违者本网将追究相关法律责任。
② 本网凡注明"来源:xxx(非本网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责,且不承担此类作品侵权行为的直接责任及连带责任。如其他媒体、网站或个人从本网下载使用,必须保留本网注明的"稿件来源",并自负版权等法律责任。
③ 如涉及作品内容、版权等问题,请在作品发表之日起两周内与本网联系,否则视为默认仪器信息网有权转载。
![]() 谢谢您的赞赏,您的鼓励是我前进的动力~
谢谢您的赞赏,您的鼓励是我前进的动力~
打赏失败了~
评论成功+4积分
评论成功,积分获取达到限制
![]() 投票成功~
投票成功~
投票失败了~