从人类基因组草图到完全图谱 ——论基因组重复片段研究
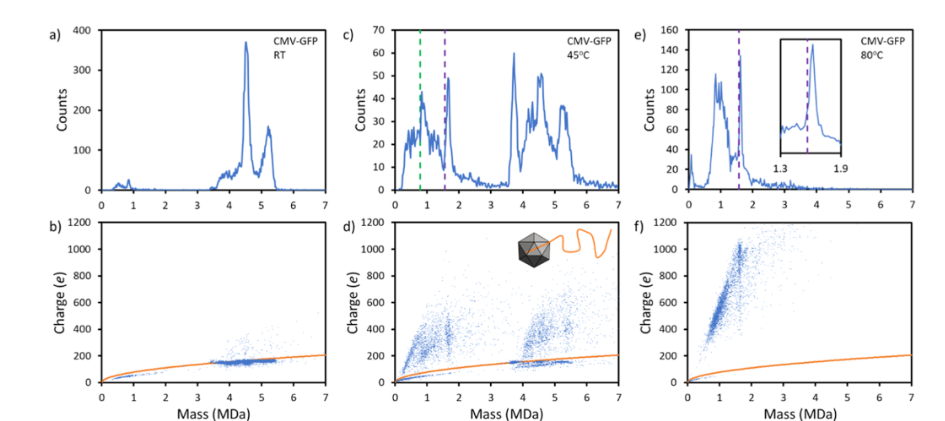
从人类基因组草图到完全图谱——论基因组重复片段研究作者:李东卫,张玉波(中国农业科学院农业基因组研究所,“岭南现代农业”广东省实验室,深圳 518120)2001年发表的人类基因组草图并没有包含全部的基因组序列,直到二十年后,科学家们才正式宣布完成了人类全序列基因组图谱,这其中主要的技术障碍就是重复片段的测序工作。重复片段(segmental duplications,SDs)是指广泛存在于基因组中的大于1 kb且序列相似性超过90%以上的大片段。它们可以通过基因组重排及拷贝数变异产生新基因和驱动进化,其大量存在于子端粒中,并与哺乳动物细胞复制性衰老以及癌症等重要生物学过程密切相关,一直以来备受科学家关注。但是其序列特点使得常规的测序技术难以完全准确测出全部序列,是基因组组装工作的一个难点。人类基因组全图谱的完成将重复片段在生物体进化、延缓衰老、疾病治疗等方面的研究提供基础。本文将就重复片段的重要性,研究的技术难点,研究现状以及未来展望等方面展开论述。重复片段的重要性重复片段是基因组中序列高度相同的大片段,具有广泛的结构多样性。它们占人类参考基因组(T2T-CHM13)中的7.0%,长度为218 Mbp[2 ],在中心体及子端粒区域富集高达10倍。中心体所包含的5个典型重复为:α卫星,β卫星,CER卫星,γ卫星,CAGGG重复,以及重复子4。子端粒所包含的典型重复为:端粒相关重复(TAR)以及传统的(TTAGGG)n重复[4 ]。重复片段可以介导染色体重排,使常染色体和异染色体之间通过同源重组产生镶嵌类型的重复的染色质[5 ]。在最近新鉴定的人类重复片段中,Mitchell R等预测了182个新的候选蛋白编码基因,并使用T2T-CHM13基因组重构了重复基因(TBC1D3,SRGAP2C,ARHGAP11B),这些基因在人额皮质增生中具有重要作用,揭示了重复片段结构在人和他们近亲物种之间的巨大进化差异[6 ]。大量的染色体子端粒区含有重复片段[8 ]。复制性衰老被认为是一种抗癌机制,限制细胞增殖。长寿的有机体经历更多的细胞分裂,因此具有更高的产生肿瘤的风险。端粒酶能够增加端粒的长度,促进癌细胞不断增殖,因此长寿动物体细胞倾向于抑制端粒酶的活性,从而抑制肿瘤发生的风险[10 ]研究难点:大片段长度、多拷贝数、序列高度相似 重复片段的大的片段长度,多拷贝数以及序列的高度相似是长期以来其研究的难点。各种测序技术的发展致力于解决这个问题。重复片段长度范围是1到400 kb [12 ]。而且,标准的长读段校正工具,例如MUMmer 或Minimap2不能够有效的捕捉低相似的重复片段,也经常将重复片段与其它调控元件混淆[14 ],为重复片段的研究带来机遇。尤其是PacBio的HiFi读段,具有长读段的同时还具有较高的准确度。但是,很多重复片段的长度要比HiFi读段的平均长度要长,因此很难完全准确的进行组装[3 ]。染色体重排,尤其是染色质断裂常发生在高GC区域[16 ]。同时,在T2T-CHM13基因组基础上,Mitchell R等首次进行了全基因组重复片段的研究。与当前人类参考基因组(GRCh38)鉴定的167 Mbp复制片段相比,鉴定了更多的(218 Mbp)非冗余重复片段(图2 a, b)。新发现91%的重复片段能更好地代表人的拷贝数,通过与非人灵长类基因组相比,前所未有的揭示了人类和其它近亲在重复片段结构中的杂合性以及广泛的进化差异[17 ]。图2 T2T-CHM13中新鉴定的染色体内(a)与染色间(b)的重复片段[1 ]。利用重复片段解析衰老机制未来可期新组装的T2T-CHM13的拷贝数比GRCh38高9倍,因此它能更好的呈现人类拷贝数变异。通过鉴定新基因的拷贝数变异,可筛选相应的药物治疗靶点。例如,CHM13鉴定到LPA、MUC3A、FCGR2基因的拷贝数变异与疾病相关[1]。此外,对于尚具争议的疾病标志基因,例如乳腺癌中ESR1 基因[18],可以通过CHM13对其进行分子进化分析,进而鉴定其突变和扩增,确定其在乳腺癌中的作用。尽管端粒作为抗衰老靶标已研究多年,但是端粒长短变化与复制性衰老的关系仍不清楚。细胞减数分裂过程中端粒变短的机制是什么?重复片段拷贝数变异与端粒变短有无相关性?很多研究已证明端粒酶具有延长端粒长度的作用,具体的机制是什么?这些问题因此前端粒不能被准确测序而长期未解决。现在,人类基因组完全图谱已基本实现,相信这些谜团会很快解开。未来可以根据人类年龄增长过程中端粒重复片段的拷贝数变异,解析其抗衰老的机制。通过人为干预其拷贝数,可能用于探索生命的极限。1. Vollger MR, Guitart X, Dishuck PC, Mercuri L, Harvey WT, Gershman A, Diekhans M, Sulovari A, Munson KM, Lewis AM et al.Segmental duplications and their variation in a complete human genome. bioRxiv.2021:2021.2005.2026.445678.2. Prodanov T, Bansal V.Sensitive alignment using paralogous sequence variants improves long-read mapping and variant calling in segmental duplications. Nucleic Acids Research.2020 48(19).3. Bailey JA, Yavor AM, Massa HF, Trask BJ, Eichler EE.Segmental duplications: Organization and impact within the current Human Genome Project assembly. Genome research.2001 11(6):1005-1017.4. Courseaux A, Richard F, Grosgeorge J, Ortola C, Viale A, Turc-Carel C, Dutrillaux B, Gaudray P, Nahon JL.Segmental duplications in euchromatic regions of human chromosome 5: a source of evolutionary instability and transcriptional innovation. Genome research.2003 13(3):369-381.5. Giannuzzi G, Pazienza M, Huddleston J, Antonacci F, Malig M, Vives L, Eichler EE, Ventura M.Hominoid fission of chromosome 14/15 and the role of segmental duplications. Genome research.2013 23(11):1763-1773.6. Young E, Abid HZ, Kwok PY, Riethman H, Xiao M.Comprehensive Analysis of Human Subtelomeres by Whole Genome Mapping. PLoS genetics.2020 16(1):e1008347.7. Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, Devon K, Dewar K, Doyle M, FitzHugh W et al.Initial sequencing and analysis of the human genome. Nature.2001 409(6822):860-921.8. Seluanov A, Chen ZX, Hine C, Sasahara THC, Ribeiro AACM, Catania KC, Presgraves DC, Gorbunova V.Telomerase activity coevolves with body mass not lifespan. Aging Cell.2007 6(1):45-52.9. Bromham L.The genome as a life-history character: why rate of molecular evolution varies between mammal species. Philos T R Soc B.2011 366(1577):2503-2513.10. Shay JW.Role of Telomeres and Telomerase in Aging and Cancer. Cancer discovery.2016 6(6):584-593.11. Sharp AJ, Locke DP, McGrath SD, Cheng Z, Bailey JA, Vallente RU, Pertz LM, Clark RA, Schwartz S, Segraves R et al.Segmental duplications and copy-number variation in the human genome. American journal of human genetics.2005 77(1):78-88.12. Hartasanchez DA, Braso-Vives M, Heredia-Genestar JM, Pybus M, Navarro A.Effect of Collapsed Duplications on Diversity Estimates: What to Expect. Genome Biol Evol.2018 10(11):2899-2905.13. Numanagic I, Gokkaya AS, Zhang L, Berger B, Alkan C, Hach F.Fast characterization of segmental duplications in genome assemblies. Bioinformatics.2018 34(17):i706-i714.14. Vollger MR, Dishuck PC, Sorensen M, Welch AE, Dang V, Dougherty ML, Graves-Lindsay TA, Wilson RK, Chaisson MJP, Eichler EE.Long-read sequence and assembly of segmental duplications. Nature methods.2019 16(1):88-94.15. Rhie A, McCarthy SA, Fedrigo O, Damas J, Formenti G, Koren S, Uliano-Silva M, Chow W, Fungtammasan A, Kim J et al.Towards complete and error-free genome assemblies of all vertebrate species. Nature.2021 592(7856):737-+.16. Nurk S, Koren S, Rhie A, Rautiainen M, Bzikadze AV, Mikheenko A, Vollger MR, Altemose N, Uralsky L, Gershman A et al.The complete sequence of a human genome. bioRxiv.2021:2021.2005.2026.445798.17. Zhu Y, Liu X, Ding X, Wang F, Geng X.Telomere and its role in the aging pathways: telomere shortening, cell senescence and mitochondria dysfunction. Biogerontology.2019 20(1):1-16.18. Tabarestani S, Motallebi M, Akbari ME.Are Estrogen Receptor Genomic Aberrations Predictive of Hormone Therapy Response in Breast Cancer? Iranian journal of cancer prevention.2016 9(4):e6565.