药物基因组学是上世纪九十年代末发展起来,基于药理学和基因组学,将传统的药物科学与基因、蛋白、单核苷酸多态性等知识结合起来的一门科学。正因为药物基因组学是研究基因序列变异及其对药物不同反应的科学,所以它是研究高效、特效药物的重要途径,通过它为患者或者特定人群寻找合适的药物,药物基因组学强调个体化;因人制宜,有重要的理论意义和广阔的应用前景。一、促进新药研发 由于药物基因组学规模大、手段强、系统性强,开辟了医药工业研究的新领域,可以直接加速新药的发现。首先药品制造商不仅把注意力放在可能引起疾病的基因上,而且对药物效应基因产生了兴趣,这些药物效应基因为新药研究提供依据。由于新一代遗传标记物的大规模发现,以及将其迅速应用于群体,流行病遗传学也可以大大推进多基因遗传病和常见病机理的基础研究。还可以帮助制药厂商在一些与基因和疾病相关的蛋白质、酶和RNA分子等基础上开发新药,这样不仅促进了药物的发现,还有利于开发出针对某一特定疾病的药物,从而增强疗效,并减少对健康细胞的损伤。对于每一个药物来说大约都有10-40%的人没有疗效,又百分之几的或更多的人有副作用。如果制药公司利用药物基因组学理论可以实现预见结果或筛选人群的话,可以大大增加新药的通过率,也可以对未通过药检的新药重新估价,这些药物中一个经常引用的例子是第一个非典型性抗精神活性药氯氮平(clozapine),在氯氮平的使用过程中,由于1%的病人服药后出现严重的粒细胞缺乏症,因而只有当其它药物使用后无效才使用。但是在粒细胞缺乏症的药物效应基因被确定后,极大地改善了氯氮平的使用,除极少数敏感的病人不能服用此药外,对于99%的病人来说,这一药物是一线治疗药物。在新药的临床试验研究中,如果事先知道人群可能对药物反应的话,如代谢酶的基因型,可以减少参试人群,试验的时间表也可以大大缩短。对药物有效或毒性变异的预测试验中,可用于筛选病人。经过药物效应基因突变筛选的受试者,可以加强临床试验的统计学意义,可以用更少的病例数达到所需的统计学意义,这样可以大大节约时间和费用。 二、用药个体化合理用药的核心是个体化用药。药物基因组学通过对患者的基因检测,如对一些疾病相关基因的单核苷酸多态性(SNP)检测,进而对特定药物具敏感性或抵抗性的患病人群的SNP差异检测,指导临床开出适合每个个体的“基因处方”,使患者既能获得最佳治疗效果,又能避免药物不良反应,真正达到“用药个体化”的目的。 医生在疾病的首次治疗过程中,往往需要临床实验来确定适合病人的药物,而药物基因组学则可以通过分析病人的遗传组成来确定最合理的治疗药物。这样就免去了先期用于药物选择的临床过程及由此带来的可能的副作用,并缩短了病热的康复期。更准确的用药剂量 通过基因组分析可以判断药物在体内的作用效果及代谢时间,并以此来确定不同个体的用药剂量,对比依据体重和年龄的方法,其具有更好的治疗效果,降低了过量服药的可能性。一些临床上经常出现的现象,例如两患者诊断相同、一般症状相同、血药浓度相同,但疗效却大相径庭,这些用传统的药代动力学原理是无法解释的。这时应考虑到与药物作用相关的位点(如受体等)是否发生了变异?是什么水平的变异?药物作用的位点的变异可能发生在基因水平,也可能发生在转录、翻译等水平,基因水平的变异相对比较容易鉴定,研究也表明基因的变异与药物效应的差异是更具相关性。研究基因突变与药效关系的药物基因组学正是适应了这一要求,因此药物基因组学在临床合理用药中的应用前景是非常之好的。将基因功能学用于合理用药,利用药物基因组学的技术和方法增加药物的有效性和安全性,减少不良反应,实现个体化、可预测及可预防的医疗,这就称之为临床药物基因组学。药物基因组学应用到合理用药中,弥补了只根据血药浓度进行个体化给药的不足,惟以前无法解释的药效学现象找到了答案,为临床个体化给药开辟了一个新的途径。这样药物基因组学原理为特定人群设计最为有效的药物,不仅提高了药效,缩短了病程,而且减少了毒副反应和成本,真正达到了“物美价廉”的要求。目前,已经有人将药物基因组学知识应用于高血压、哮喘、高血脂、内分泌、肿瘤等的药物治疗中。如原发性高血压是多因素诱发的疾病,对于许多患者,高血压药物的不同药效和耐受性与遗传变异有关。Ferrari发现,一种细胞骨骼蛋白(cytoskeletalprotein)、内收蛋白(adducin)的基因多态性与高血压的发病、对钠敏感性以及对利尿剂的效果相关。因此在抗高血压治疗需要用利尿剂时,可以对患者预先进行基因检测,以确定是否选择使用此药。通过对β2肾上腺素受体的基因多态性及其对β2肾上腺素受体激动剂的敏感性关系的研究,发现β2肾上腺素受体的基因多态性影响β2肾上腺素受体激动剂福莫特罗(formoterol)的脱敏效果,β2肾上腺素受体激动剂改善肺通气的作用对Gly纯合子个体明显比Arg纯合子个体要强,杂合子个体介于两者之间。 载脂蛋白E(APOE)的基因多态性,影响绝经后妇女用雌激素替代疗法(ERT)时的血脂和脂蛋白的浓度。人群中的APOE有3个等位基因:E2、E3、E4,ERT能使具有E2型基因的妇女血中总胆固醇含量大大高于E3、E4型。提示医生在绝经期妇女中使用ERT时,可事先检测患者的APOE基因,对具有E2型基因的妇女在治疗过程中密切监测甘油三酯浓度。如此,通过对不同个体的药物代谢相关酶、转运因子、药物作用靶点的基因多态性的研究,对突变的等位基因进行分离和克隆,在分子诊断水平上建立以聚合酶链反应(PCR)为基础的基因型分析方法,在治疗患者各种疾病前检测其基因型,更精确地选择适当的治疗药物和合适的剂量以减少不良反应的发生,对患者的治疗具有很大的意义。 随着基因分析技术的飞速发展,越来越多的药物效应的个体差异与基因多态性的关系被阐明,药物基因组学将更广泛地指导和优化临床用药。
“10年前,我们参与人类基因组计划,完成了1%的工作,其实是‘搭了别人的车’。现在,面对即将到来的基因组学新时代,我们不能再搭别人的车了。”当年曾参与人类基因组计划承接1%测序工作、如今已是中科院北京基因组研究所副所长的[url=http://sourcedb.big.cas.cn/zw/zjrc/brjh/200907/t20090724_2194384.html][color=#800000]于军[/color][/url],对中国未来基因组学的发展和应用前途有点担心。2010年6月26日是人类基因组图谱公布10周年,国内外的一些研究机构都在这一天举行了纪念会。中国科协普及部、中科院北京基因组研究所、遗传与发育学研究所、中国遗传学会等单位也在北京举行了纪念会。会上,于军兴奋地回忆起当年他那个义无反顾的决定:1998年4月一天的早晨4点左右,他正在美国西雅图的家中睡觉。忽然自动传真机响了起来,“我爬起来一看,是邀请我回国工作的,我拿起笔签上自己的名字就传了回去”。这是于军回国工作的起点,也是中国参加人类基因组计划的起点。正是于军带回国内的技术和人才奠定了完成1%任务的基础。他是“1%计划”的“始作俑者”之一。与那时的热血沸腾相比,今天的于军更多了一份冷静与思考。“10年前,测定一个人的基因组,大约花了近10亿美元,用了13年的时间;而现在测一个人的基因组也就1万美元、一周左右的时间。美国现在已研制出第三代基因测序仪,用它测定一个人基因组的费用可降到1000甚至100美元,用时仅需15分钟。”于军说,当第三代基因测序仪广泛应用时,大规模应用基因组技术的“个体化基因组时代”就到来了。“个体化基因组时代”为人类描绘了一个美好的未来:那时,我们可以知道某一种药物为什么会对一部分人有治疗作用而对另一部分人不起作用,甚至起负作用;那时还会针对个体疾病的状态和遗传基础的独特性对症下药;也会针对个体化的药靶研制出个性化的治疗药物和治疗手段……这将是一个巨大的医疗市场。而测定每一个人的基因组本身也是一个大市场。“对于有十几亿人口的中国来说,假如使用美国研制的第三代基因测序仪来工作的话,那要进口多少台?按一个人测序需100美元计算,又要花费多少钱?”于军向记者言及此事,表现出了一种内心深处的忧虑。“中国一定要加快研制自己的DNA测序仪。”据了解,于军团队正与有关单位合作研制第二代和第三代测序仪器。“但我们的力量仍然有限,应该有更多的团队和单位加入到这个行列中来。这是我们迎接基因组学新时代的必要准备。”还有一种必要的准备,那就是做好有关基因、遗传学、基因组学等相关科学的科普宣传工作。“美国人十分重视基因组学的科普宣传。”于军回忆说,当年,美国立项测定人类基因组图谱时,就把这项工作的科普宣传列入了计划。10年前图谱完成时,时任美国总统克林顿发表致辞,电台、电视台现场直播,上万名美国人参加了当时各种各样的庆祝活动。“关于科普的作用,一个明显的例子是对待转基因食品的态度。”于军说,美国人就不像中国人那样对转基因食品“过分惶恐”。因为他们知道转基因食品并不像有人说的那样可怕和有危害。中国在面对转基因食品的问题上,好像是由大众的好恶来决定,而不是由对转基因的科学认识来决定。未来的基因组学时代、个性化基因组时代,我们可能会遇到比转基因食品更棘手的问题:法律问题、伦理道德问题、个人隐私问题等等。“从现在起我们就应该做好基因组新时代的科普宣传,未雨绸缪,为基因组学的发展和应用提供更加广阔的发展空间。”于军如是说。(转自科技日报)

http://ng1.17img.cn/bbsfiles/images/2014/09/201409091606_513258_271_3.png8月20-22日,由中国遗传学会植物遗传与基因组学专业委员会主办、安徽农业大学承办的“第十五届全国植物基因组学大会”在合肥隆重召开。为期三天的大会有来自北京大学、浙江大学、复旦大学、中国科技大学、中国农业大学、华中农业大学、南京农业大学、西北农林科技大学等45所高校,以及中国科学院、中国农业科学研究院等44个研究所的460余名专家学者出席。大会还从美国、法国、西班牙和香港邀请5位专家出席。http://ng1.17img.cn/bbsfiles/images/2014/09/201409091605_513256_271_3.jpg上图为会议现场照片RAININ作为此次活动的参与者,在现场进行了产品宣讲及样机展示。通过品牌推广,收集到了众多潜在客户的购买需求,现场产生了很多Liquidator96工作站的线索和机遇。通过与其他参会者的充分沟通,把握到了竞争对手的最新动态,也寻找到了可以长期合作的业务伙伴。RAININ Liquidator™ 96 手动移液系统,可以一次性为 96 个孔进行移液简化了微量应用的工作,如 ELISA、细胞分析、qPCR 和蛋白结晶等,具有较高的准确度和精度,简化了任何 96 或 384 孔板应用的工作。它体积小巧,无需用电,可在两个量程范围内使用 - 0.5 至 20μL 和 5 至 200μL。http://ng1.17img.cn/bbsfiles/images/2014/09/201409091606_513257_271_3.jpg上图为RAININ展位,支持人员:上海办销售文振
霍华休斯医学研究所,Baylor医学研究所的科学家们近期在PloS One上发表最新研究性文章,文章标题为:Big Genomes Facilitate the Comparative Identification of Regulatory Elements,该文章解析了基因组大小对基因组学的研究带来的影响。基因组越大则更容易找出控制基因活性的DNA区域。在小基因组上,功能性元件紧紧地结合在一起。而在大基因组上,功能性元件分得比较散,于是也更容易找到控制基因活性的区域。 基因组分为结构基因和调控基因,要从基因组上找到功能元件并不难,难的是找到调控基因表达的机制,因此,对小的基因组来说,紧凑的结构给寻找调控区域带领更多的困难,而相对来说大基因组却容易多了。功能元件散落在基因组上,更便于寻找调控区域。大的基因组更便于研究非编码DNA和RNA,对研究基因调控也更为有利。而目前,研究生命的遗传物质DNA的科学家一直觉得,基因组越小越受欢迎,因为操作简单,可以节省大量的时间和精力,尤其在金钱方面也能更节约成本,测序的费用更低。甚至有科学家说,基因组小则基因排列更紧凑,垃圾DNA也越少。 [img]http://www.instrument.com.cn/bbs/images/affix.gif[/img][url=http://www.instrument.com.cn/bbs/download.asp?ID=137848]Big Genomes Facilitate the Comparative Identification of Regulatory Elements[/url]
国家“863计划”现代农业技术领域在主要动植物功能基因组研究方面,利用“十五”建立的水稻功能基因组的技术平台,系统开展水稻产量、品质、抗病抗逆、营养高效性状的功能基因组研究,克隆验证新基因和调控因子,应用芯片技术建立水稻重要农艺性状的全基因组表达谱,并开展比较基因组学研究和第3、4染色体功能基因的系统鉴定。 利用水稻、拟南芥等模式植物功能基因组的技术平台,开展小麦、玉米、棉花、油菜、大豆、花生、番茄等作物的功能基因组研究,克隆验证重要农艺性状基因;建立家蚕和家鸡的功能基因组研究技术平台,分离克隆与家蚕丝蛋白质合成、性别决定、发育变态、分子免疫和对微生物抵抗性、鸡的生长、品质、抗性、繁殖等重要经济性状相关的重要功能基因和调控因子。
大熊猫基因组测序研究项目近日正式完成,并绘制出大熊猫基因组精细图。这是中国科学家第一次全面系统地对大熊猫基因组进行测序研究。 据介绍,大熊猫基因组测序研究结果表明,大熊猫有染色体21对,基因组大小2.4G,重复序列含量36%,基因2万多个。 这项研究由深圳华大基因研究院领衔,中国科学院昆明动物研究所、中国科学院动物研究所、成都大熊猫繁育研究基地和中国保护大熊猫研究中心共同参与。 研究结果还表明,大熊猫基因组仍然具备很高的杂合率和较高的遗传多态性;在已经进行全基因组测序的物种中,大熊猫基因组与狗的基因组最接近;数据分析结果同时还进一步支持了大多数科学家所持的“大熊猫是熊科的一个亚种”这种观点,证明了熊科内部各类群的分类情况。 据悉,大熊猫基因组精细图这一研究成果,填补了大熊猫基因组及分子生物学研究的空白,将从基因组学的层面上为大熊猫的保护、疾病监控及其人工繁殖提供科学依据。
近日,深圳华大基因研究院宣布,我国科学家将参与全球最大微生物基因组研究项目,对来自全球的20万个样本进行环境DNA测序或宏基因组测序,从而建立一个全球性的基因图谱,并承担核心工作。该项目旨在全方位、系统性研究全球范围内微生物群落功能及进化多样性,以便更好地造福社会及人类。与以往的微生物研究有所不同,该项目的研究对象不仅集中于海洋和人体环境中微生物群落,还包括土壤、空气、淡水生态系统等整个地球表面的绝大多数的微生物群落。华大基因将负责亚洲地区所有样本的收集和鉴定,并对整个项目提供DNA提取、扩增、建库、宏基因组测序以及研发生物信息学分析流程所需的计算资源。这些信息学分析流程将为项目研究产生的海量数据提供一个分析框架。项目负责人、芝加哥大学和阿贡国家实验室的教授杰克·吉尔伯特博士表示:“华大基因在测序能力、测序技术和信息分析等方面已展现出卓越的能力。此项目是一个前所未有的最大的基因组测序项目,作为全球最大基因组学研究中心,华大基因的参与至关重要。”华大基因理事长杨焕明院士表示,微生物对地球上所有的生命具有至关重要的作用,而我们对微生物的复杂性和多样性认识不足,征服这个未知的领域非常有必要。华大基因拥有国际先进水平的测序平台和强大的生物信息学分析能力,可以为促进人类对微生物群落重要性的了解贡献力量。(来源:科技日报)
由来自中国、美国、荷兰、以色列等14个国家的300多位科学家组成的“番茄基因组研究国际协作组”,历时8年多的艰苦努力,于近日完成了对栽培番茄全基因组的精细序列分析。今天,国际权威学术期刊《自然》以封面文章发表了这项重大科学成果。 番茄是研究果实发育的经典模式植物,我国科学家在这项国际番茄基因组研究中作出了重要贡献。作为中方协调人,中科院遗传与发育生物学研究所研究员李传友和薛勇彪负责第3号染色体的测序工作,中国农科院蔬菜花卉研究所研究员黄三文和杜永臣负责第11号染色体的测序工作。番茄基因组有12条染色体,中国科学家高质量地完成了番茄基因组测序总任务的1/6,标志着我国成为番茄基因组学研究的强国之一。 8年来,国际协作组采用“克隆连克隆”和“全基因组鸟枪法”相结合的测序策略,在解码的番茄基因组中,共鉴定出约34727个基因,其中97.4% (33840个)的基因已经精确定位到染色体上。番茄基因组的解读,是科学家通过国际合作完成的又一个高质量的模式植物的基因组序列分析,对于不同物种之间的比较基因组学研究具有重要价值,这项工作将极大推动番茄乃至包括马铃薯、辣椒、茄子等在内的茄科植物的功能基因组研究,为培育具有高产、优质、抗病虫害、抗逆等优良性状的番茄新品种打下了良好的基础,对推动全世界的番茄生产具有重要意义。 有关专家表示,我国蔬菜种业面临着强大的国际竞争。中国在国际蔬菜基因组研究领域具有优势地位,而如何把基础科研的优势转化为产业优势,是目前面临的主要挑战。科学家建议,应在进一步巩固蔬菜基因组研究优势的基础上,加强蔬菜作物分子设计育种体系的建设,并与常规育种相结合,加速有自主知识产权优良品种的培育,这对于支撑我国蔬菜产业可持续发展、提升我国蔬菜种业的国际竞争力具有重要意义,也是不可错过的历史机遇。
古老病毒通过入侵重塑人类基因组译者:Docofsoul《每日科学》2010年9月13日报道 —— 新加坡基因组研究院(GIS, 隶属于新加坡科技研究局(A*STAR)的生物医学研究院)的科学家以及来自新加坡国立大学、新加坡南洋理工大学、杜克-新加坡大学医学研究院与普林斯顿大学的同事们最近发现:数百万年前“入侵”人类基因组的病毒已经改变了人类胚胎干细胞(ES细胞)中的基因开启与关闭方式。科学家已经发现数百万年前“入侵”人类基因组的病毒已经改变了人类胚胎干细胞(ES细胞)中的基因开启与关闭方式。(照片来源:iStockphoto/Martin McCarthy)这一研究为生理学与医学诺贝尔奖获得者芭芭拉•麦克林托克(Barbara McClintock)于上世纪五十年代提出的理论提供了明确的证据。芭芭拉•麦克林托克的理论推测:转座因子,即可移动的遗传物质(DNA)片段(比如说病毒序列),一旦插入基因组,就能成为影响基因调节的“控制因子”。本发现对于推进干细胞研究进程、增强干细胞研究为再生医学效劳的潜力都算得上是重要贡献。 由新加坡基因组研究院精英小组负责人吉拉姆•布尔克(Guillaume Bourque)博士率队领导了本研究。本研究的论文发表于2010年6月6日的《Nature Genetics》(《自然•遗传学》)。通过运用新的测序技术,科学家们研究了人类与小鼠胚胎干细胞(ES细胞)中三种调节蛋白质(OCT4、NANOG 与 CTCF) 的染色体组定位(基因组定位)。令人感兴趣的是,在科学家发现大量的相似点的同时,他们也发现了在人类中受到调控的基因方式与基因类型的许多不同点。尤其是,他们发现:数百万年前自行插入人类基因组的特定类型病毒已经戏剧性地改变了人类干细胞基因调控网络。德克萨斯州大学阿灵顿分校副教授Cedric Feschotte 博士说:“本研究是计算与实验双管齐下的代表作,提供了无可置疑的全新的证据:一些经常被斥责为纯粹垃圾DNA的转座因子,恰恰正是人类发育调控密码的关键成分。”在基因调控网络的研究中,人类模型系统与小鼠模型系统之间的比较研究有助于增进对干细胞分化成体内不同细胞类型的具体过程的理解。布尔克博士说:“这种理解在促使再生医学的百尺竿头更进一步地发展 —— 从而解决诸如帕金森病与白血病等问题方面是至关重要的。除了在本研究中利用基因调控网络中的小鼠胚胎干细胞的优势外, 深入研究必须更加直接地集中于人类干细胞。这是因为将某一种类上完成的研究成果转向对另一种类的研究上时必然会遇上的挑战。为了让干细胞方面的发现能够用于临床实践,在人类与(非人类的)灵长类干细胞两个方面还有更多的研究工作需要完成。” 加利福尼亚州立大学神经学Rudi Schmid 特聘教授、哲学博士雷蒙德•怀特(Raymond L. White)教授说:“本论文报告了令人非常激动的新发现,证实了一个全新的、迥然不同的基因表达的调控机制。通过将小鼠的基因组与人类基因组的直接比较,科学家能够显示:在两种种类之间,基因调控因子的结合点经常不在同一位置。这本身就足够令人惊讶的了,但是研究者作了进一步的探索,证实许多位点都嵌合在称之为‘转位’因子的一类DNA序列中,这是因为他们具有在基因组中移动到新的位置的能力。存在很多这样的相信是病毒基因组进化残余部分的因子,但我们所了解到的(信息中)还有着非常出人意外的情形:它们到达新的(基因组)位置时,还携带着调控因子结合位点。这些在调控方面的变化估计可能在携带它们的有机体上产生重大变化。确实,许多学者相信调控方面的变化处于物种形成的核心,可能在人从其祖先的进化历程中扮演了一个重要角色。本论文可能成为这一研究领域的里程碑式的论文。”美国能源部联合基因组研究所所长、劳伦期•伯克利国家实验室伯克利实验分室基因组学部主任埃迪•拉宾(Eddy Rubin)博士补充说:“这个运用了比较基因组学策略的研究在人类胚胎干细胞(ES细胞)中发现了重要的人类特异性属性。该论文所提供的信息意义重大,应该有助于推进再生医学领域的发展,相信会有不俗的积极表现。”参考文献:Galih Kunarso, Na-Yu Chia, Justin Jeyakani, Catalina Hwang, Xinyi Lu, Yun-Shen Chan, Huck-Hui Ng, Guillaume Bourque. Transposable elements have rewired the core regulatory network of human embryonic stem cells.Nature Genetics, 2010; 42 (7): 631 DOI: 10.1038/ng.600(《转位因子重新连接人类胚胎干细胞的核心调控网络》)
http://img.dxycdn.com/trademd/upload/userfiles/image/2013/01/B1357710940_small.jpg梅花因其独特的花香,在很多诗词中成为人们吟诵的对象。那么,它的花香到底来自何处呢?我国科学家从基因组水平,揭示了合成梅花花香中重要成分乙酸苯甲酯的BEAT基因家族34个成员,并构建完成了首张梅花全基因组精细图谱。其研究论文在2012年12月27日《自然—通讯》亮点论文在线发表。我国梅花基因组项目首席专家、北京林业大学教授张启翔率领项目组,选取位于梅花起源中心的西藏野生梅花进行基因组测序,从基因组水平,揭示了合成梅花花香中重要成分乙酸苯甲酯的BEAT基因家族34个成员,在梅花基因组中显著扩增并且其中12个成员串联重复分布,从而使梅花具有独特的花香;推测梅花基因组中6个串联重复的DAM基因和其上游过多的CBF结合位点是梅花提早解除休眠的关键因子,从而解释“踏雪寻梅”之说。张启翔告诉记者,梅花全基因组测序的完成以及高密度遗传图谱构建,有助于揭示梅花花期早、花香独特等重要观赏性状的遗传基础,有助于挖掘与诸多重要性状相关的功能基因,为今后进一步揭示梅花花期、抗病调控机制、梅花及相关种属的分子育种奠定基础。研究中,项目组还揭示了蔷薇科植物进化规律。张启翔说,通过分析梅花的进化发现,梅与苹果发生分化后,并没有出现近期的全基因组复制事件,同时结合已完成的苹果和草莓基因组序列,成功重建了蔷薇科9条原始染色体,揭示了蔷薇科植物进化规律,为开展蔷薇科物种比较基因组学研究奠定重要的理论基础。据介绍,该科研成果由北京林业大学、深圳华大基因研究院及北京林福科源花卉有限公司等多家单位合作完成。目前,转录组数据组装及基因功能注释数据已在相关网站对外公开。
基因组测序和序列的组装,为快速研究该致病菌株的致病机理创造了条件。与此同时华大基因与德国汉堡-Eppendorf医疗中心合作,也宣布完成了对致病菌株的测序工作。Guenther说:"在有限的时间里完成了对微生物的全基因组测序,极大的方便了研究者从一个整体的水平上去研究微生物,进而揭示在这些目标微生物的基因组究竟发生了哪些改变。"事实上也的确如此,科学家根据从基因组测序的数据所获得的证据,将本次的致病型大肠杆菌鉴定为致病型大肠杆菌的一个新杂交品种,并且携带了一些抗性基因。"从宏观的基因组水平上来研究这类细菌,将在很大程度上革新我们对传染病暴发的认识,3-4天内完成对某种微生物的全基因组测序及基因标注,将会开启一个新的研究领域。"在新奥尔良召开的美国微生物学会年度会议上,一些研究者指出,分子鉴定的方法正被用来打造基因组传染病学这一领域,基因组传染病学致力于重构传染病暴发的过程,以求在将来能够对传染病能进行实时有效的监控和快速反应。
欢迎大家一起交流讨论~代谢组学是继基因组学和蛋白质组学之后新近发展起来的一门学科,是系统生物学的重要组成部分。基因组学和蛋白质组学分别从和蛋白质层面探寻生命的活动,而实际上细胞内许多生命活动是与代谢物相关的,如细胞信号(cell signaling),能量传递等都是受代谢物调控的。代谢组学正是研究代谢组(metabolome)——在某一时刻细胞内所有代谢物的集合——的一门学科。基因与蛋白质的表达紧密相连,而代谢物则更多地反映了细胞所处的环境,这又与细胞的营养状态,药物和环境污染物的作用,以及其它外界因素的影响密切相关。因此有人认为,基因组学和蛋白质组学能够说明可能发生的事件,而代谢组学则反映确实已经发生了的事情。新陈代谢网络是十分复杂的网络,特别是人体的代谢网络,一直被认为是最复杂的代谢网络。现在多数信号通路的研究都是集中在代谢网络的一个很小的领域。基因组学、蛋白组学研究已经揭示了部分调节通路,但是和代谢网络直接相关的是代谢产物。但是从茫茫多的代谢产物中选取研究对象,无疑是大海捞针。代谢组学研究通过一定的手段能够帮助研究员从代谢产物海中跳出来,提供一个“航拍”的视角,一目了然地发现差异性代谢产物。然后通过已知的代谢通路逆推找出调节酶和基因,完成疾病发病机制、药物治疗机制等方面的研究。代谢组学主要研究的是作为各种代谢路径的底物和产物的小分子代谢物(MW1000)。其样品主要是尿液,血浆或血清,唾液,以及细胞和组织的提取液。主要技术手段是核磁共振(NMR ),液-质联用(LC-MS),气-质联用(GC-MS),色谱(HPLC,GC)等。通过检测一系列样品的谱图,再结合化学模式识别方法,可以判断出生物体的病理生理状态,基因的功能,药物的毒性和药效等,并有可能找出与之相关的生物标志物(biomarker)。代谢组学在新药的安全性评价,毒理学,生理学,重大疾病的早期诊断,个性化治疗,功能基因组学,中医药现代化,环境评价,营养学等科学领域中都有着极其广泛和重要的应用前景,是一门充满朝气的学科。 从近年来发表的相关SCI论文的数量可以看出代谢组学研究呈一个蓬勃发展的局面。从近年来国家拨付的相关研究基金也可以看出国家对代谢组学相关研究的重视。
历时6年,300余研究者花费5300万美金,牛的基因组序列终于呈现在世人面前,相关的文章发表在Science杂志上。这是继2000年人类基因组破解以来,又一动物基因组序列被破译。负责人称,牛的基因组的破译不仅有助人们更深入了解牛的驯化过程,提高牛肉,牛奶的质量改善人类的生活质量,还有助了解人类的疾病。最新的一期Science杂志刊登了两篇独立研究牛基因组的文章,一篇Genome-Wide Survey of SNP Variation Uncovers the Genetic Structure of Cattle Breeds;一篇The Bovine Genome Sequencing and Analysis Consortium,该项目对牛的基因组进行了分辨率精细的测序。另外还有一篇评论性的文章,The Genome Sequence of Taurine Cattle: A Window to Ruminant Biology and Evolution,将研究焦点放在对牲畜进化和驯养历史的追踪工作上。研究人员发现,牛的基因组含有至少2万2000个基因,其中大约有14345个基因在7种其它的哺乳动物种系中具有对应的基因。 这些发现显示,在牛的进化和驯养过程中,基因的数量和构成的变化是如何改变牛的生物学系统并对它们的繁殖、免疫能力、乳汁分泌和消化造成了最为显著的影响的。 这些研究人员还对来自19个不同地理和在生物学上混杂繁殖的497头不同牛只DNA中的3万7470种差异进行了调查。他们发现,母牛的进化与我们人类本身的进化截然不同,它们从一个有着非常大的有效祖先群体到近期发生的快速的群体下降,而不是反过来的那种一种情形。 文章的作者将这种进化归因于与以往驯化活动、因农业专门化所作的选择以及与动物豢养的形成相关的遗传学瓶颈。 但是,牛品种中的多样性的现有水平看来至少与那些在人类群体中的水平一样地强健有力。 在一篇Perspective中,Harris Lewin对这些发现进行了更为详细的探讨,并重点介绍了其对人类健康和可持续性农业的意义。
美国《科学》杂志近日公布的2012年度十大科学突破中,“基因组改造”的技术革新榜上有名,这一项中引用了清华大学结构生物学中心的重要工作成果。位列今年十大之首的是希格斯玻色子的发现,此外,丹尼索瓦人基因组、让干细胞形成卵子、“好奇”号着陆系统、基因组的精密工程、大脑/机器界面等入选。 “基因组改造”的技术革新引用了清华大学结构生物学中心的重要工作成果。这已经是清华的科研成果在近三年内第二次上榜《科学》的年度十大。 对基因组特别是高等生物基因组的定点改造,一直是生物学研究的一个难题。相关技术近年不断取得突破,特别是以TALEN(转录激活因子样效应蛋白核酸酶)为代表的技术突破,使得基因组改造便捷有效。科学家利用TALEN成功实现了对于斑马鱼、爪蟾、家畜猪,甚至人类细胞的定向改造。清华大学结构生物学中心颜宁教授和施一公教授合作解析了TALE蛋白与DNA结合的高分辨率晶体结构,从而揭示了这些蛋白特异识别其靶标基因的分子基础。这一工作今年1月5日发表于《科学》杂志,12月21日入选该杂志的年度十大。2009年,颜宁教授研究组的研究成果也曾入选当年的十大。
近日,深圳华大基因研究院和美国科学家共同发起“共生体基因组计划”。该计划将对海蛤蝓(又称绿叶海蜗牛)及藻类饵料进行基因组测序。有科学家认为,海蛤蝓可能是“生命之树”中动植物界的交叉点。海蛤蝓的细胞能够从藻类获取叶绿素,进行光合作用,从而为其所有生命活动提供足够的能量,包括繁殖。迄今为止,科学家在海蛤蝓基因组里发现了大约十多种藻类基因,这些基因使这种生物在叶绿素合成通道和碳固定循环中具有集光蛋白质和酶类的功能。随着研究的深入,不断有新的藻类基因在海蛤蝓基因组中被发现。海蛤蝓通过自身内被转移的藻类基因合成叶绿素,进行光合作用。这种神奇的共生现象第一次证明了一套完整的生物合成途径可以从一种多细胞生物传递到另一种多细胞生物。华大基因有关专家表示,通过对藻类和海蛤蝓的基因组进行比较,不仅将在宿主细胞中发现一组能够进行持续光合作用的基因,而且能够找到转移的特性,包括转移基因片段的大小、数量;更重要的是了解这种转移的运行机制。这些发现将对基因组的人工调控和基因治疗新技术的开发产生重大现实意义。此外,这两类生物的基因组测序将有利于比较基因组研究、进化规则、发展生物学及分类学的发展。据悉,这次联合研究是华大基因“千种动植物参考基因组计划”的一部分。该计划将在未来两年内建立1000种动植物的参考基因组序列。在“共生体基因组计划”中,华大基因主要负责测序和生物信息分析工作。《科学时报》 (2010-3-23 A1 要闻)
人类基因组单核苷酸多态性的研究进展与动态The research development of single nucleotide polymorphisms in human genome 摘要:第一张人类基因组序列草图已经公布,正式图预计也将于2003年4月完成。但序列图只基于少数个体,它反映了基因组稳定的一面,并未反映其变异或多态的一面,而正是这种多态性,即基因组序列的差异构成了不同个体与群体对疾病的易感性、对药物与环境因子不同反应的遗传学基础。人类基因组中存在广泛的多态性,最简单的多态形式是发生在基因组中的单个核苷酸的替代,即单核苷酸多态性(single nucleotide polymorphisms, SNPs)。SNP通常是一种二等位基因的(biallelic),即二态的遗传变异,在CG序列上出现最为频繁。在转录序列上的SNP称为cSNP。SNP的数量大、分布广。按照1%的频率估计,在人类基因组中每100~300个核苷酸就有一个SNP。因此,整个人类基因组(3.2 X 109bp)中至少有1,100万以上的SNPs,在任何已知或未知基因内和附近都可能找到数量不等的SNP 目前普遍认为,作为数量最多且易于批量检测的多态标记,SNP在连锁分析与基因定位,包括复杂疾病的基因定位、关联分析、个体和群体对环境致病因子与药物的易感性研究中将发挥愈来愈重要的作用。迄今,对多基因疾病候选基因的SNPs研究已积累了丰富的数据,基于这些SNPs的关联分析也正方兴未艾。本文阐述了SNP的特征、不同研究者对基于SNP进行关联分析的观点以及SNP的研究进展与动态。 关键词: SNP;遗传标记;关联研究 中图分类号:Q75 随着分子遗传学的进展,疾病遗传学研究从简单的单基因疾病转向于复杂的多基因疾病(如骨质疏松症、糖尿病、心血管疾病、精神性紊乱、各种肿瘤等)与药物基因组学的研究中。与前者相比,多基因性状或遗传病的形成,受许多对微效加性基因作用,即其中每种基因的作用相对较微弱。这些不同基因构成的遗传背景中,可能有易感性主基因(major gene)起着重要作用。它们同时还受环境因素的制约,彼此间相互作用错综复杂,所以任一基因的多态性对疾病发生仅起微弱的作用。鉴于此,需要在人类基因组中找到一种数目多、分布广泛且相对稳定的遗传标记,单核苷酸多态性(single nucleotide polymorphisms, SNPs)正是代表了这样一种标记,所以它成为继第一代限制性片段长度的多态性标记、第二代微卫星即简单的串联重复标记后,第三代基因遗传标记。 1. SNP作为遗传标记的优势 SNP自身的特性决定了它比其它两类多态标记更适合于对复杂性状与疾病的遗传解剖以及基于群体的基因识别等方面的研究。 (1)SNP数量多,分布广泛。据估计,人类基因组中每1000个核苷酸就有一个SNP,人类30亿碱基中共有300万以上的SNPs。SNP 遍布于整个人类基因组中,根据SNP在基因中的位置,可分为基因编码区SNPs(Coding-region SNPs,cSNPs)、基因周边SNPs(Perigenic SNPs,pSNPs)以及基因间SNPs(Intergenic SNPs,iSNPs)等三类。 (2)SNP适于快速、规模化筛查。组成DNA的碱基虽然有4种,但SNP一般只有两种碱基组成,所以它是一种二态的标记,即二等位基因(biallelic)。 由于SNP的二态性,非此即彼,在基因组筛选中SNPs往往只需+/-的分析,而不用分析片段的长度,这就利于发展自动化技术筛选或检测SNPs。主要的技术方法包括单链构象多态性(single strand conformation polymorphisms, SSCPs)法、异源双链分析(heteroduplex analysis, HA)、DNA直接测序分析、变异检测阵列(variant detector arrays, VDA)法以及基质辅助激光解吸附电离飞行时间(MALDI-TOF)质谱法等。 (3)SNP等位基因频率的容易估计。采用混和样本估算等位基因的频率是种高效快速的策略。该策略的原理是:首先选择参考样本制作标准曲线,然后将待测的混和样本与标准曲线进行比较,根据所得信号的比例确定混和样本中各种等位基因的频率。 (4)易于基因分型。SNPs 的二态性,也有利于对其进行基因分型。对SNP进行基因分型包括三方面的内容:(1)鉴别基因型所采用的化学反应,常用的技术手段包括:DNA分子杂交、引物延伸、等位基因特异的寡核苷酸连接反应、侧翼探针切割反应以及基于这些方法的变通技术;(2)完成这些化学反应所采用的模式,包括液相反应、固相支持物上进行的反应以及二者皆有的反应。(3)化学反应结束后,需要应用生物技术系统检测反应结果。目前许多生物技术公司发展出高通量检测SNP的技术系统,如荧光微阵列系统(Affymetrix)、荧光磁珠技术(Luminex,Illumina, Q-dot)、自动酶联免疫(ELISA)试验(Orchid Biocomputer)、焦磷酸的荧光检测(Pyrosequencing)、荧光共振能量转移(FRET)(Third Wave Technologies)以及质谱检测技术(Rapigene, Sequenom)。 2. 基于SNP的关联研究 如果某一因素可增加某种疾病的发生风险,即与正常对照人群相比,该因素在疾病人群中的频率较高,此时就认为该因素与疾病相关联。如非遗传因素吸烟与肺癌相关;在遗传因素中,如APOE4与Alzheimer`s相关。对疾病进行关联分析需要在年龄与种族相匹配的患者和对照人群中确定待测因素(环境的或遗传的)的频率分布,患者和对照人群的选择是否恰当直接影响结果的可靠性。对常见的由高频率、低风险等位基因导致的疾病,采用致病等位基因的关联分析比连锁分析更有效。 应用SNP进行关联研究,首先需明确多少SNPs才可满足在全基因组范围内的分析。Kruglyak应用计算机模拟法预测人类基因组中超过3Kb就不存在连锁不平衡,据此推出完成全基因组扫描将需要500,000个SNPs。而Collins等收集通过家系研究得到的常染色体单倍型的信息发现,在染色体上相距0.2cM到0.4cM(约200-400kb)之间的标记仍存在连锁不平衡,如按每100kb需要一个SNP计算,那么完成全基因组扫描仅需约30,000个SNPs,平均每3-4个基因用一个SNP就可识别出整个基因组内任何位置上的具表型活性的变异。最近发现SNP与SNP之间的连锁不平衡甚至可延伸到更远的区域(0.35cM-0.45cM),那么进行基因组扫描需要的SNP数量就更少。导致上述估算SNP 数量差异的主要原因是Kruglyak进行模拟计算时,假设现在的人群在5000年前起源于共同的祖先,且人群规模的有效大小保持在10,000左右,然后经过连续的指数扩增,直至达到现在的50亿左右。Collins认为这种假设是不现实的,在人类发展的历史过程中,人群数目的增长是迂回曲折的,经历扩张与萎缩的周期性变化。 Weiss等认为Collins及其同事的结果可能低估了问题的复杂性。因为他们的结果或是基于小样本资料推断出来的,就会使连锁不平衡(LD)程度的估算偏高;或是从理论上预测LD的水平,而忽略了基因组中大量的随机变异。如大多数位点的信息是来源于小样本中测序得到的资料,据此得到的单倍型结构不可靠。目前的研究集中于基因组中LD相对广泛存在的区域,在此区域内,基因相对容易作图。如基于这些经验来进行基因组其它区域的LD分析,就可能发生偏离。如两个相距较远的SNPs 之间具有强的LD性质,就认为它们之间的SNPs及该SNP侧翼的SNPs也存在强烈的LD,这种假设仅适合于其中一些多态位点,但它并不是通则。当然,在一些罕见人群中,如Saami,在较长的区域内广泛存在大量的LD,但对Fihland人群,则在较长区域内几乎不存在LD,对全球整个复杂人群而言,LD肯定变得更复杂一些。 Gray等认为随着人类基因组测序计划的进展,人类基因组的结构逐渐被阐明,因此就可在那些富含基因的区域选择SNP进行全基因组扫描,这样所需的SNP数量还会减少。Halushka等根据他们对75个基因检测的实验结果推测,SNPs在单个基因或整个基因组中的分布是不均匀的,在非转录序列中要多于转录序列,而且在转录区也是非同义突变的频率比其它方式突变的频率低得多。Templeton 等对LPL基因突变与重组热点的研究结果提示,SNP集中分布于基因组的CG二核苷酸处或单核苷酸重复区或αDNA聚合酶的识别位点(TGGA)处。将人类基因组不同区域物理图谱与遗传图谱的进行比较,发现遗传距离和物理距离的比值有很大的差异,提示基因组不同区域的重组水平存在差异。如Dunham等将22号染色体STR的物理位置与遗传位置进行了对比,发现该染色体的重组率差异很大,提示存在重组热点。根据基因组内不同区域重组频率的高低可进一步选择SNP的数量,重组热点需要的标记数量就多,相反就少。这种设计也可能会进一步减少基因组扫描所需的SNP标记。 使用SNP进行关联分析面临的另一个问题是如何选择SNP。如果对每一个SNP都进行独立研究,那么对几百万SNPs 的研究就会导致成千上万次的假关联,结果就掩盖真实的关联性,所以,进行关联分析前,一定要对所研究的SNP进行选
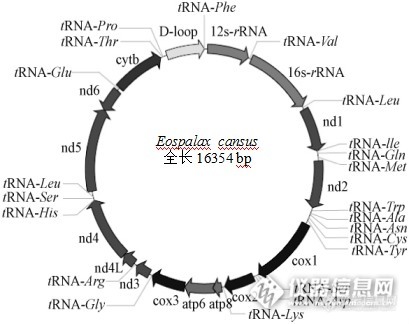
[b][/b][align=center]鼢鼠([i]Eospalax[/i])线粒体基因组测定及注释分析[/align][align=center]西安国联质量检测技术股份有限公司[/align][align=center]安平中心:李瑞[/align][b]摘要【[/b]目的】获得鼢鼠线粒体基因组全序列,为线粒体基因组功能标记及进化生物学等研究提供基础资料。【方法】参考鼹型鼠等动物的线粒体基因组序列,设计出可覆盖鼢鼠线粒体基因组的16对引物,采用[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]产物直接测序法测得甘肃鼢鼠线粒体基因组全序列,分析其基因组的特点和基因结构。并结合GenBank中发表的啮齿类动物基因组全序列,探讨啮齿类动物的系统进化关系。【结果】鼢鼠线粒体基因组全长16354bp,其中包括22个tRNA基因、13个蛋白质编码基因、2个rRNA基因和2个D-loop区。碱基组成为33.5%A、24.2 %C、12.3 %G、30.0 %T。【结论】鼢鼠线粒体基因组结构及其信息和其他啮齿类动物的结构一致,线粒体变异保守。研究结果为鼢鼠的低氧适应、系统发育关系等提供了基础资料。[b]关键词 [/b]鼢鼠;线粒体基因组;序列分析 鼢鼠([i]Eospalax[/i])是分布于我国的主要啮齿类动物之一,其体型较小,栖息于洞穴内有挖掘活动,扩散能力强,数量波动大,是生态系统中重要的初级消费者,处于生态系统中的中心位置,草原生态系统中其能流比重很大[sup][/sup]。动物线粒体([color=#333333]Mitochondrion[/color])基因组为双链闭合环状分子[sup][/sup],少数也有线性的,它们具有分子量相对较小、结构简单、缺少重组、母性遗传和进化速率快等特点,已成为动物系统发育与进化、群体遗传学、分子生态学以及疾病机理研究等领域的理想材料[sup][/sup]。甘肃鼢鼠是仅分布于我国西北部的土著物种,其外形似中华鼢鼠,主要分布于甘肃临潭县及其附近地区。目前对线粒体DNA的研究主要在动物分子遗传学、分子生态学、种群遗传结构分析、遗传多样性、物种和品系鉴定、保护遗传学等方面得到了广泛应用[sup][4[/sup][sup],[/sup][sup]5][/sup]1. [b]实验材料和方法[/b]1.1 实验材料鼢鼠:采集于天祝(经度102.84、纬度 37.2)1个群体;鼢鼠解剖采集肝脏及肌肉组织样品,-20℃保存备用。1.2 线粒体DNA的提取用剪刀将肝脏及肌肉材料剪成小块,取0.1cm左右的小块肝脏及肌肉材料,采用常规的SDS/蛋白酶K裂解,酚氯仿提取DNA[sup][/sup],使用琼脂糖凝胶电泳检测其完整性。1.3 引物设计和[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]扩增通过Clustal X1.83比对,寻找相对应保守区域位置,用Primer Premier5.0引物设计软件设计引物,并对每条引物进行评价和修改,最终确定16对引物。以所提取的DNA为模板,用16对引物扩增覆盖整个线粒体基因组。利用引物进行[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]扩增,反应体系总体积为50μL,其中含有6μL [url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url] buffer、3μL MgCl[sub]2[/sub](1.5mmol)、MgCl[sub]2[/sub],2μL dNTPs (100μL mol)、上下游引物各2μL (0.25μL mol)、Taq DNA聚合酶2μL (1U)、总DNA约为2μL (25ng)、去离子水31μL。反应程序为:94℃预变性4 min,94℃变性50s,48-45℃退1min,72℃延伸1 min 30s,循环30次,之后72℃延伸10min,并根据不同引物的退火温度和扩增反应的实际效果进行优化。取 5 μL [url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]扩增产物,和2 μL DNA marker 2000,进行1.0%琼脂糖凝胶(1×TBE)5V/电泳,用紫外观察[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]产物扩增情况,凝胶成像仪扫描记录结果。1.4 纯化、测序和序列拼接 在[url=https://insevent.instrument.com.cn/t/jp][color=#3333ff]PCR[/color][/url]产物中加入5 U SAP和2 U ExoⅠ,震荡混匀,37℃保温1 h,然后75℃保温15 min以灭活SAP和ExoⅠ酶,纯化好的模板可以在4℃保存24 h或-20℃长期保存。将纯化后的引物送往上海生工生物技术服务有限公司用ABI-3730序列自动分析仪进行双向测序。利用DNASTAR和测序峰图结果分析软件Chromas 2.22校对测序图,DNAMAN拼接序列。得到甘肃鼢鼠线粒体全基因组全序列。2. [b]结果[/b]2.1 鼢鼠线粒体基因组基因定位2.2.1 鼢鼠线粒体2个rRNA的分析哺乳动物线粒体的rRNA具有高度的保守性,它们的位置固定,12S rRNA位于tRNA-phe 和tRNA-Val之间,16S rRNA位于tRNA-Val和 tRNA-Leu之间,12S rRNA起始位置为68,终止位置为1019,长度为952bp,16S rRNA起始位置为1086,终止位置为2651,长度为1566。同时我们比对了鼢鼠和中华鼢鼠的rRNA基因和蛋白质基因,12S rRNA和16S rRNA的相似性分别为91.0%和87.3%,高于蛋白质编码基因之间的相似性。2.2.3 鼢鼠线粒体基因组结构 除NADH脱氢酶亚基6外均在H链上,虽然鼢鼠染色体数目少、染色体大,但与其它哺乳动物线粒体全基因组相比,它的线粒体基因组的结构与其它哺乳动物是十分相似的。甘肃鼢鼠线粒体基因组结构见图1。[align=center][img=,409,324]http://ng1.17img.cn/bbsfiles/images/2017/09/201709081454_02_2904018_3.png[/img][/align]注:ND: NADH脱氢酶亚基(NADH dehydrogenase subunit)、Cox:细胞色素氧化酶亚基(cytochrome oxidase subunit)、Atp:ATP合成酶亚基(ATP synthase F0 subunit)、Cyt b:1个细胞色素b编码基因(cytochrome b)。[align=center][b]图1[/b] 甘肃鼢鼠线粒体基因组结构简图[/align][align=center]Fig.1 The gene organization of [i]Eospalax cansus[/i] mitochondrial genome[/align]3. [b] 讨论[/b] 甘肃鼢鼠线粒体基因组的D-loop区,长度为933bp,比中国地鼠D-loop区(867bp)长。D-loop区对目的基因是不可缺少的,虽然D-loop区不能编码蛋白质但对于遗传信息表达是不可缺少的,在它上面有调控遗传信息表达的核苷酸序列,具有遗传效应的,比如RNA聚合酶结合位点是具有遗传效应的。8只甘肃鼢鼠中有5个单倍型:3只临潭群体共享1个单倍型,2只天祝群体独享单倍型;其余个体均独享单倍型,表明了甘肃鼢鼠线粒体DNA D-loop区碱基变异快、进化快的特性,符合啮齿动物线粒体变异大的现象。随着研究的深入,以线粒体DNA中完整的基因序列或多个基因序列协同而获得遗传信息来探讨物种的系统进化关系,将是以后研究发展的主要方向[sup][/sup]。目前,线粒体DNA已经在许多哺乳类动物的起源进化的研究中取的了重大进展,而对甘肃鼢鼠的起源进化的研究却很少,并且存在着甘肃鼢鼠属于[url=http://baike.baidu.com/view/113192.htm][color=#000000]瞎鼠科[/color][/url]和仓鼠二者之争,因此,为了更好的阐明甘肃鼢鼠的起源,还需要做更多、更深入的研究。
7月20日,我国科学家宣布“兰花基因组计划”正式启动。两岸科学家将联手对被喻为“植物界大熊猫”的兰科植物进行全基因组测序和生物信息分析,同时对10种最具代表性的兰科植物进行基因表达的转录组测序和分析。 国家兰科植物种质资源保护中心刘仲键教授介绍,对兰科植物的科学研究历史悠久,其成果为达尔文进化论提供了强有力的支持。兰花研究为进化生物学乃至整个生命科学的发展贡献巨大,至今仍是研究生命与进化的理想模式,占有特殊地位。同时,兰花也是世界性濒危物种,是国际公约保护物种的重中之重。兜兰与国宝大熊猫同列为一级保护,其余兰花全部被列入二级以上保护。 清华大学黄来强教授称,兰花全基因组及转录组测序分析,将为人类提供用现代生物学的新技术和理念从分子生物学的层面审视达尔文的研究,为进化生物学和进化论注入新鲜血液。在基因组和转录组的研究基础上进一步结合生物信息、分子生物、蛋白质组、代谢组、生化、生物物理等多学科和研究手段的融合,对加深其基因组结构及功能的了解,揭示兰科的进化,对生命科学研究具有普遍的重要意义。“兰花基因组计划”涉及的不仅是植物学,还将为世界上相关研究提供全新的起点和平台,是对全球基因组科学的又一重大贡献。 “兰花基因组计划”项目,由深圳兰科植物保护研究中心(国家兰科植物种质资源保护中心)、清华大学、深圳华大基因研究院、中国科学院植物所、台湾成功大学等单位科学家共同承担。
无需进行文库制备,所用DNA样本比标准方法更少2012年12月13日 来源: 中国科技网 作者: 陈丹 中国科技网讯 据物理学家组织网12月12日(北京时间)报道,英国研究人员简化了基因组测序的标准流程,首次无需进行文库制备便完成了DNA(脱氧核糖核酸)单分子测序,而且新方法只要很少量的DNA就能获得序列数据,用量可低至不到1纳克(10亿分之一克),仅为常规测序方法的500分之一到600分之一。 文库制备是指从测序前基因组样本中提取不同长度的DNA片段,这一过程不仅费力、费时,还会浪费DNA,而新技术能极大地减少DNA的损耗,并缩短测序时间。 该研究论文的第一作者、英国威康信托基金会桑格研究所的保罗·库普兰说:“我们用这种方法对病毒和细菌的基因组测序后发现,即使在相对较低的水平,我们也能够确定所检测的是何种有机物,不论样本中是否存在特定的基因或质粒(这对于确定抗生素耐药性很重要),或者其他信息,如对特定DNA碱基的修改等。”他表示,一旦技术得到优化,将在快速、高效地识别医院和其他医疗场所中的细菌和病毒方面具有很大的应用潜力。 研究小组利用第三代单分子测序系统PacBio RS演示了这种简化的直接测序方法。他们仅仅用800皮克(千分之一纳克)DNA来分析一个生物体的基因组,尽管测序仪只读取了基因组的70个序列片段,相对于常规测序方法获得的数据来说不过是很小的一部分,但这些信息足以让研究人员确定他们所检测的生物体的品种。 这项技术也使得科学家能够对此前无法识别的宏基因组(也称微生物环境基因组)样本中的生物体进行确认。“为微生物测序,首先需要能够在实验室中培养它们。”论文的主要作者、英国巴布拉汉研究所的塔米尔·钱德拉说,“这不仅耗费时间,而且有时候微生物不生长,为它们的基因组测序极其困难。”他表示,新方法可以直接对微生物测序,短时间内便可确定其“身份”。 论文的另一主要作者、威康信托基金会桑格研究所的哈罗德·斯维尔德洛说:“我们的技术可以在对所测序列没有任何先验知识、没有特定微生物试剂的条件下,在很短的时间内操作,这是一种很有前途的替代手段,可应用于控制感染等临床需要。”(记者陈丹) 总编辑圈点 长久以来,基因测序等围绕基因科学所展开的研究,都被人们贴上了从本源上解开人体生命奥秘、彻底解除遗传疾病威胁等殷切的标签。多国为提高社会健康水平,都开展了解码国民DNA的活动,有些甚至覆盖全基因组。然而,面对由30亿个碱基对构成的人类基因组,精确测序注定将是一场浩大而又漫长的工程。如何能快速、准确地将海量DNA数据转化为有帮助的实用信息,已经成为该领域科学家们面临的重大挑战之一。因而我们说,英国科学家此番取得的突破,不管是从整个学科研究的方法论层面,还是从临床应用的角度,都提高了基因研究服务于人类的速度。 《科技日报》(2012-12-13 一版)
代谢组学研究代谢组学(metabonomics/metabolomics)是继基因组学和蛋白质组学之后新近发展起来的一门学科,对生物体内所有代谢物进行定量分析,并寻找代谢物与生理病理变化的相对关系的研究方式,是系统生物学的组成部分。基因组学和蛋白质组学分别从基因和蛋白质层面探寻生命的活动,而实际上细胞内许多生命活动是发生在代谢物层面的,如细胞信号释放(cell signaling),能量传递,细胞间通信等都是受代谢物调控的。代谢组学正是研究代谢组(metabolome)——在某一时刻细胞内所有代谢物的集合——的一门学科。其研究对象大都是相对分子质量1000以内的小分子物质。先进分析检测技术结合模式识别和专家系统等计算分析方法是代谢组学研究的基本方法。化学分析技术中最常用的是^1H核磁共振(^1HNMR)以及色谱(毛细管电泳)-质谱联用(X—MS)。代谢组学属于全局系统生物学(Global systems biology)研究方法,便于对复杂体系的整体进行认识.譬如,一个正常工作的人体包括“人体”本身和与之共同进化而来且共生的消化道微生物群体(或称菌群),孤立地研究“人体”本身的基因,转录子以及蛋白质当然可以为人们认识人体生物学提供重要信息,但无法提供使人体正常工作不可缺少的菌群的信息.人体血液和尿液的代谢组却携带着包括菌群在内的每一个细胞的信息,因此代谢组学方法对研究如人体这样复杂的进化杂合体十分有效.早在20世纪60年代,代谢物组学的核心技术——核磁共振技术(NMR),就已经被应用到代谢研究中。但直到20世纪90年代,随着模式识别分析技术的发展,代谢谱的定量分析才得以实现,并应用于药物和基因功能的研究。利用代谢物组学研究药物对整体的作用主要依赖于多参数检测外源物质攻击所导致的机体新陈代谢改变。这种方法也适合研究基因突变和转基因所产生的代谢改变以及疾病诊断和疗效评价。在药物发现阶段,它可以进行体内毒性研究、先导药物的筛选和目标化合物的优化及体内动物模型的药效筛选。在药物开发阶段,它可以在临床前安全性评价方面进行生物标志物的发现和毒性机制的研究,从而可有效地利用动物模型研究人类疾病的治疗,发现与临床安全性和有效性有关的生物标志物。代谢组学已经广泛地应用到了包括药物研发,分子生理学,分子病理学,基因功能组学,营养学,环境科学等重要领域.在代谢组学诞生的过去6年里,有关代谢组学的研究论文和专利以指数的形式逐年增长.可以预见,这门新兴学科将应用到更为广泛的领域.
据8月28日的《科学》杂志报道说,蚕虫驯养已经有1万多年历史了。蚕为人类提供了宝贵的丝绸和蛋白。但是,现在对蚕基因进行序列测试还为人们提供了一张有关这些随时会为我们提供如此多宝贵物质的昆虫的基因变异图。由西南大学、深圳华大基因带领的国际研究团队为29种家蚕和11种野蚕世系的基因组成功地进行了测序并找到了这些世系之间的差别。共获得了40个家蚕突变品系和中国野桑蚕的全基因组序列,共测632.5亿对碱基序列,覆盖了99.8%的基因组区域,是多细胞真核生物大规模重测序研究的首次报道;绘制完成了世界上第一张基因组水平上的蚕类单碱基遗传变异图谱,这是世界上首次报道的昆虫基因组变异图。科学家还发现了驯化对家蚕生物学影响的基因组印记,从全基因组水平上揭示了家蚕的起源进化。 研究发现,家蚕很明显地在基因上与其野生对应物不同,但即使在各家蚕世系之间,它们仍然维持着大量的变异性。这提示,家蚕只经历了一次牵涉有大量个体的单一且短暂的驯养过程,并在此后在家蚕与野蚕种群之间很少有基因流动。研究人员还能够识别出特别的能够增进丝的生产、蚕虫的繁殖和生长的基因(这些基因很可能是被人类挑选出的)。他们甚至还寻找到了在驯养过程中由蚕虫所获取的行为特征,例如极端的拥挤和容忍人的靠近和操作,以及它们在驯养过程中所丧失的如逃逸及躲避掠食者和疾病等的特征。(
来自英国Sanger研究院,Illumina Cambridge公司等处的研究人员发表了题为“Genome Sequencing and Analysis of the Tasmanian Devil and Its Transmissible Cancer”的文章,完成了一种传染性癌症的基因组测序,并从中发现了一些突变,解析了这种癌症的来源,以及如何变得具有传染性的。相关成果公布在Cell杂志上。这种癌症主要发生在世界上最大的肉食性有袋动物:袋獾身上,这种动物也被称为塔斯马尼亚恶魔(Tasmanian Devil),现今只分布于澳大利亚的塔斯马尼亚州。袋獾是袋獾属中唯一未灭绝的成员,其在研究领域最著名的就是袋獾面部肿瘤疾病。袋獾面部肿瘤是一种独特癌症,常出现于袋獾面部或嘴部,但通常会扩散至袋獾的内脏,它与另外一种在犬类中传播的恶性肿瘤是世界上仅有的两种可通过上述方式传播的癌症。这项研究离心机揭示了这种能通过撕咬在动物间传播的肿瘤的奥秘,首次针对一个雌性袋獾的单细胞进行分析。这个雌性袋獾被称为“永恒恶魔(The Immortal Devil)”,因为其死于15年前,但它的DNA仍然在传染癌细胞系中流传。文章的第一作者,Sanger研究院Elizabeth Murchison博士表示,“袋獾癌症是目前发现的唯一一种威胁到整个物种灭绝的癌症”,“通过其测序,将有助于我们整理引发整个袋獾群体癌症的突变。”研究人员从中找到了肿瘤细胞之间的遗传差异,这表明这种癌症在袋獾群体中传播的时候,发生了遗传突变。他们在塔斯马尼亚州不同地区找到了69种不同袋獾的肿瘤样品,构建袋獾面部肿瘤传播的图谱,研究结果表明一些癌症亚型比其它亚型更具有侵染性。Illumina Cambridge公司David Bentley说,“我们发现这种癌症的基因组具有大约两万个突变,这比某些人类癌症中发生的突变更少,这说明癌症变得具有传播性,基因组极度不稳定并不是必要条件”,“追踪这种癌症的进化历史,以及其传播过程,将有助于我们了解这种疾病发生的原因,以及预测其未来的发展。”癌症在个体之间的传播正常来说,会受到免疫系统牛血清蛋白的干涉,因为免疫系统可以鉴别外来组织,这一研究组发现了一些有趣的线索——这种癌症如何能“智斗”免疫系统,比如免疫系统中的一组基因突变。但是还需要更进一步的研究,揭示这种癌症是如何从免疫系统中逃脱出来的。“这项研究十分重要,因为这将会帮助我们理解疾病传播的模式,也有助于疫情的研究,但是我们还需要利用这一基因组测序,更进一步分析这种癌症如何变得具有传染性。癌症具有群体传播性,显示是非常罕见的,我们通过袋獾这一例子来分析这一过程,以防未来在人类身上发生”,Sanger研究院,文章通讯作者Mike Stratton教授说。研究组下一步将进行更多袋獾基因组测序,绘制上千袋獾肿瘤样品基因组图谱,从而更好的了解这种癌症的遗传多样性,并分析癌症与袋獾群体之间的遗传关联性。去年这一研究组在Science杂志上发表文章,发现培养基袋獾面部肿瘤起源于雪旺细胞。他们从分布在澳大利亚塔斯马尼亚岛14处的袋獾群落中采集了25个袋獾面部肿瘤样本,进行基因分析,结果发现,袋獾面部肿瘤起源于雪旺细胞,在大约20年前,袋獾雪旺细胞内的某种基因变异导致了这一癌变。
[font=宋体][size=3]中国医学科学院药用植物研究所与广药集团今天在京宣布“丹参基因组框架图”绘制完成。这是世界上首个药用植物基因组框架图。[/size][/font][font=宋体][size=3] 广州白云山和记黄埔中药有限公司与中国医学科学院药用植物研究所合作,利用第二代高通量测序技术对丹参全基因组进行测序,并完成丹参基因组框架图的组装。丹参基因组框架图的完成,对其它药用植物的研究具有很好的借鉴和示范作用,促进现代前沿生命科学研究和传统中药学的有机结合,将改变中药研究领域被动追赶其它学科发展的局面。[/size][/font]
1.前言 随着科学与技术的发展,新药研发的速度正在日益加快,使得新药安全性评价工作的压力也变得越来越大。在新药研究开发过程中,因为安全性问题而被淘汰的候选药物占相当大的比例。一旦潜在的药物分子通过了初步的生物学筛选过程,就应该尽量减少这些候选药物分子在产品研发过程中的流失,以免造成巨大的资金和时间的浪费。因此,人们努力寻找新的分析方法,以便从功效和安全性两方面使得先导化合物的筛选更有效,从而尽可能地减少这种浪费。目前的生物分析手段主要利用基因组和蛋白质组方法,分别从基因水平和细胞蛋白质表达水平上测量生物体系对药物的反应。这两种方法都较昂贵,且劳动强度较大,然而却可能是研究在不同水平上对生物异源物质的生物应答的有力工具。但是,基因组学和蛋白质组学都不能提供可以了解生物体中整体细胞功能的信息,因为两者都忽略了整体器官中动态的代谢状态。因此,Nicholson等人提出了一种基于核磁共振的新方法,叫做metabonomics,我们暂且称之为代谢组学,以便与由代谢物组(metabolome)衍生而来的metabolomics相区别。Metabolomics研究的是一个细胞或细胞类型中所有的小分子成分,而metabonomics则是通过分析生物体液和组织来对完整的生物体(而不是单个细胞)中随时间改变的代谢物进行检测、确定、定量和分类;然后将这些***代谢轨迹与病生理过程中的生物学事件关联起。从药物研究和毒理学评价的角度来看,基因组学方法是观察给药后基因表达的改变,主要采用基因芯片技术。然而,基因调节/表达与系统的整体功能之间的关系在目前还很不清楚,主要是因为决大部分DNA是非编码的,而编码蛋白质的基因不能孤立地发挥作用,而是需要与其邻近的基因和非编码DNA一起才能发挥其功能。正式由于这个原因,人们才发展了蛋白质组学。蛋白质组学方法可以对由给药或其它病生理过程引起的细胞蛋白质组成变化进行半定量的测量。蛋白质组方法所采用的技术主要包括双向凝胶电泳和质谱技术。与基因组方法相比,蛋白质组方法较慢,且劳动强度较大。需要强调的是,虽然这些方法能够在很大程度上揭示毒理学机理,并且给出与疾病相关的新的生物标记物,却很难将这些发现与经典的毒理学指标相关联。原因很简单,因为目前的技术和方法不能对给药后反应的整个进程进行测量,也不能对生物整体的应答进行测量。因此需要发展一种新的方法来实时给出多器官生物整体的在体信息。基于NMR的代谢组学(metabonomics)方法可以满足这样的要求。2.Metabonomics在药物毒理学研究中的应用 代谢组学的目的是要扩展和补充由基因组学和蛋白质组学方法得到的对生物异源物质应答的信息。其任务是定量测量生物体对病生理刺激或基因改变的动态多参数代谢反应,是研究药物毒性和基因功能的技术平台。这个概念是根据Nicholson小组近二十年来利用1H NMR技术研究生物体液、细胞和组织中多组分代谢组成的工作而提出的。在这些研究中,还利用了模式识别,专家系统和相关的生物信息学工具。在许多情况下,药物通过与遗传物质直接作用而产生毒性,或通过诱导系统合成与药物代谢有关的酶,从而产生有毒的产物。在这种情况下,用基因组和蛋白质组学方法来评价毒性是有用的。然而,在生物异源物质有可能只在药理学水平上产生作用,因而可能不会影响基因的调节和表达。再者,显著的毒理学效应可能与基因的改变和蛋白质的合成完全不相关。因此,在许多情况下,从基因组和蛋白质组角度考虑到的反应可能不能预测药物毒性。但是,所有的由药物引起的病生理紊乱都会由于直接的化学反应,或通过与控制代谢的酶或核酸相结合而引起内源生化物质在比例、浓度、代谢通量等方面的失调。如果这种变化足够大的话,就会影响整个生物体的功能。生物体液中的代谢物是与细胞和组织中的代谢物处于动态平衡,因此,生物体中由于中毒或代谢损害而引起的细胞功能异常一定会反映在生物体液成分的变化中。要检测血浆、尿液、胆汁等生物基质中的一些具有特殊意义的微量物质,选择合适的分析方法致关重要。高分辨1H NMR波谱就非常适合用来检测生物体液中的成分异常,因为该方法可以同时对所有的代谢物进行定量分析,而且不需要样品前期准备,对任何成分一样灵敏。虽然也可以采用如质谱等其它方法,但对不同成分离子化程度的差别会影响定量和检测的可靠性。NMR方法还可以有效地用来从组织萃取物或细胞悬液中找出异常的代谢物。还可以利用高分辨魔角旋转(HR-MAS)探头来检测完整组织中的代谢物组成。由1H NMR谱检测到的生物体液中的内源性代谢物模式完全依赖于动物体内的毒素的类型。每一种类型的毒物都会在生物体液中产生特征的内源代谢物浓度和模式变化,这种特征给我们提供了毒性作用的机理和毒性位置的信息。右图所示为一系列尿样的1H NMR谱图,是大鼠经不同的毒物处理后得到的。每一张谱图只需几分钟的时间,是非常有效的。可以看出,不同毒素引起的代谢物变化是有特征性的。因为几乎所有的代谢物都有其特征的NMR谱,因而可以作为毒物引起的代谢变化的指纹图谱。利用NMR方法,人们已经成功地发现了许多新的器官特异相关毒性的代谢标记物。作为分析生物化学技术,NMR正是在这种探索性的工作上具有优势。
人类基因组计划的顺利实施,使生命科学研究的重心正逐渐转到生物功能的整体研究。基因组学由于自身的局限性,它不能回答诸如:蛋白质的表达水平和表达时间,翻译后修饰以及蛋白质与蛋白质或与其他生物分子的相互作用等问题。作为基因研究的重要补充,蛋白质组学在蛋白质的水平上定量的、动态的、整体的研究生物体。蛋白质组(Proteome)概念是最早是由澳大利亚学者Wilkins和Williams于1994年提出的,即基因所能表达的全部蛋白质,更为清楚的表达是细胞或组织或机体在特定时间和空间上表达的所有蛋白质。具体说它是对不同时间和空间上发挥功能的特定的蛋白质组群进行研究,进而在蛋白质的水平上探索其作模式、功能机理、调节调控以及蛋白质组群内的相互作用,从而为临床诊断、病理研究、药物筛选、新药开发、新陈代谢途径研究等提供理论依据和基础。 详情请见:[url=http://www.instrument.com.cn/hot/HA_56.htm]热点应用:蛋白质组学研究[/url]
人类基因组计划的顺利实施,使生命科学研究的重心正逐渐转到生物功能的整体研究。基因组学由于自身的局限性,它不能回答诸如:蛋白质的表达水平和表达时间,翻译后修饰以及蛋白质与蛋白质或与其他生物分子的相互作用等问题。作为基因研究的重要补充,蛋白质组学在蛋白质的水平上定量的、动态的、整体的研究生物体。蛋白质组(Proteome)概念是最早是由澳大利亚学者Wilkins和Williams于1994年提出的,即基因所能表达的全部蛋白质,更为清楚的表达是细胞或组织或机体在特定时间和空间上表达的所有蛋白质。具体说它是对不同时间和空间上发挥功能的特定的蛋白质组群进行研究,进而在蛋白质的水平上探索其作模式、功能机理、调节调控以及蛋白质组群内的相互作用,从而为临床诊断、病理研究、药物筛选、新药开发、新陈代谢途径研究等提供理论依据和基础。 详情请见:[url=http://www.instrument.com.cn/hot/HA_56.htm]热点应用:蛋白质组学研究[/url]
来自美国加州大学旧金山分校格拉斯通研究所的研究人员揭示出利用胚胎心脏细胞构建出完全功能性的心脏所需的上百个基因开关的精确开闭次序和时间。这项发现有助于对一些人先天性心脏病的遗传基础产生新的认识。在一项刊登于Cell期刊上的研究中,研究人员利用干细胞技术、下一代DNA测序和计算工具来将心脏细胞如何变成心脏的“基因组蓝图”拼接在一起。这些发现提供新的希望来对抗威胁生命的心脏缺陷,如心律不齐和室间隔缺损(ventricular septal defect)。在这项研究中,研究人员获取来自小鼠的胚胎干细胞,然后通过在盘碟中模拟胚胎发育而让它们分化为跳动的心脏细胞。接着,他们提取发育中的心脏细胞和成熟的心脏细胞内的DNA,并利用一种被称作ChIP-seq的高级基因测序技术来观察DNA中的表观遗传标记。论文共同第一作者Jeffrey Alexander说,“但是发现这些标记只是成功的一半,因此我们接着不得不破解它们编码心脏形成的哪些方面。为此,我们利用格拉斯通研究所生物信息学核心(Gladstone Bioinformatics Core)的计算能力。这允许我们获得基因测序中所收集的大量数据,并且将这些数据组装成一种可读的和有意义的将心脏细胞如何变成心脏的蓝图。”研究人员获得了一些意料之外的发现。他们发现在心脏细胞中,一组基因似乎以一种协作的方式一起发挥作用:在胚胎发育的指定时间,这组基因一起开启和关闭。他们不仅鉴定出很多参与心脏形成的新基因,而且也精确地确定地这些新发现的基因如何与之前已知的基因相互作用。绘制出心脏的基因组蓝图对人类健康的影响非常深远。鉴于研究人员理解这些基因如何控制心脏形成,他们能够开始将心脏病如何破坏这种调节的细节汇聚在一起。最终,他们能够寻找疗法来阻止、中断或抵消患有先天性心脏病的儿童体内这种调节遭到的破坏。
第三张“基因变异图谱”与第二代基因组测序技术——评“千人基因组计划”首期研究成果的医学意义世界上任意两个人的基因99%都是相同的,而恰是那1%不同,负责着个体间的表型差异。《自然》杂志近期披露,当人体内携带有250到300基因变异位点的时候,相关基因就就会“沉默”。甚至,一个人只携带了 50到100基因变异位点,就可能患上某种疾病。10年前,“人类基因组计划”这一耗资30亿美元、历时10余年的伟大科学工程完成之际,人们以为得到了揭开自身生命奥秘的天书,生命科学也划时代地进入了“后基因组时代”。如今看来,当时得到的仅仅是人类基因组的“参考图谱”,对于人群里个体间的基因差异,或是更具医学意义的“基因变异图谱”来说,人们知之甚少。第三张“基因变异图谱”为了探寻个体间的基因差异,科学界在2002年启动了HapMap(人类基因组单体型图谱)计划。Hapmap在2005年完成的“第一张基因变异图谱”含有一百万个“单核苷酸多态性”(SNPs)位点;HapMap在2008年完成的“第二张基因变异图谱”含有三百一十万个SNPs位点。而此次“千人基因组”所公布的一期结果——“第三张基因变异图谱”,已经包含了一千五百万个SNPs位点。今年10月28日,《自然》杂志为此刊出的文章题目为“基于群体规模的基因变异图谱”,鲜明的指出,“千人基因组计划”首期研究成果,其最大优势在于:“第三张基因变异图谱”所采用的样本,针对了“大规模人群”。 远超过此前两张“基因变异图谱”所测定的样本数。绘制“第三张基因变异图谱”的所有数据,是基于两个核心家庭,6个个体的精确基因组测序,179个个体的低覆盖率基因组测序,以及七百多人的蛋白编码区的基因测序。检测人群数目庞大,人种涉及中国人、日本人、西欧人等。因此,第三张“人类基因变异图谱”的问世,可以从更深的层次上了解,种族之间、个体之间的基因差异。更具医学意义的是,对于人群中发生频率在1%以上的基因变异,本次研究的覆盖率达到95%以上。这就意味着:此前Hapmap计划所绘制的两张“基因变异图谱”中,没能涉及的“罕见病”致病基因,可能在“第三张基因变异图谱”中已经被标出。“基因变异图谱”的医学应用随着,“人类基因变异图谱”绘制的日臻完善,和商业化全基因组SNP 分型芯片成本的不断降低,以及新的统计方法和软件的出现, “全基因组关联分析”( Genome-Wide Associat ion Study , GWAS) 越来越多的应用于复杂疾病“易感基因”的确定。今年6月6日,安徽医科大学的张学军教授领衔的团队,通过对中国汉族和维吾尔族人群近2万份样本进行分析,在人类基因组的3个区域内发现与白癜风发病密切相关的4个易感基因。今年8月2日,中***事医学院贺福初院士领衔的蛋白质组学国家重点实验室,通过对大陆5个肝癌高发区的4500多名肝癌病例和对照的研究,发现了肝癌易感基因新区域(1p36.22)今年8月23日,新乡医学院的王立东教授联合国内18家医院,建立了数十万份的食管癌标本资料库,并首次在人类第10号和20号染色体上,发现两个食管癌易感基因(PLCE1和C20orf54)。基因变异有着很强的人种差异,相比国外此领域的研究成果,以上研究成果的临床意义,在于其是针对我国的特有人群。也就是说,以上研究成果在我国的临床上更具医学价值。更为可喜的是,以上研究成果均发表在此领域最为权威的《自然 遗传学》杂志上。我国在利用GWAS需找复杂疾病易感基因领域的研究,已经得到了世界的公认。
科学家首次测序癌症患者基因组美国科学家近日首次成功测序了一个癌症患者的基因组,这一开创性工作为利用新方法揭开癌症的遗传学基础创造了条件。相关论文发表在11月6日的《自然》(Nature)杂志上。测序的基因组来自于一位女性,50多岁死于急性骨髓性白血病(AML)。美国华盛顿大学的研究人员利用来自皮肤样本的遗传材料,测序了她2套染色体的DNA,同时根据骨髓样本检测了其肿瘤细胞中的遗传突变。所有样本均采自患者接受癌症治疗前,以防DNA受到进一步损伤。随后,研究人员将患者的肿瘤基因组与其正常基因组进行了比较,以期发现遗传差异。在患者肿瘤基因组中接近270万个单核苷变异中,将近98%同样也在患者皮肤样本的DNA中检测到,这就大大缩小了进一步筛选的范围。研究人员最终在患者的肿瘤DNA中仅发现了10个可能与AML有关的遗传突变,其中8个很罕见,它们所处基因之前从未被认为与AML有关。研究人员还显示,肿瘤样本中的每个细胞拥有9个突变,而且较少发生的那个突变可能是最后形成的。研究人员怀疑,所有这些突变对于患者的癌症都很重要。美国国立人类基因组研究所前任主管Francis Collins说:“首次确定人类癌症基因组的完全DNA序列,并与同一个体的正常组织相比较,这在癌症研究中是一个真正的里程碑。”美国俄勒冈健康与科学大学癌症研究所的Brian Druker说:“虽然这一研究尚不能告诉我们怎样治疗癌症患者,但它是这条路上关键的第一步。它为大规模癌症基因组测序和揭示癌症秘密打下了基础。”目前,研究小组正在测序其他AML患者的基因组,同时他们还计划将这种全基因组方法扩展到乳腺癌和肺癌。
一、实验目的 通过采用改进的SDS法提取植物叶片基因组DNA,使学生学习和掌握从植物组织中提取DNA的方法和原理。二、实验原理 基因组DNA的提取通常用于构建基因组文库、Southern杂交、RFLP、PCR分离基因和分子标记分析等。利用基因组DNA序列较长的特性,可以将其与细胞器或质粒等小分子DNA分离。加入一定量的异丙醇或乙醇,大分子的基因组DNA形成沉淀,而小分子DNA则附于管壁及管底,通过离心方法即可将它们分离,从而达到提取的目的。在提取过程中,若操控不当,基因组DNA会发生机械断裂,产生大小不同的片段,因此分离基因组DNA时应尽量在温和的条件下操作,如尽量减少酚/氯仿抽提、混匀过程要轻缓等,以保证得到较完整的基因组DNA。一般来说,构建基因组文库,初始DNA长度必须在100kb以上,否则酶切后两边都带合适末端的有效片段很少。而进行RFLP和PCR分析, DNA长度可短至50kb, 在该长度以上,可保证酶切后产生RFLP片段(20kb以下),并可保证包含PCR所扩增的片段(一般2kb以下)。不同生物(植物、动物、微生物)的基因组DNA的提取方法有所不同; 不同种类或同一种类的不同组织因其细胞结构及所含的成分不同,分离方法也有差异。在提取某种特殊组织的DNA时可参照文献和经验建立相应的实验方法, 以获得可用的DNA大分子。组织中的多糖和酶类物质对随后的酶切、PCR反应等有较强的抑制作用,因此用富含这类物质的材料提取基因组DNA时, 应考虑除去多糖和酚类物质。三、实验仪器和材料 台式高速离心机恒温水浴陶瓷研钵1.5ml 离心管移液器无菌枪头无菌牙签液 氮吸水纸四、实验试剂 DNA提取洗涤液100 mmol/L Tris•HCl(pH8.0),3%可溶性PVP,20 mmol/L 巯基乙醇,20 mmol/L EDTA(pH8.0))DNA裂解液(100 mmol/L Tris•HCl(pH8.0),20 mmol/L EDTA(pH8.0),500 mmol/L NaC1,1.5%SDS)酚/氯仿/异戊醇(v:v:v=25:24:1)5M KAc无水乙醇异丙醇70%乙醇含5g/ml RNase 的TE缓冲液