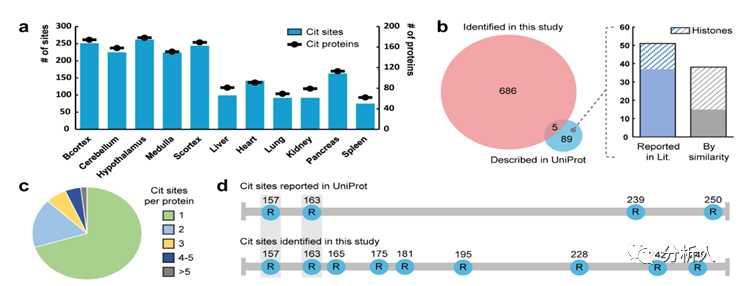
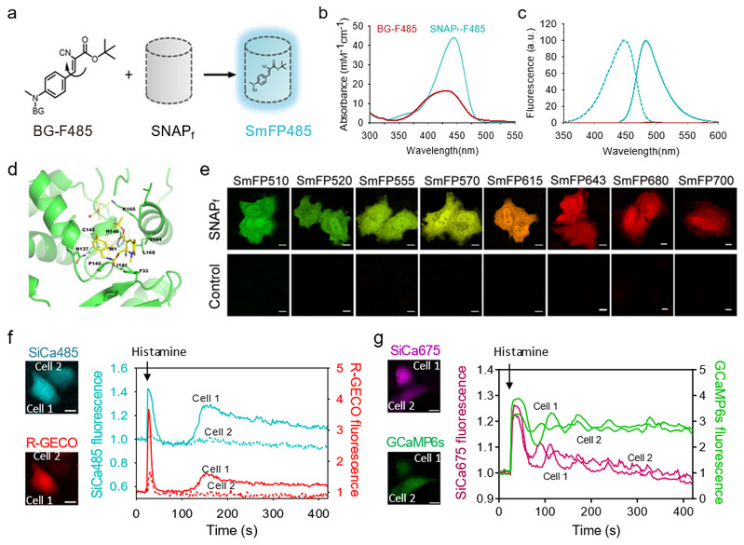
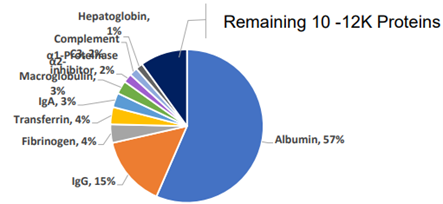
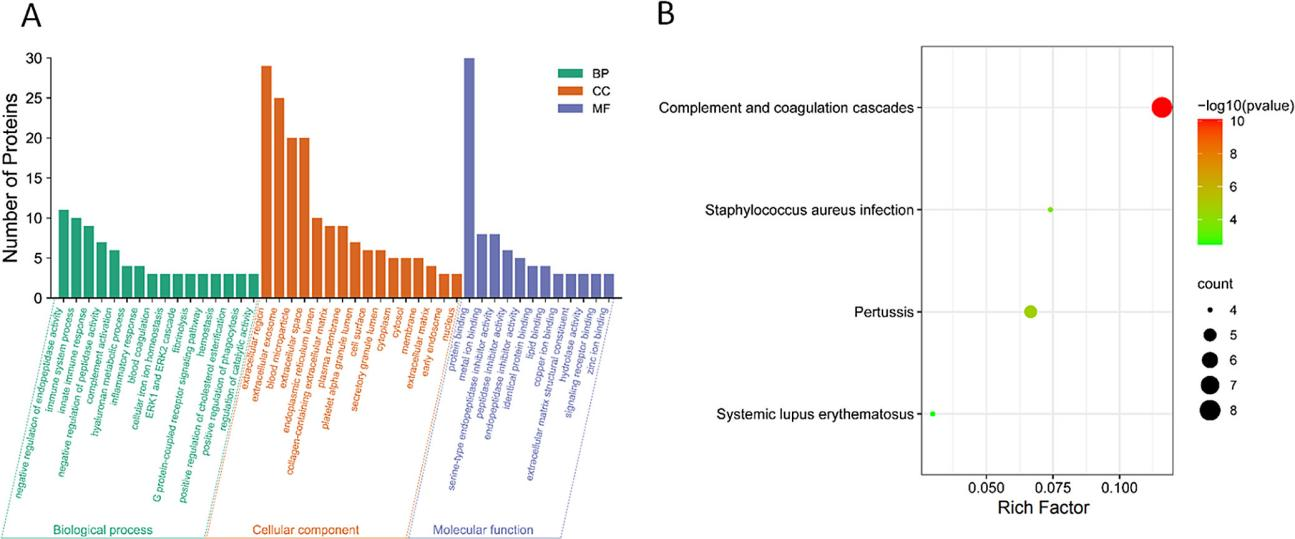
超大孔填料在蛋白质分离纯化中的应用
p /pp 层析纯化技术由于其高选择性、灵活性、易放大性等优点,已经成为蛋白质药物纯化中不可或缺的技术。传统的层析填料为多糖基质,孔径一般在100 nm以下。1970年代出现了大孔和微孔无机材料硅填料,虽然增大了孔道、提高了层析的分辨率和流速,但只能在PH2-7.5范围内稳定,不利于分离纯化在碱性范围内稳定的蛋白质或是需要碱性层析条件的分离,从而限制了其在大规模快速分离蛋白质层析上的应用。多孔聚合物微球由于其高的比表面积、高的机械强度和多样的表面特征,常被用作层析分离纯化的填料。目前已发展出了多种表面基团、基质种类的层析填料,成功用于疫苗、病毒、抗体、酶、细胞因子等的分离纯化。/pp span style="color: rgb(0, 176, 240) "strong 层析纯化病毒、病毒样颗粒等生物大分子的瓶颈问题/strong/span/pp 随着病毒、病毒样颗粒在疫苗、肿瘤治疗、免疫治疗中的地位越来越重要,这类复杂生物大分子的分离纯化需求也逐渐增加。然而传统填料由于孔径较小,蛋白质只能以扩散方式通过填料,传质速率慢,处理量低,造成分离时间长、容易失活等问题[1]。当蛋白质体积较大时,填料表面在吸附一层蛋白后,由于体积位阻以及静电排斥作用,会阻碍其它的蛋白质进一步进入孔内,造成填料的载量下降。另一个限制是病毒或疫苗,尤其是带有包膜的病毒或疫苗,在狭窄的填料孔径内发生吸附时非常容易发生结构变化,破坏其整体结构。在乙肝病毒表面抗原(HBsAg)的纯化中发现这种病毒样颗粒在层析时会发生解聚[2],经过离子交换层析分离后,疫苗的回收率通常不到50%[3, 4]。而抗原的结构发生变化以后,就会对其免疫原性产生影响,所以需要在纯化过程中尽可能维持抗原的结构。/pp 为了解决针对病毒及病毒样颗粒纯化的瓶颈问题,目前已有采用膜色谱、超大孔贯穿孔颗粒填料及整体柱的策略进行纯化的案例,成功纯化了包括人乳头瘤病毒、番茄花叶病毒、流感病毒、腺病毒、慢病毒及各种病毒样颗粒。/ppspan style="color: rgb(0, 176, 240) "strong 病毒及病毒样颗粒的分离纯化/strong/span/pp 根据文献报道,超大孔填料相比传统层析填料不仅在载量及处理速度上有极大的优势,还更有利于病毒及病毒样颗粒的结构保持。/pp 例如,在重组乙肝病毒表面抗原的分离纯化中,采用具有120nm及280nm超大孔径的离子交换填料DEAE-AP-120 nm和DEAE-AP-280 nm(商品名为中科森辉的Giga系列)具有比传统填料DEAE-FF高7倍以上的动态载量[1]。此外,采用ELISA测定抗原收率,发现采用超大孔填料能够减少重组乙肝病毒表面抗原在层析过程中的裂解,从而显著提高活性抗原的收率。/pp style="text-align: center "img width="576" height="450" title="1.jpg" style="width: 415px height: 282px " src="http://img1.17img.cn/17img/images/201808/insimg/3b67db18-4291-4ab6-9874-209cd57644af.jpg"/ /pp style="text-align: center "重组乙肝病毒表面抗原在不同孔径离子交换填料上/pp style="text-align: center " 的吸附动力学[1]/pp style="text-align: center "img width="497" height="345" title="2.jpg" style="width: 387px height: 289px " src="http://img1.17img.cn/17img/images/201808/insimg/07fdf233-77a5-4c30-8d20-faf7f044b54a.jpg"/ /pp style="text-align: center " 重组乙肝病毒表面抗原从不同孔径的填料上洗脱下来的/pp style="text-align: center " ELISA回收率[1]/pp 对病毒的分离纯化同样有类似的效果。例如在灭活口蹄疫病毒的纯化中,DEAE-FF导致严重的病毒裂解。而采用具有100nm以上孔径的超大孔填料,不仅载量提高10倍以上,还能显著提高病毒在填料上吸附时的热稳定性,从而减少病毒的裂解,具有更高的收率。最终的分离纯化单步收率达90%以上[5]。/pp style="text-align: center " span style="font-size: 14px "strong灭活口蹄疫病毒在传统填料与超大孔填料上的吸附解离过程/strong/span/pp 与商品填料的小孔道填料相比,超大孔结构可能从以下几方面提高对蛋白质构象的稳定性:/pp 1)增大孔道(受限空间):根据蛋白质折叠行为计算显示,蛋白质的折叠速率与空腔大小、形状密切相关,也即当填料孔道与蛋白的相对尺寸超过某一阈值后,蛋白的折叠行为将不受空腔大小影响。与数十纳米中孔结构的传统填料的相比,数百纳米超大孔结构会因孔道增大、与蛋白接触面积减小,从而对某一尺寸下蛋白质的变构行为有所改善。/pp 2)界面曲率:小孔径填料孔道曲率大,填料与蛋白质接触面积大,因此受更大吸附力影响,蛋白质二级结构变化越严重。而曲率更大的超大孔孔道对蛋白二级结构的保护比狭窄孔道更有优势。/pp style="text-align: center " span style="font-size: 14px "strong 表面曲率变化对蛋白接触面积的影响/strong/span/pp 3)改善配基与蛋白活性区域的接触面积:超大孔微球内部数百纳米孔道在修饰配基后可能会有效改善传统填料狭窄孔道内由于配基拥挤造成的蛋白质失活现象。/pp 4)减少蛋白在孔道内的静电排斥作用:有研究者认为,在离子交换填料上蛋白质起初会在孔道入口处形成一圈静电层,这一静电层会对后来蛋白继续进入孔道产生排斥作用从而使孔道关闭,动态载量下降。如果将超大孔填料修饰为离子交换树脂,由于孔道尺寸显著扩大可能会有效改善蛋白吸附静电层对孔道的封闭作用,从而有效引导蛋白质进入超大孔道,提高回收率。/ppspan style="color: rgb(0, 176, 240) "strong 快速分离蛋白质及pDNA/strong/span/pp 除了应用于病毒及病毒样颗粒的分离纯化的分离纯化,利用超大孔填料传质速度快的优势,将超大孔填料镀上亲水表层,再接上不同配基制成多种形式的层析填料,用于快速高分辨率的纯化蛋白混合物或质粒。超大孔填料制备成的亲和层析、反相层析和离子交换层析填料广泛的应用在蛋白质的分离纯化方向,显示出超大孔填料比传统分离填料高速高分辨率的蛋白质纯化优势。/pp 例如以肌红蛋白、转铁蛋白和牛血清白蛋白的混合溶液为模拟体系,考察不同流速下超大孔聚苯乙烯阴离子交换介质(DEAE-AP,商品名为Giga系列)的分离效果,并与DEAE 4FF介质进行了对比。实验结果(图2)显示,作为对照的DEAE-4FF介质在流速达到361 cm/h时,分离效果已明显降低,而超大孔介质可以在流速高达1084 cm/h的条件下操作,分离效果良好,能够在6 min内实现三种生物大分子的快速分离。/pp style="text-align: center "img width="588" height="170" title="3.jpg" style="width: 473px height: 144px " src="http://img1.17img.cn/17img/images/201808/insimg/65df31ac-bd00-4a08-8a5a-feedfa1aa990.jpg"//pp span style="color: rgb(0, 176, 240) "strong 超大孔填料应用前景与展望/strong/span/pp 近年来,随着生命科学的发展,生物样品越来越复杂,如人的血样、尿样、组织样品等,对生物分离分析技术提出更高的要求。根据超大孔填料固有的诸多优点,通过合成不同种类的超大孔固定相及在固定相上做不同功能的衍生,超大孔填料已经被广泛应用于生物分离分析中,但也存在一些问题。因此,发展新的制备手段,优化制备条件和过程,探索制备和分离机理,对于开辟新的应用领域以及开展实际样品的分离分析有更大的理论和现实意义。/pp 根据已有的文献报道,我们可以预测今后几年的相关工作仍会集中在以下几个方面:/pp (1)规则的聚合物整体材料内部形态。如获得规则的3D网络骨架,可控的孔径尺寸和分布。/pp (2)继续在微分离系统中扩展其应用。如在加压电色谱、微流控芯片材料、微流色谱和纳流色谱系统,甚至纳米器件开发等诸多方面大显身手。/pp (3)表面物理化学性质的调控向功能化、智能化方向发展。如基于分子印迹技术、温度响应以及pH响应的表面智能化的整体材料。/pp (4)制备规模整体柱的开发及其在生物下游技术中的应用。/pp 目前,已经有一部分整体柱实现了商品化,但种类有限,还无法与种类繁多的颗粒型填充柱相提并论,也远未能满足分离分析的需求。而颗粒型的超大孔填料,由于其制备较困难、批次间重复性较差、价格昂贵等,也没有得到广泛的应用。相对于超大孔填充柱,有机相整体柱存在因流动相变会发生溶胀或收缩、机械强度差、比表面积小、柱容量差以及聚合过程中产生的微孔不利于小分子样品的分析等问题,现有报道大都用于生物大分子的分离。硅骨架整体柱也存在必须预先聚合好装入套管中,制备繁琐,比表面积较小的问题。因此,如何以更简便、有效的方式制备高效新型的超大孔填料并将其应用于实际样品的分离分析仍然是今后工作的重心。在实际工作中所面临的层出不穷的问题也是推动新型超大孔填料制备技术和方法发展的源源不竭的动力,在诸多的尝试中很可能就会出现某些性质优良的超大孔填料,这也预示着将来商品化的超大孔会越来越多。/ppspan style="color: rgb(0, 176, 240) "strong 部分商品化的超大孔层析介质/strong/span/pp strong超大孔填料因其具有独特的多孔结构,与传统填料相比具有更加优良的渗透性和传质速率,可以在较低的操作压力下实现高效和快速的分离,已成为继多聚糖、交联与涂渍、单分散之后的第四代分离填料。可以预测,随着制备技术的不断提升,超大孔填料在生命科学、医药、环境和化学化工等领域必将大有可为。/strong/pp 参考文献/pp [1] M.R. Yu, Y. Li, S.P. Zhang, X.N. Li, Y.L. Yang, Y. Chen, G.H. Ma, Z.G. Su, Improving stability of virus-like particles by ion-exchange chromatographic supports with large pore size: Advantages of gigaporous media beyond enhanced binding capacity, Journal of Chromatography A, 1331 (2014) 69-79./pp [2] P.M. Kramberger P, Boben J, Ravnikar M, ?trancar, A.S.m.c.a.b. in, p.a.f.q.o.t.m. virus., J. Chromatogr. A 1144(1)./pp [3] W. Zhou, J. Bi, J.-C. Janson, A. Dong, Y. Li, Y. Zhang, Y. Huang, Z. Su, Ion-exchange chromatography of hepatitis B virus surface antigen from a recombinant Chinese hamster ovary cell line, Journal of Chromatography A, 1095 (2005) 119-125./pp [4] W. Zhou, J. Bi, J.C. Janson, Y. Li, Y. Huang, Y. Zhang, Z. Su, Molecular characterization of recombinant Hepatitis B surface antigen from Chinese hamster ovary and Hansenulapolymorpha cells by high-performance size exclusion chromatography and multi-angle laser light scattering, Journal of Chromatography B, 838 (2006) 71-77./pp [5] S.Q. Liang, Y.L. Yang, L.J. Sun, Q.Z. Zhao, G.H. Ma, S.P. Zhang, Z.G. Su, Denaturation of inactivated FMDV in ion exchange chromatography: Evidence by differential scanning calorimetry analysis, BiochemEng J, 124 (2017) 99-107./pp/p