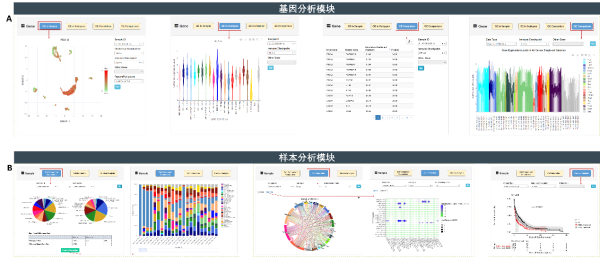
微生物所开发出新型微滴反应筛选技术及单细胞分析应用
p style="text-indent: 2em "微生物所strong微生物资源前期开发国家重点实验室/strong杜文斌研究组和黄力研究组共同开发了一种新型的strong微流控界面纳升注射技术/strong(Interfacial Nanoinjection, INJ),该技术可以将传统的生化反应体系微缩在一个纳升体积的油包水微液滴体系中完成。/pp strongspan style="color: rgb(0, 112, 192) "界面纳升注射(INJ)具有诸多显著优势/span/strong/pp 针对这一技术创新,团队申请了多项中国发明专利和美国专利,并研制了基于INJ技术的小型桌面系统。/pp style="text-align: center"img style="max-width:100% max-height:100% " src="https://img1.17img.cn/17img/images/201910/uepic/fe470173-c411-4a4f-857f-952adad6e8d5.jpg" title="界面纳升注射(INJ)系统.jpg" alt="界面纳升注射(INJ)系统.jpg"//pp style="text-align: center "界面纳升注射(INJ)系统/pp 该系统和国外同类产品如美国Labcyte公司的Echo超声纳升移液系统、以及美国TTP Labtech公司的mosquito HTS微量筛选系统相比,strong在仪器成本、耗材成本、最小液滴体积、流式细胞仪兼容性、操作的灵活性、以及污染控制等方面,均具有显著优势,/strong适用于各类单细胞微体积反应分析,也可应用于其他微体积反应分析,在微生物培养筛选、合成生物学、药物筛选、蛋白结晶条件筛选等方面均具有应用潜力。/pp 在性能方面,INJ系统通过高精密度的微体积控制实现不同试剂组分的纳升体积分步添加,兼容96和384孔板,可以在预先填装矿物油的孔板上,按照程序设定加入纳升样品或试剂液滴,用于实现高通量筛选。利用低成本探针可以精确加注的最小体积达到1 nL,当加样体积为5 nL时,体积标准偏差小于11 %。加注的液滴通过离心可以沉降到孔板底部并融合,液滴的融合效率最高,达到99%以上。利用多次加注样品、试剂的方法,可以实现多步反应和浓度梯度配置。系统加注的体积精确性、线性和重现性良好。/pp strongspan style="color: rgb(0, 112, 192) " FACS-INJ单细胞分析流程和应用/span/strong/pp style="text-align: center"img style="max-width: 100% max-height: 100% width: 600px height: 281px " src="https://img1.17img.cn/17img/images/201910/uepic/7cc806a5-55aa-475f-a110-69b3c5038b70.jpg" title="杜文斌-流式细胞分选+界面纳升注射技术图示.jpg" alt="杜文斌-流式细胞分选+界面纳升注射技术图示.jpg" width="600" height="281" border="0" vspace="0"//pp style="text-align: center "span style="text-align: center "FACS-INJ单细胞分选分析流程/span/pp strong单细胞分析是一项变革性技术/strong,在单细胞基因组异质性研究及复杂微生物群落中稀有微生物种群多样性研究等领域应用广泛。然而,如何进一步降低单细胞分析的成本,提高可靠性和效率,仍然面临重大挑战。流式细胞荧光激活细胞分选(FACS)是目前最高效的单细胞分选技术,可实现病毒、细菌、真菌和动物细胞的多参数检测和分选 利用荧光标记,可对不同类型的细胞进行有效的区分,分选成功率高。研究团队将INJ与FACS平台相结合,建立了FACS-INJ单细胞分选分析流程,应用覆盖了单细胞表型分析、基因型分析、基因表达分析以及全基因组扩增测序。/pp 研究团队首先利用FACS-INJ系统实现了strong病原菌微生物单细胞耐药基因的PCR筛查和单细胞药敏表型筛查/strong。经优化,多孔板可预先装载纳升体积的PCR引物或不同浓度的抗生素液滴。PCR筛查体积缩小到500 nL,试剂消耗和成本和常规体系相比降低至原先的1/40,耐药检测的体积控制在200 nL,试剂消耗和成本和常规体系相比降低至原先的1/1000,时间从 12小时缩短至5小时,这对于大幅降低临床病原检测的成本,实现脑脊液、房水等难获取微量样品的耐药基因和表型筛查具有重要意义。/pp 其次,FACS-INJ系统还可用于strong动物细胞的单细胞基因表达分析/strong。以小鼠巨噬细胞RAW264.7在细菌胞外多糖处理前后的炎症反应为例,通过荧光激活流单细胞式分选处理前后的小鼠巨噬细胞,基于一步法反转录实时荧光PCR扩增,在单细胞水平解析了次黄嘌呤鸟嘌呤转磷酸核糖基酶(HPRT)基因(看家基因)和白介素1β(IL-1β)基因(炎症反应)表达水平的变化。/pp 最后,团队与北京大学黄岩谊课题组合作,建立了strong基于FACS-INJ的微生物全基因组扩增测序流程/strong,以获得未培养微生物的全基因组信息。流程包括流式分选微生物单细胞、单细胞裂解、酸碱中和、MDA扩增和建库测序。以热泉来源的古菌硫化叶菌(Sulfolobus sp. A20)菌株为模型,将单细胞扩增的体积优化至360nL,硫化叶菌全基因组覆盖度达到80%以上。在纳升级微液滴中实现西南印度洋未培养单细胞微生物全基因组DNA的MDA扩增与测序,拼接后获得15个单细胞基因组,大小在0.1~3.7Mb大小。该方法获得的微生物基因组污染度较传统的MDA扩增方法显著降低( 5%),显著提高了微生物单细胞基因组数据质量。平台也适用于肿瘤、胚胎等动物细胞的全基因组扩增测序,对肿瘤细胞的单细胞测序的覆盖度达到60-80%。/pp 上述研究工作近期作为特邀论文在线发表在Small上。微生物研究所助理研究员贠娟莉博士、郑小伟博士、徐鹏博士为论文共同第一作者,杜文斌研究员、黄力研究员和北京大学黄岩谊教授为论文共同通讯作者。该研究该研究得到了中国大洋协会大洋十三五重点项目、中科院战略性先导科技专项(B类),中国科学院重点部署项目、中国科学院前沿科学重点研究项目、国家自然科学基金面上项目和优青项目等支持。/pp 论文出处:/pp Yun, J.L.sup#/sup Zheng, X.W.sup#/sup Xu. P.sup#/sup Zheng, X. Xu, J.Y. Cao, C. Fu, Y.S. Xu, B.X. Dai, X. Wang, Y. Liu, H.T. Yi, Q.L. Zhu, Y.X. Wang, J. Wang, L. Dong, Z.Y. Huang, L.* Huang, Y.Y.* Du, W.B.* Interfacial Nanoinjection-based Nanoliter Single-cell Analysis, Small, 2019, doi:10.1002/smll.201903739./pp a href="https://onlinelibrary.wiley.com/doi/abs/10.1002/smll.201903739" target="_self" style="text-decoration: underline font-family: 楷体, 楷体_GB2312, SimKai color: rgb(255, 0, 0) "strongspan style="font-family: 楷体, 楷体_GB2312, SimKai color: rgb(255, 0, 0) "查看原文戳这里/span/strong/a/pp strongspan style="font-family: 楷体, 楷体_GB2312, SimKai "杜文斌/span/strongspan style="font-family: 楷体, 楷体_GB2312, SimKai ",男,中科院微生物研究所研究员。2007年于浙江大学化学系,获博士学位。2007-2011年于美国芝加哥大学化学系从事博士后研究工作。2013年11月加入中科院微生物研究所微生物资源前期开发国家重点实验室。主要从事微流控芯片技术及新型分析微生物技术与应用研究。已发表论文60余篇,申请中国和美国专利30余项,授权20多项。主持国家优秀青年科学基金项目,国家重点研发计划 “数字诊疗装备研发”项目,中国科学院前沿科学重点研究项目等。/span/p