









近年来,合成生物学作为一门新兴交叉学科获得了蓬勃发展,为破解人类面临的资源、健康、环境等重大挑战提供全新解决途径。合成生物学的核心理念是通过对DNA等生物大分子的逆向设计重构获得具有特定功能的人工生物系统。然而,这些生物分子编码的组合排列空间十分庞大,序列与功能的映射关系复杂。因此,对科学家而言,生物大分子序列的精准设计将极具挑战性。
该领域,AI在提取复杂模式、生成复杂对象方面具备潜力,而人类专家则更擅长在小样本中识别明确模式。正所谓"独行快、众行远",近期,清华大学自动化系汪小我团队在《Nature Communications》发表了题为:“Deep flanking sequence engineering for efficient promoter design using DeepSEED ”的研究论文。

该研究提出了一种将上述二者"强强联合"的设计方法--DeepSEED。他们将专家知识与大数据学习相融合的合成启动子人工智能辅助,发现了转录因子结合位点旁侧序列在启动子优化设计中的重要作用,为突破基因调控元件设计中面临的高维度、小样本的核心难题提供了新的思路。
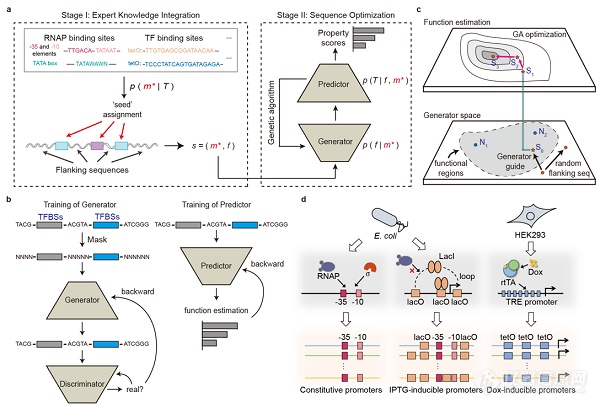
知识引导与数据驱动相融合的启动子辅助设计方法DeepSEED
[来源:仪器信息网] 未经授权不得转载

2023.10.16

2024.08.16

2024.07.29
2024.07.26

深耕固相合成领域,布局智能化、自动化实验室技术——访合肥科晶副总经理安唐林
2024.07.26
版权与免责声明:
① 凡本网注明"来源:仪器信息网"的所有作品,版权均属于仪器信息网,未经本网授权不得转载、摘编或利用其它方式使用。已获本网授权的作品,应在授权范围内使用,并注明"来源:仪器信息网"。违者本网将追究相关法律责任。
② 本网凡注明"来源:xxx(非本网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责,且不承担此类作品侵权行为的直接责任及连带责任。如其他媒体、网站或个人从本网下载使用,必须保留本网注明的"稿件来源",并自负版权等法律责任。
③ 如涉及作品内容、版权等问题,请在作品发表之日起两周内与本网联系,否则视为默认仪器信息网有权转载。
![]() 谢谢您的赞赏,您的鼓励是我前进的动力~
谢谢您的赞赏,您的鼓励是我前进的动力~
打赏失败了~
评论成功+4积分
评论成功,积分获取达到限制
![]() 投票成功~
投票成功~
投票失败了~