







愿历尽千帆,归来仍少年


在二代测序基础上发展起来的RAD-seq技术是一项基于全基因组酶切位点的简化基因组测序技术。由于特异性酶切位点在全基因组范围内广泛分布,通过RAD-seq能够在大多数物种内获得数万至数十万的单核苷酸多态性(single nucleotide polymorphism,SNP)标记。在此基础上,RAD-seq技术又衍生出多种简化基因组测序方法,包含GBS,ddRAD,ezRAD以及2b-RAD等。其中,ddRAD-seq通过一个稀有酶与一个常见酶相结合对基因组DNA进行双酶切,免去随机打断的过程,是一种非常有前景的简化基因组测序技术。不过,ddRAD技术虽然在建库流程上比RAD做了一定程度的简化,但仍然包括12步,实验耗时较长。因此,有必要对现有的ddRAD简化基因组测序文库构建方法进行改进,以克服其需要多次选酶、使用仪器复杂、成本偏高等缺陷,提高测序效率。
中国科学院昆明植物研究所博士研究生杨国骞在研究员郭振华与李德铢指导下,与中科院海洋研究所李莉研究组一起,开发出一种被子植物中通用的双酶切简化基因组(Modified ddRAD,简称MiddRAD)二代测序文库构建方法。该方法首先发现一种被子植物通用酶切组合“AvaII + MspI”,减少了不同物种间需要多次选酶的步骤;其次,该方法将复杂的建库专用仪器优化为通用的分子生物学仪器;再次,该方法将P1测序接头由37个碱基简化为25个碱基(barcode按5个碱基计算)并设计出一套新的barcode-adapter体系;最后,该方法还减少了纯化酶切产物、连接前定量及混样后纯化连接产物的步骤,简化了建库流程,允许仅使用50ng DNA即可完成文库构建。该技术操作简单灵活、检测成本低、检测通量高,更易被科研人员掌握并能在通用分子实验室中实现,特别适用于需要对大量个体进行SNP标记开发及分型的遗传图谱构建、系统发育分析及群体遗传学等研究。因此,MiddRAD-seq在农业分子育种、进化生物学和保护生物学等领域具有良好的应用价值和推广前景。
研究以Development of a universal and simplified ddRAD library preparation approach for SNP discovery and genotyping in angiosperm plants 为题发表于Plant Methods上。
该研究得到国家自然科学基金项目(31470322、31430011)的支持。
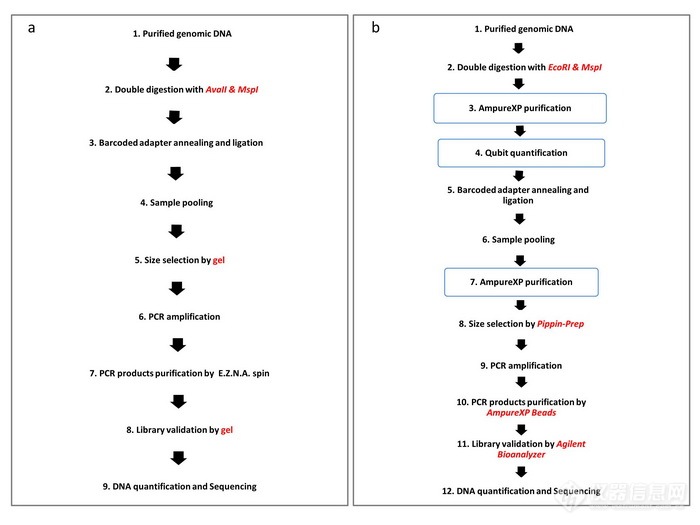
MiddRAD(a)与ddRAD(b)实验流程图
[来源:昆明植物研究所]
 完整的洁净室环境监测系统
完整的洁净室环境监测系统
2024.08.05

关注!国家标准《纳米技术 动态光散射法粒度分析仪技术要求》正式发布
2024.08.02

3i流式新品|盛涵生物Curiox层流洗涤新品,升级助力流式样本前处理
2024.08.02


星赛生物董事长马波受聘“青岛市科学技术信息研究院科技情报顾问”
2024.07.31

2024.07.31
版权与免责声明:
① 凡本网注明"来源:仪器信息网"的所有作品,版权均属于仪器信息网,未经本网授权不得转载、摘编或利用其它方式使用。已获本网授权的作品,应在授权范围内使用,并注明"来源:仪器信息网"。违者本网将追究相关法律责任。
② 本网凡注明"来源:xxx(非本网)"的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责,且不承担此类作品侵权行为的直接责任及连带责任。如其他媒体、网站或个人从本网下载使用,必须保留本网注明的"稿件来源",并自负版权等法律责任。
③ 如涉及作品内容、版权等问题,请在作品发表之日起两周内与本网联系,否则视为默认仪器信息网有权转载。
![]() 谢谢您的赞赏,您的鼓励是我前进的动力~
谢谢您的赞赏,您的鼓励是我前进的动力~
打赏失败了~
评论成功+4积分
评论成功,积分获取达到限制
![]() 投票成功~
投票成功~
投票失败了~