瀚辰小课堂丨基因型填充在动物育种中的应用
基因型填充
基因型填充是指根据现有的基因型信息对没有测定的基因型进行推断的方法。基因型填充具有控制成本、提高准确性、获取更多信息等优点,是全基因组关联分析研究的重要工具之一。
背景
单核苷酸多态性(Single Nucleotide Polymorphism, SNP)是基因组上最常见的一种遗传变异,由单个核苷酸突变引起,占所有已知多态性的90%以上,广泛存在于基因组中。SNP标记信息在基础研究中发挥着巨大的作用,在动植物育种中,可利用基因组SNP标记信息和表型信息,进行主效基因的定位和基因组育种值的估计,进而了解复杂性状的遗传机制,进行早期选择,缩短育种年限,加快动植物的遗传进展。
近年来随着育种研究的深入,学者们意识到,想要对复杂性状的育种值进行更准确的估计,需要大量的遗传变异信息。固相芯片中有限的SNP数目,只能反映个体的部分遗传信息;而二代测序提供的数据能够反映个体的完整遗传信息,但测序的高额成本极大地制约了其应用空间。基于此,人们通常利用基因型填充技术,可以将低密度芯片填充至高密度芯片甚至测序数据,从而在不增加育种成本的同时保证基因组选择或全基因组关联分析的准确性。
基本概念
基因型推断的基本思路是:任何两个个体,即使明显不相关,也可以共享来自一个遥远的共同祖先的短段染色体。因此基因型填充主要是利用参考群体提供的完整基因型信息,构建出彼此连锁标记的单倍型信息,然后利用单倍型信息,将目标群体基因型缺失位点的信息填充完整。
如下图所示,显示了在21个标记上测序的单倍型参考面板对6个标记的研究单倍型(SG)基因分型的推断过程。SG中的等位基因用于匹配参考面板中的短片段。在第一个基因组片段中,等位基因T和G暗示了相应的片段可能是从单倍型X3中复制而来的。在第二段中,等位基因A和T暗示单倍型X5可能已经被复制。同样,研究单倍型可以表示为来自单倍型X3、X5和X6的DNA片段的马赛克。因此,可以计算缺失的位点得到最终的单倍型SI。

主要方法
根据是否利用家系信息,目前提出的主要填充方法包括两大类:
(一)利用群体的连锁不平衡信息构建单倍型,相应的软件有:FAMHAP、fastPHASE、MaCH、Beagle、PLINK、BIMBAM、IMPUTE2、PHASEBOOK等。
(二)利用家系信息和标记信息构建单倍型,相应的软件有Findhap、Fimpute、AlphaImpute、PEDIMPUTE等。根据算法不同,又可将这些方法分为3类,即基于马尔可夫链(MCMC)算法、简化约束法(parsimony)以及期望最大化算法(EM算法)。
下图中的表格展示了基因型填充的各种软件及所对应算法和是否利用家系信息。
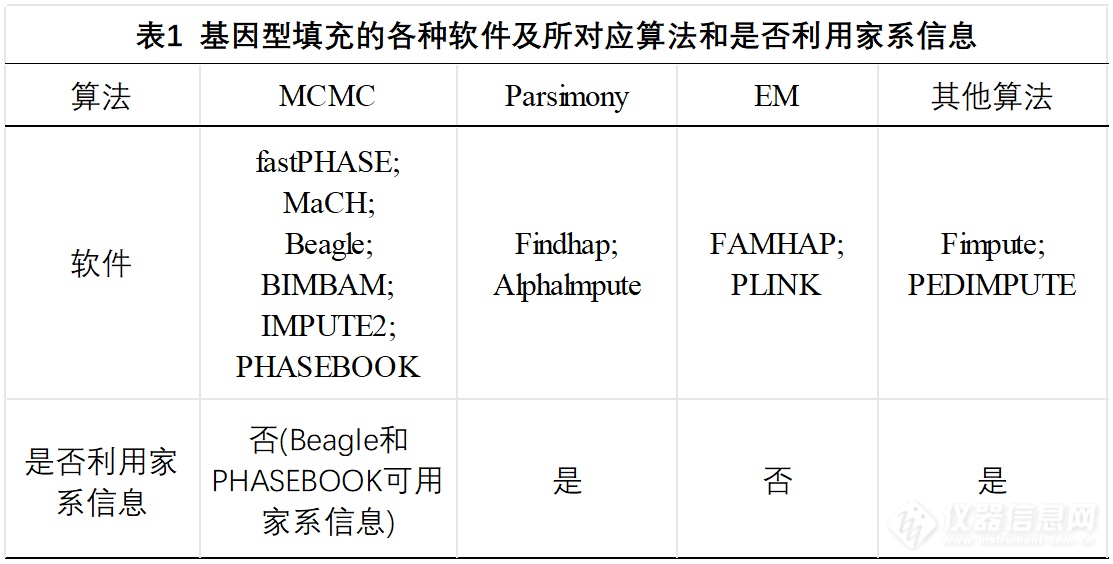
不同的软件及其所对应的算法具备不同的优点,需要根据需求选择最合适的软件。何桑等(2016)对几种基因型填充方法的效率进行了研究。结果发现,在填充率方面,Beagle软件可以保证位点100%填充,而Fimpute和Findhap的填充率略差,会有部分丢失;然而在填充准确率方面,Fimpute软件又优于Findhap和Beagle。
研究进展
近年来,部分学者着力于研究将低密度芯片填充至高密度芯片。王珏等(2019)在1068头杜洛克公猪群体中使用不同密度的SNP芯片进行全基因组选择效果比较分析,结果表明使用基因型填充后芯片和高密度SNP芯片所获得的基因组估计育种值(GEBV)之间可以达到 99% 的相关,并发现个体间亲缘关系的远近对同群体内基因型填充结果的准确率影响不大。
然而随着高通量测序技术的发展,以及千牛基因组等公共数据库的完善,研究者不再满足于将低密度芯片填充至高密度芯片数据,而是想要通过基因型填充获取全基因组序列数据。对此,学者们研究了一步法填充和两步法填充。
(一)一步法里包含将低密度或高密度芯片直接填充至测序数据;
(二)两步法则是先将低密度芯片填充至高密度芯片,再将高密度芯片填充至测序数据。
叶绍潘(2017)的研究表明,使用一步法填充时,芯片密度越大填充准确性越高;而使用两步法填充时,会损失4-9%的准确性,但两步法的分型成本会降低90%。以Beagle为例,当600K芯片数据直接填充测序数据,四条染色体的平均基因型填充正确率为0.812(0.780~0.867),而平均的相关系数为0.807(0.767~0.866);采用两步法将 60 k 填充至 600K 再填充至序列数据,四条染色体的平均基因型填充正确率为0.742(0.732~0.753),而平均的相关系数为0.728(0.712~0.741)。
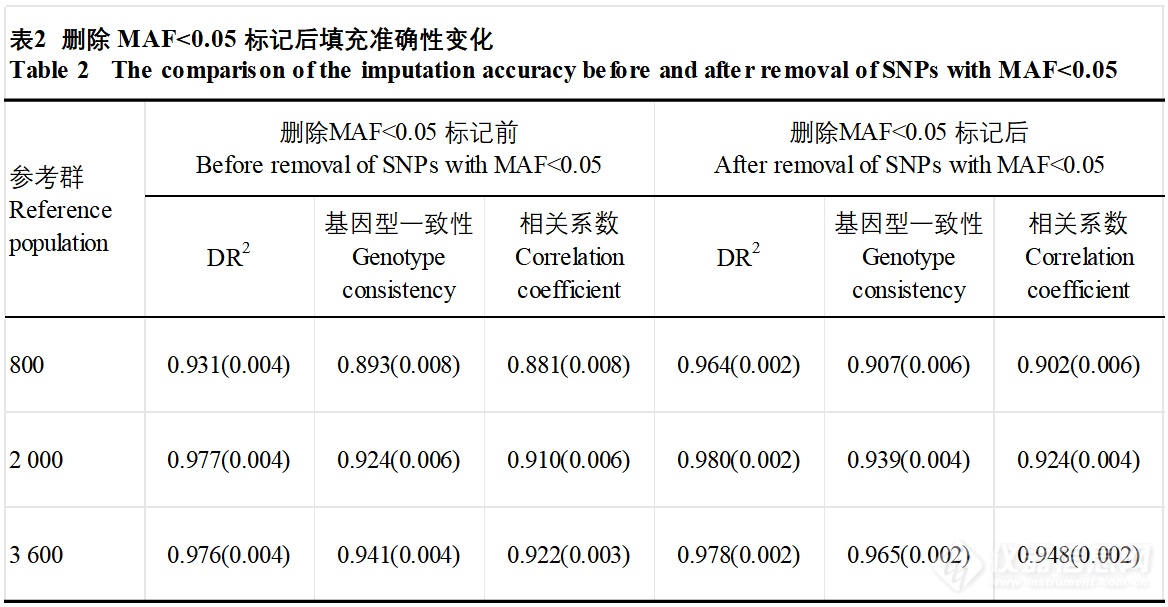
除了围绕固相芯片为主的基因型填充外,还有围绕液相芯片填充的研究。陈宇等(2022)将10k与50k液相芯片相比,标记间距虽然从44kb扩大到226kb,但是SNP标记间的连锁不平衡程度却几乎没有下降。将10K的芯片填充至50k,随着参考群体的加大,填充准确性提高,但参考群体增加至一定规模后,提升幅度较小,部分数据见下表所示。
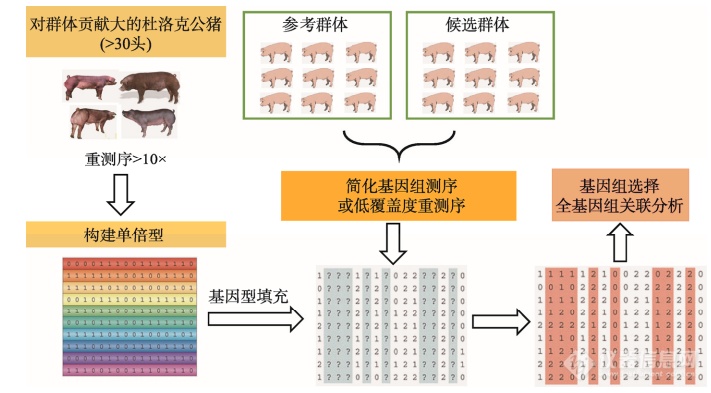
此外,也有学者将重测序和基因型填充结合,一部分小群体进行中高深度的测序,剩余样本进行简化基因组测序或低覆盖度重测序,然后基于连锁不平衡填充没有被充分测序区域的具体基因型, 即此时高深度测序数据作为低深度测序数据填充时的群体参考单倍型集合,用来识别更多的罕见变异和低频率变异,如下图:
结束语
基因型填充可以让学者们获得更多的信息,但在投入、准确率、时间周期上会有相应的取舍。近年来,学者们一直在各个方向优化基因型填充方案,就个人看来,未来会持续在填充测序结果研究及算法上深入发力,相信在测序技术及AI技术的不断发展下,得到基因型填充在育种中的最优解并不遥远。
参考文献
[1] Das S ,Abecasis R G ,Browning L B .Genotype Imputation from Large Reference Panels[J].Annual Review of Genomics and Human Genetics,2018,19(1):73-96.
[2] Ruifei Y ,Xiaoli G ,Di Z , et al.Accelerated deciphering of the genetic architecture of agricultural economic traits in pigs using a low-coverage whole-genome sequencing strategy.[J].GigaScience,2021,10(7):giab048-giab048.
[3] 谈成,边成,杨达等.基因组选择技术在农业动物育种中的应用[J].遗传,2017,39(11):1033-1045.DOI:10.16288/j.yczz.17-286.
[4] 王珏,刘成琨,刘德武等.基于不同密度SNP芯片在杜洛克公猪中的全基因组选择效果分析[J].中国畜牧杂志,2019,55(12):75-79.DOI:10.19556/j.0258-7033.20190927-02
[5] 叶绍潘.基于全基因组测序数据的基因型填充准确性研究[D].华南农业大学,2017.
[6] 陈宇,邱奥,张梓鹏等.猪SNP液相芯片10K~50K基因型填充效果研究[J].畜牧兽医学报,2022,53(10):3368-3376.
[7] 何桑,丁向东,张勤.基因型填充方法介绍及比较[J].中国畜牧杂志,2013,49(23):95-100.
[8] 李乐义,邵东东,丁向东等.SNP芯片基因型填充至测序数据的策略[J].中国科技论文,2016,11(12):1431-1436.
特别鸣谢:瀚辰光翼生物应用部对此文的贡献
更多![]()
全力以"复" | 超值服务产品助力用户复工复产
新品
2022.06.13
支持大规模设备更新行动方案,将传统氮吹仪更新为常压闭路快速蒸发浓缩系统
厂商
2024.06.11
聚光科技生命科学板块携创新技术与产品亮相2024检验医学大会
厂商
2024.08.26
【高校设备更新实施方案】来了!焕新国产—扫描电镜助力科研
厂商
2024.08.27











