【产品推介】BTX电穿孔—助力CRISPR研究

什么是CRISPR
最近几年,CRISPR技术发展如火如荼,一直是角逐诺奖的大热门。CRISPR全称为Clustered Regularly Interspaced Short Palindromic Repeats,规律成簇的间隔短回文重复,是大多数细菌及古细菌中一种不断进化适应的免疫防御机制。CRISPR/Cas原本是细菌抵御病毒的重要武器,而现在它已经成为了基因组编辑的强大工具。风头正劲的CRISPR/Cas不仅操作简便,而且还有很强的可扩展性,被广泛应用到各种生物中,催生了大量的研究成果。
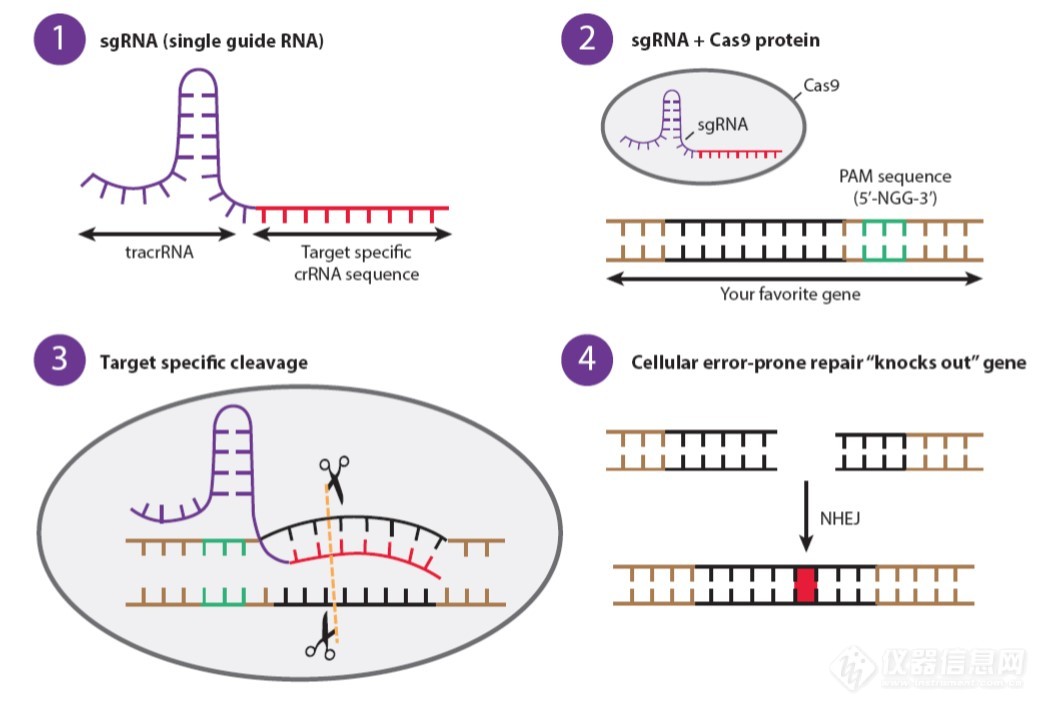
CRISPR导入方式的对比
作为基因编辑技术的弄潮儿,CRISPR/Cas系统具有巨大的潜在应用。然而CRISPR技术并不复杂,将质粒高效导入离体细胞或活体内是关键所在。对比几种常用的转染方法,可以发现电穿孔法操作简便、重复性好、高效低毒,已经成为CRISPR复合体导入细胞(如哺乳动物细胞、细菌、真菌、植物细胞、寄生虫和昆虫细胞)的最佳选择。
特点 | 电穿孔法 | 试剂法 | 显微注射/基因枪 | 病毒法 |
转染效率高 | 是 | 是(有限细胞类型) | 是(有限细胞类型且低通量) | 是(有限细胞类型) |
结果可重复 | 是 | 是(有限细胞类型) | 否 | 是(有限细胞类型) |
单次实验成本低 | 是 | 否(试剂昂贵) | 是 | 否(试剂昂贵) |
适用于所有细胞 | 是 | 否 | 否 | 否 |
适用于所有质粒 | 是 | 否 | 否 | 否 |
操作简便 | 是 | 否(实验准备复杂) | 否(费时费力,需要技巧) | 否(实验准备复杂) |
BTX产品助力CRISPR研究
BTX是美国著名的细胞融合、电穿孔仪的生产厂家。自从1983年发布第一款商用电穿孔仪,BTX只注重于电穿孔和电融合产品的开发,一直走在电穿孔技术的前沿。
BTX电穿孔系统适用于各种类型的CRISPR应用,包括贴壁/悬浮细胞、原代细胞、干细胞、神经元细胞、免疫细胞、受精卵、子宫、卵内、胚胎、活体组织等。

应用举例如下:
*应用于遗传改造
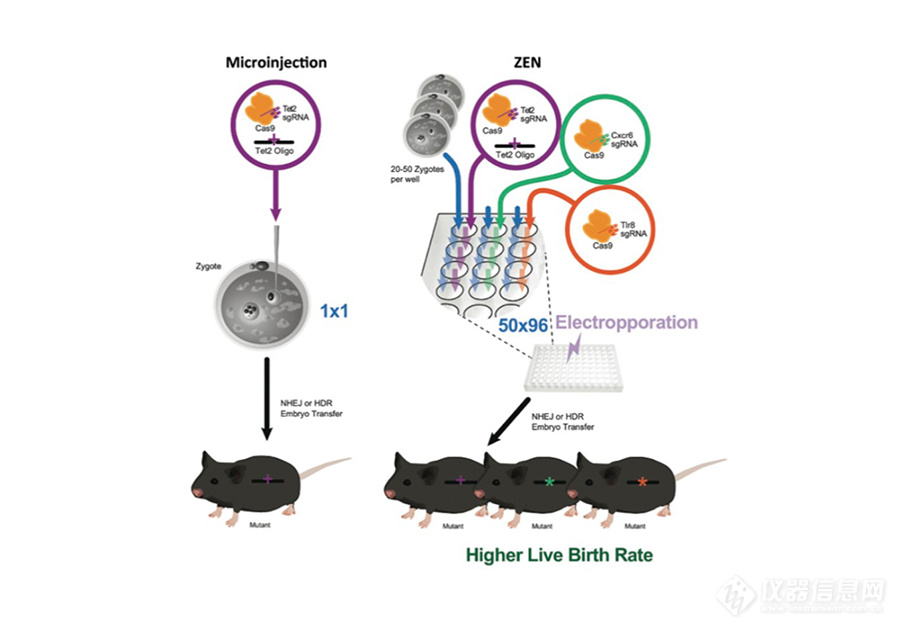
中国科学院动物研究所的王皓毅研究员等证实,使用BTX电穿孔来导入CRISPR/Cas9系统,不仅使得CRISPR/Cas9能够高效地实现实验室小鼠遗传改造,并且可显著提高这一系统的通量(Qin W, et al. Genetics, 2015.)。
*应用于胚胎/活体基因编辑研究
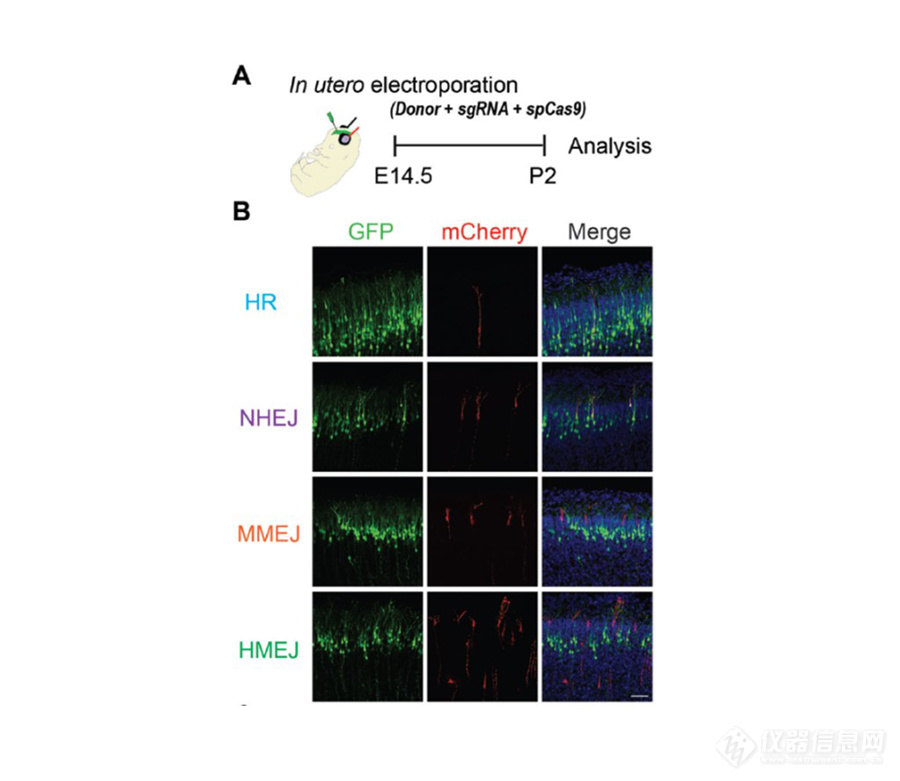
借助BTX产品,中国科学院上海生命科学研究院神经科学研究所杨辉研究组等成功设计了一种以HMEJ为基础的基因敲入策略,在多种系统(体外培养的细胞、动物胚胎和体内组织)中均比现有的基因敲入策略效率高(Xuan Y, et al. Cell Research, 2017.)。
*应用于基因组学研究
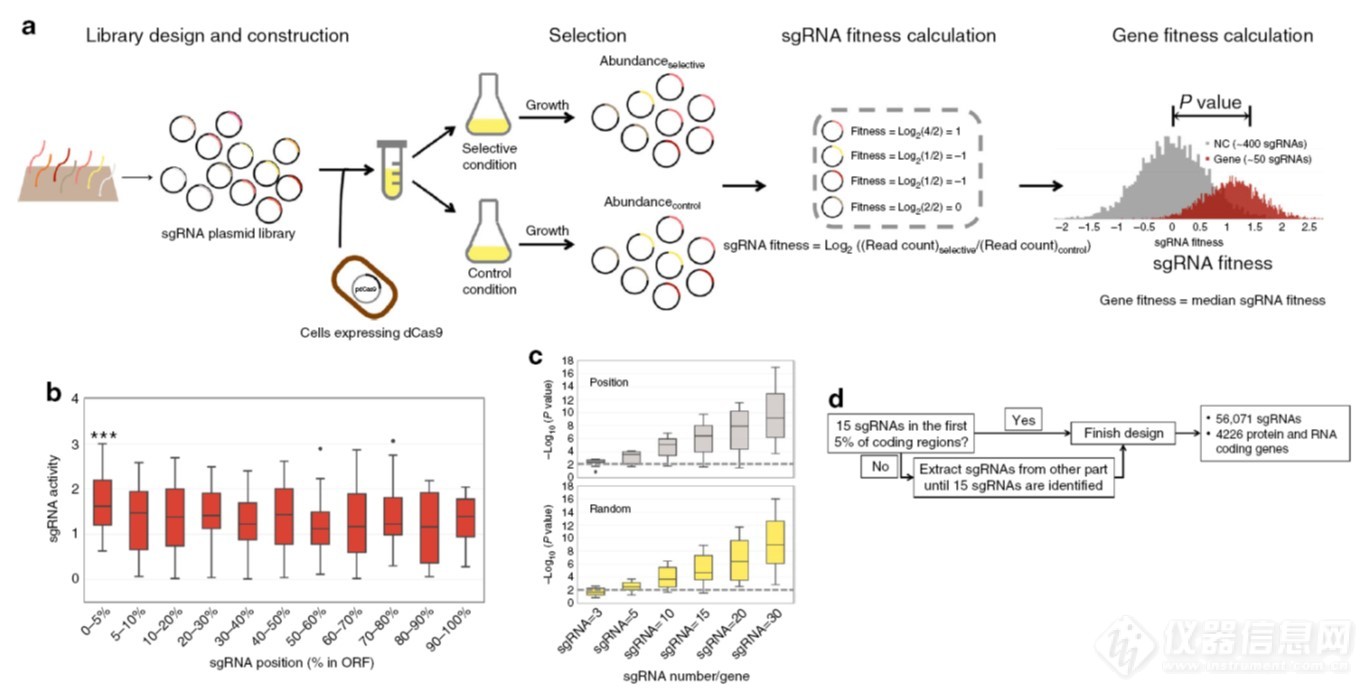
应用BTX电穿孔系统,清华大学化工系生物育种技术与装备团队报道了一种基于 CRISPR/dCas9 敲低技术 (CRISPRi) 的新型细菌功能基因组学方法,可以一次性研究细菌中数千个基因和特定表型的关系(Wang T, et al. Nature Communications, 2018.)。
更多研究者选择的电穿孔系统
经过35年的市场考验,BTX产品在全球各地得到广泛验证和认可,有超过10000篇文献的应用基础,提供海量应用程序、使用指南和解决方案。
近一年内,使用BTX产品在Nature及子刊上发表的部分CRISPR相关Paper请在文末查看.
BTX电穿孔系统选型指南
高配系统:Gemini X2
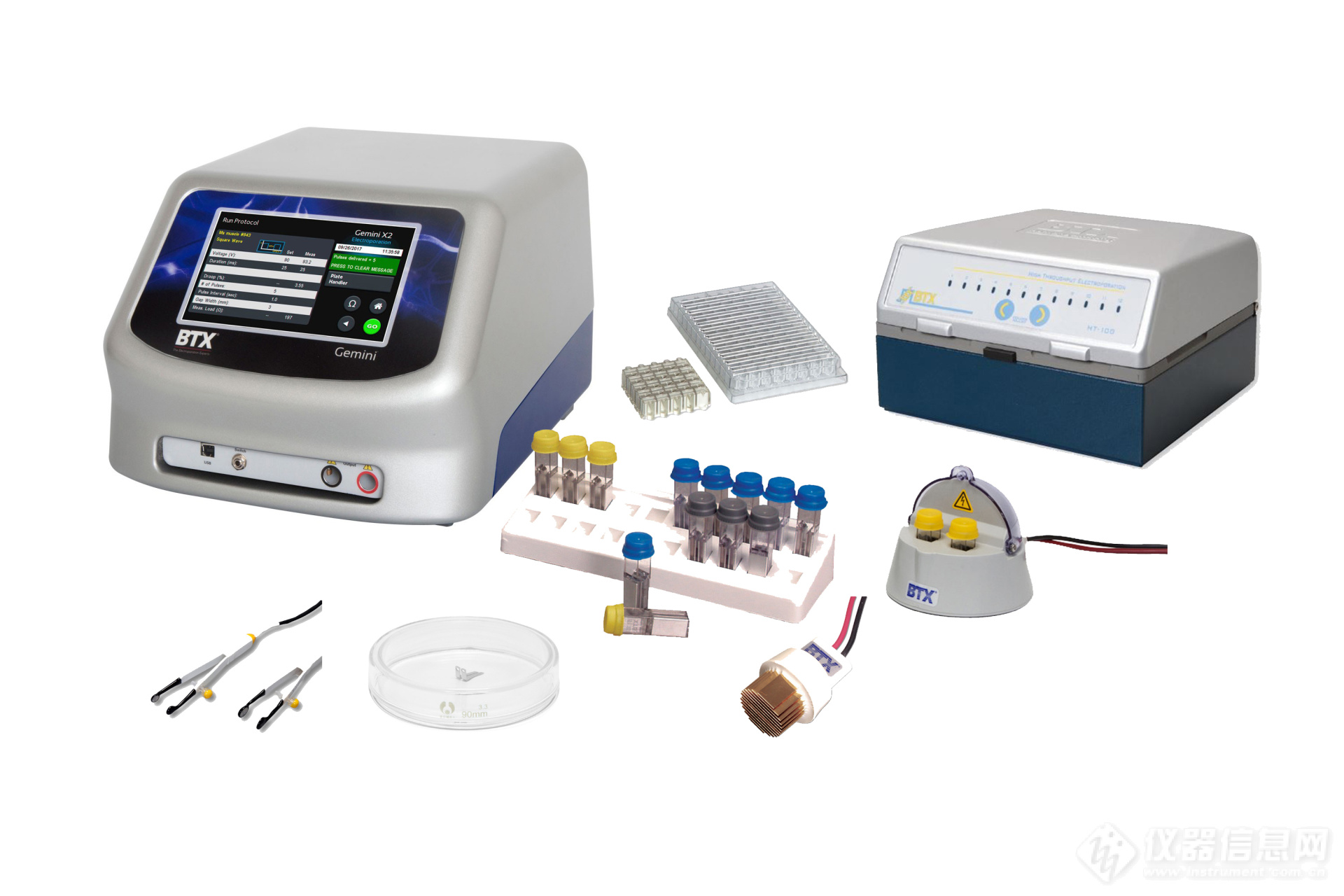
适用于任何类型细胞——提供方波和指数衰减波
7英寸触屏操作,预置大量程序,包括CRISPR预设程序
海量数据记录,无限量存储用户自定义程序,可实现更快参数优化
可与电极杯、活体电极、高通量电极室等配合使用
经典系统:ECM830
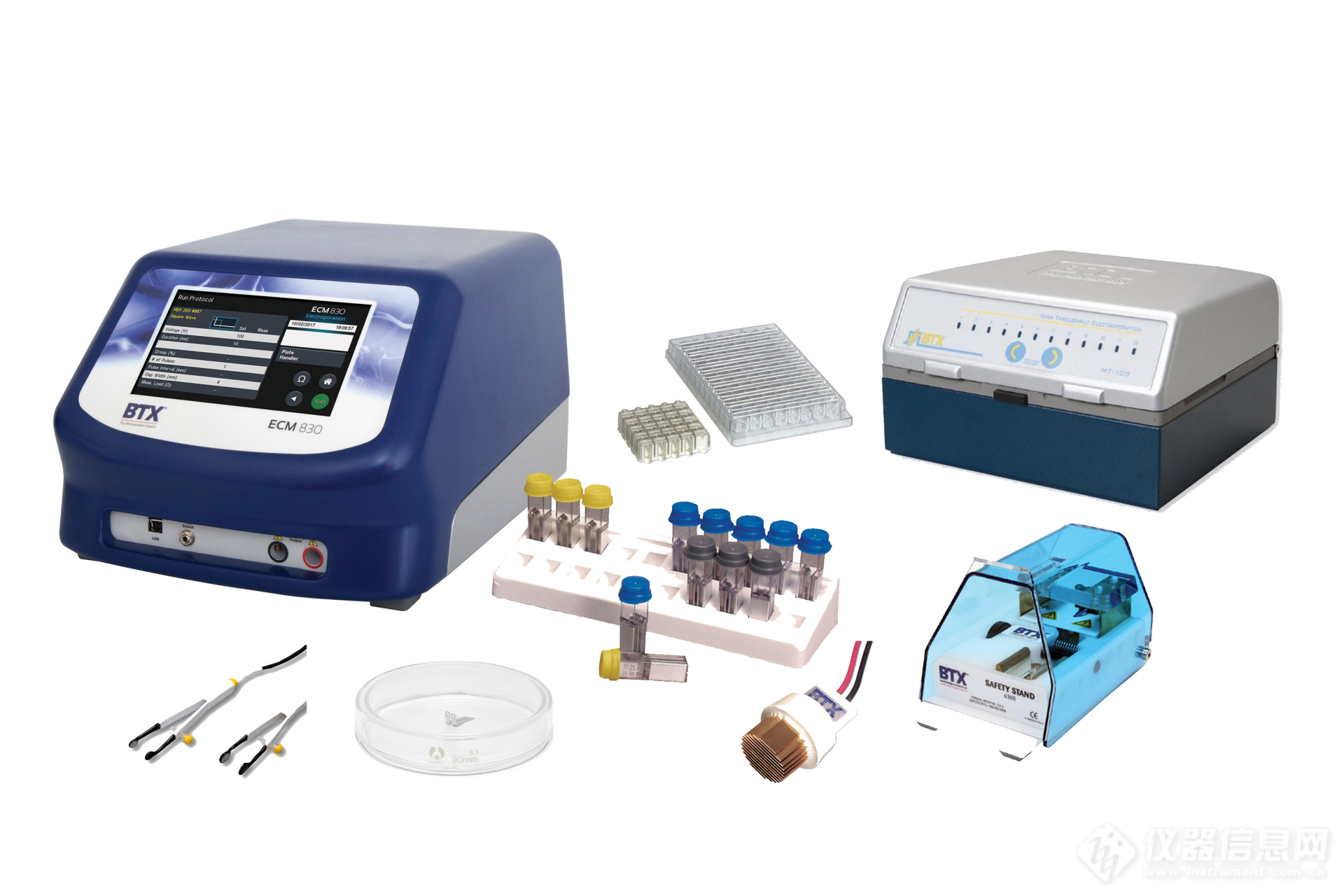
灵活而高效的方波电穿孔系统
7英寸触屏操作,预置大量程序
助力CRISPR技术,大量文献应用记录
可与电极杯、活体电极、高通量电极室等配合使用
1.Glasgow S M, Carlson J C, Zhu W, et al. Glia-specific enhancers and chromatin structure regulate NFIA expression and glioma tumorigenesis[J]. Nature Neuroscience, 2017, 20(11):1520.
2. Guérin A, Corrales R M, Parker M L, et al. Efficient invasion by Toxoplasma depends on the
subversion of host protein networks[J]. Nat Microbiol, 2017, 2(10):1358.
3. Salvatore I, Javier D, Jillian I, et al. The CaMKII/NMDA receptor complex controls hippocampal synaptic transmission by kinase-dependent and independent mechanisms[J]. Nature communications, 2018, 1:2069.
4. Jiang W, Zhu T F. Targeted isolation and cloning of 100-kb microbial genomic sequences by Cas9-assisted targeting of chromosome segments[J]. Nature Protocols, 2016, 11(5):960-975.
5. Kwart D, Paquet D, Teo S, et al. Precise and efficient scarless genome editing in stem cells using CORRECT[J]. Nature Protocols, 2017, 12(2):329.
6. Huang J, Chen M, Whitley M J, et al. Generation and comparison of CRISPR-Cas9 and Cre-mediated genetically engineered mouse models of sarcoma[J]. Nature Communications, 2017, 8:15999.
7. Reijnders M, Kousi M, Van G W, et al. Variation in a range of mTOR-related genes associates with intracranial volume and intellectual disability[J]. Nature Communications, 2017, 8(1).
8. Wang T, Guan C, Guo J, et al. Pooled CRISPR interference screening enables genome-scale functional genomics study in bacteria with superior performance[J]. Nature Communications, 2018, 9:2475.
9. Xie Z, Pang D, Wang K, et al. Optimization of a CRISPR/Cas9-mediated Knock-in Strategy at the Porcine Rosa26 Locus in Porcine Foetal Fibroblasts[J]. Sci Rep, 2017, 7(1):3036.
10. Ikeda M, Matsuyama S, Akagi S, et al. Correction of a Disease Mutation using CRISPR/Cas9-assisted Genome Editing in Japanese Black Cattle[J]. Scientific Reports, 2017, 7(1).
11. Guérin A, Hajj H E, Penaretevargas D, et al. RON4 L1 is a new member of the moving junction complex in Toxoplasma gondii[J]. Scientific Reports, 2017, 7(1).
12. Sidik S M, Huet D, Lourido S. CRISPR-Cas9-based genome-wide screening of Toxoplasma gondii[J]. Nature Protocols, 2018, 13(1):307.
13. Wallis A M, Wallace E C, Hostager B S, et al. TRAF3 enhances TCR signaling by regulating the inhibitors Csk and PTPN22[J]. Scientific Reports, 2017, 7(1):2081.
14. Xuan Y, Xing W, Hu X, et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9[J]. Cell Research, 2017, 27(6):801.
15. Eyquem J, Mansillasoto J, Giavridis T, et al. Targeting a CAR to the TRAC locus with CRISPR/Cas9 enhances tumour rejection[J]. Nature, 2017, 543(7643):113.
更多![]()
天平新品专栏 | 如何应对复杂恶劣环境下的称量?
厂商
2024.08.07
科研界的加速器:挑选合适酶标仪,让实验效率飙升!
厂商
2024.08.07
EAD Talk | 声音专栏:庖丁解牛,看EAD解析双键异构
厂商
2024.07.30
应用案例 | 如何应用DSC测试聚合物结晶度
厂商
2024.07.30